检测过表达株系,可以检测两类基因:(1)真核抗性基因,常见的是潮霉素磷酸转移酶基因(HptII)、草丁膦乙酰转移酶基因(Bar)、新霉素磷酸转移酶基因(NPTII)等,这些基因序列固定,每个实验室估计都有固定的、“传承”下来的引物序列;(2)目的基因,在T-DNA上设计一条引物,在目的基因上设计另一条引物,用这两条引物来进行PCR扩增。
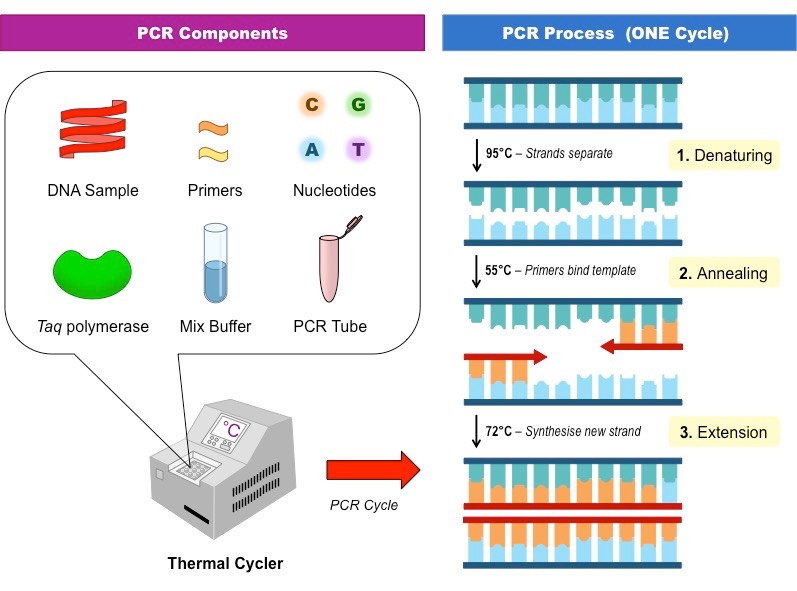
图1 PCR体系。PCR反应体系由DNA模板、引物、dNTPs、Taq DNA聚合酶和PCR反应缓冲液组成。PCR反应在PCR仪中通过控制反应体系的温度来实现。
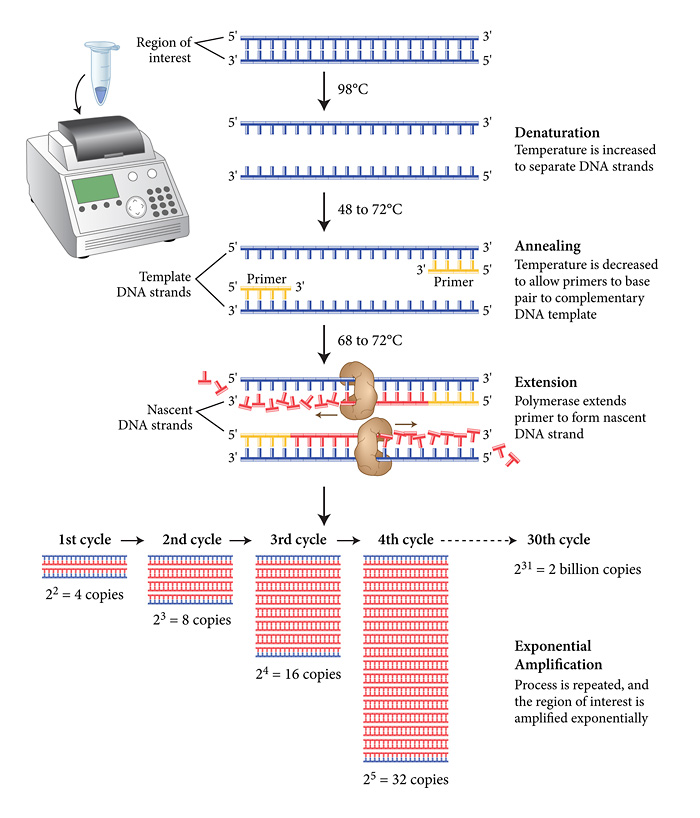
图2 PCR的扩增过程。PCR包含下列三步反应:变性(denaturation)、退火(annealing)、延伸(elongation),此三步反应为一个循环,如此反复循环可以使DNA以指数形式进行扩增。

检测过表达株系,可以先提取目标株系的总RNA,然后将RNA逆转录成cDNA,通过qRT-PCR实验检测靶标的相对表达量。当然,一个内参基因和一个对照样本也是实验中必要的。再利用∆∆Ct法即2–([Ct(样品基因)-Ct(样品内参基因)]–[Ct(对照目的基因)-Ct(对照内参基因)])计算过表达株系目的基因的转录水平变化。有的实验室可以用软件直接生成相对表达量的柱状图,有的实验室也有固定的、“传承”下来的Excel表格可用来计算数据,再根据数据做图。
当然,大家也别忘记,qRT-PCR技术也可用于绝对定量检测、拷贝数鉴定、基因型鉴定等实验。
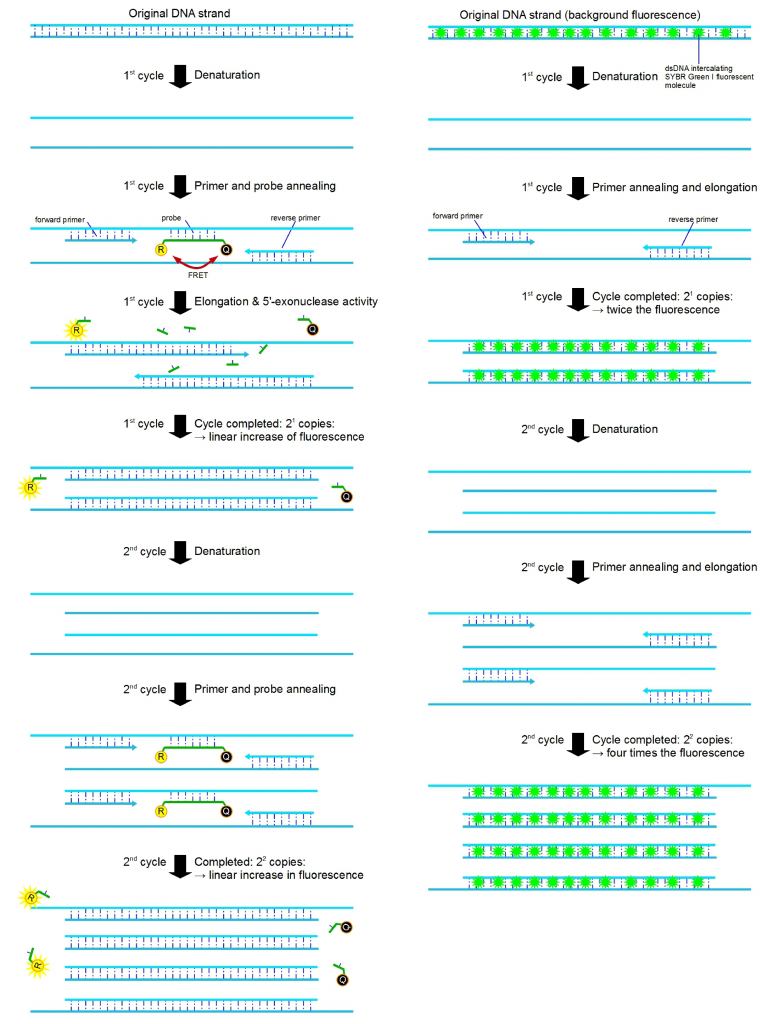
图4 两种不同原理的荧光定量PCR扩增过程。(左)Taqman探针法:使用含有荧光基团的Taqman探针,当探针完整时,报告基团发射的荧光信号被淬灭基团吸收,但在进行延伸反应时,聚合酶的5’-3’外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,即每扩增一条链,就有一个荧光分子形成,因此荧光信号的累积与PCR产物的形成完全同步。该方法可以在同一反应中进行多靶点的检测。(右)SYBR Green I法:使用染料分子SYBR Green I,SYBR Green I能与DNA双链的小沟特异性的结合,游离的SYBR Green I几乎没有荧光信号,但结合双链DNA后,其荧光信号可呈数百倍的增加。随PCR产物的增加,PCR产物与染料的结合量也增大,其荧光信号强度代表双链DNA分子的数量。
Western blot中有两种一抗可以使用:(1)目的蛋白的特异性抗体,一般需要委托公司来生产,成本昂贵。(2)针对特定标签的标签抗体,商业化程度高、特异性强。使用这种抗体时,别忘记在构建转基因载体时,在目的蛋白的N端或C端加上标签,常见的例如His,Flag,Myc等。标签蛋白与目的蛋白就融合在一起,通过检测标签蛋白就可以来表征目的蛋白的表达情况。
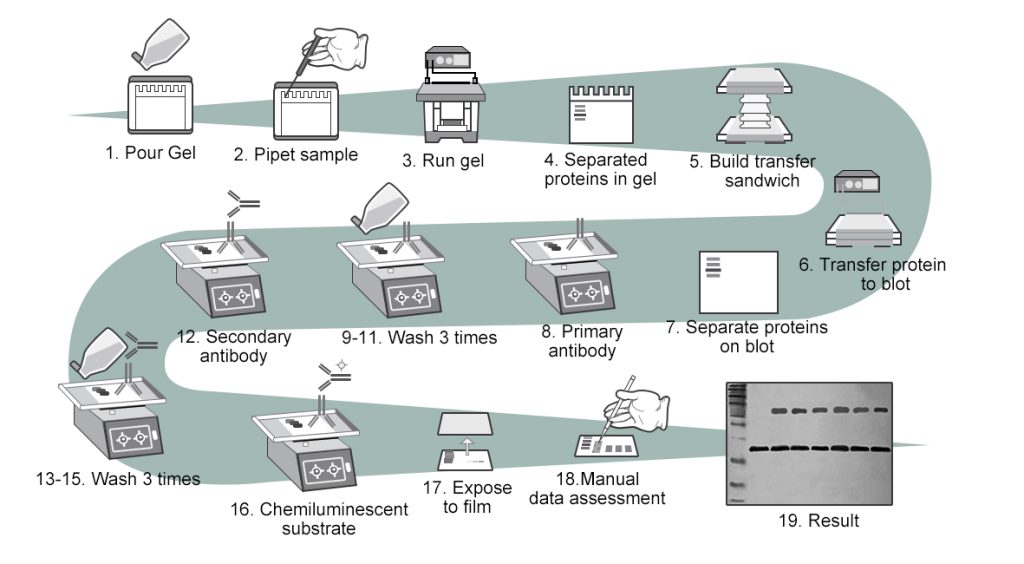
图6 Western blot的实验过程。实验流程大致分为:提蛋白,蛋白含量测定,制胶,聚丙烯酰胺凝胶电泳(SDS-PAGE)分离蛋白样品,转膜,封闭,孵育一抗,漂洗,孵育二抗,漂洗,加入化学发光底物,胶片曝光,得到结果图。
常见的免疫层析试纸条是基于对转基因株系中外源蛋白的检测,如Cry1Ac、Cry2A、BAR、PAT、CP4-EPSPS等蛋白。只需将植物的叶片或种子经过简单的破碎处理,就可进行检测,5-15min即可得到结果。
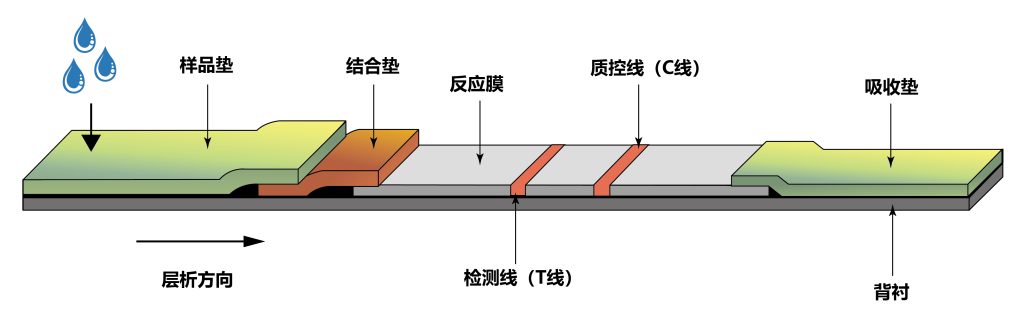
图8 免疫层析试纸条结构 (夏启玉等, 2017)。试纸条由样品垫、结合垫、反应膜、检测线(T线)、质控线(C线)、吸收垫及背衬组成。免疫层析试纸条可分为两类,夹心法和竞争法。以胶体金夹心法为例,简单介绍一下原理:将金标抗体(Au)Ab1吸附在结合垫上,捕获抗体Ab2喷涂于T线,抗Ab1二抗喷涂于C线。待测样品通过毛细管作用向前层析,层析至结合垫上时溶解其上的金标抗体,如果样品中含有待检抗原(Ag)则会与(Au) Ab1结合,形成(Au) Ab1-Ag继续向前层析,前行到T线时被抗体Ab2捕获形成(Au) Ab1-Ag-Ab2的复合物,T线显现红色条带,过量的(Au) Ab1继续前行,在C线处被抗Ab1二抗捕获形成复合物,C线也显示红色条带,为阳性结果;若T线无条带,C线为红色条带,为阴性结果。

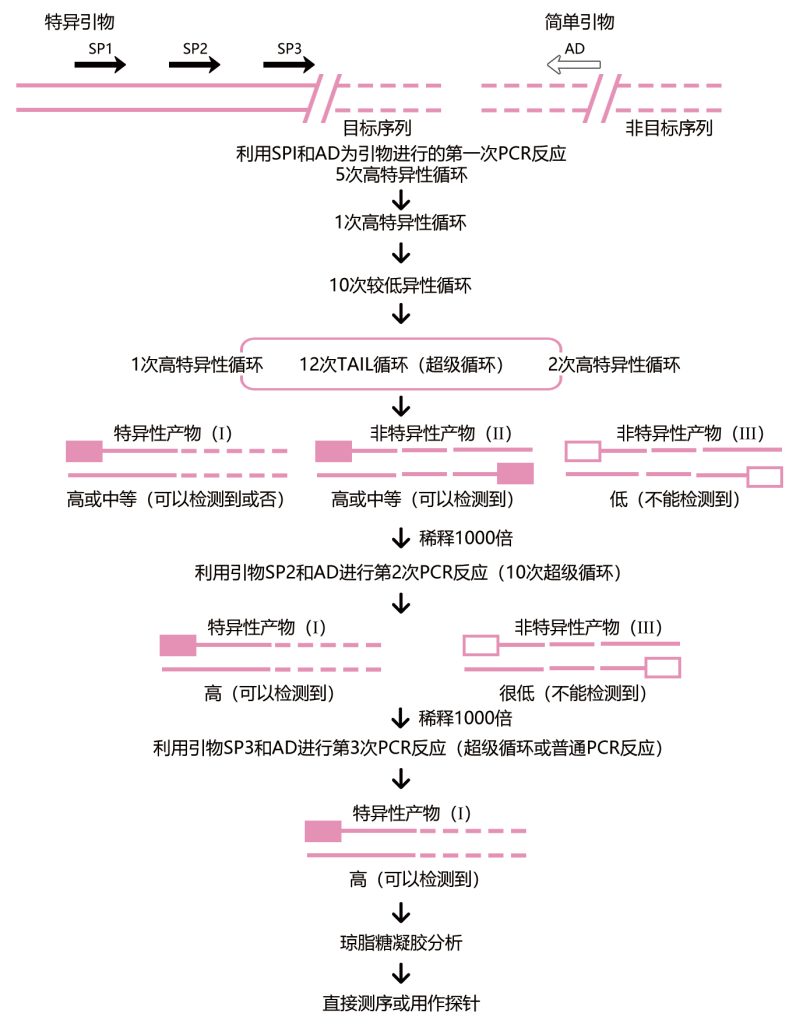
TAIL-PCR技术的基本原理是利用目标序列旁的已知序列设计3个嵌套的特异性引物(简称sp1、sp2和sp3,约20bp),用它们分别和1个具有低Tm值的短的(14bp)随机简并引物(简称AD)相组合,以基因组DNA为模板,根据引物的长短和特异性的差异设计不对称的温度循环,通过分级反应来扩增特异引物。由一个特异性引物和一个简并引物相组合构成的PCR反应叫做“半特异性PCR”。这种反应会产生3种不同类型的产物:(1)由特异性引物和简并引物扩增出的产物;(2)由同一特异性引物扩增出的产物;(3)由同一简并引物扩增出的产物。在TAIL-PCR反应中,后两种非目标产物可以通过以嵌套的特异性引物进行的后续反应来消除。
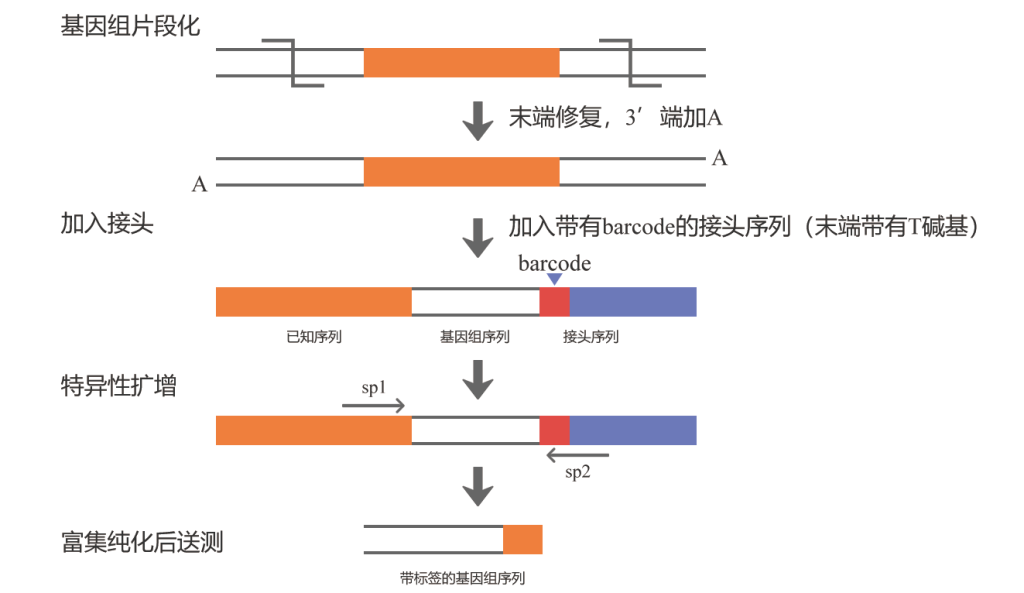
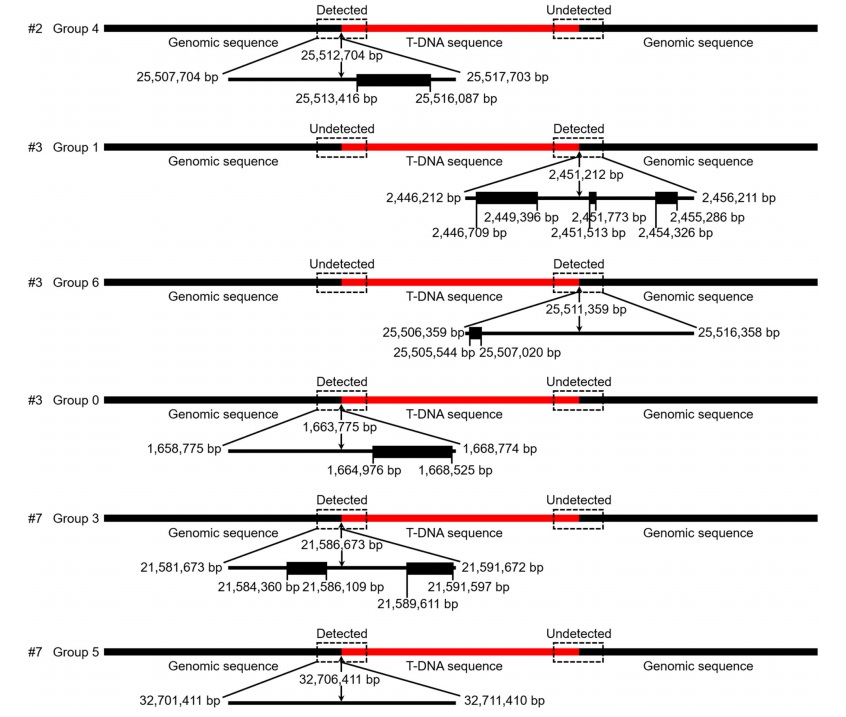
首先将基因组用超声波片段化,末端修复加A后,连接一个带有barcode的接头序列,barcode用于区分不同的样品,接头序列为Illumina测序平台通用测序接头P5和P7,然后根据插入片段和接头序列,分别设计特异性引物扩增片段,再进行二代测序。当然,也可以不进行特异性扩增及纯化而直接进行测序,也就是单纯的全基因组重测序(图12),这种方法也是可以的,只是测序量较大、成本较高。
图11 基于二代测序技术鉴定T-DNA插入位点的实验原理。图片来源:伯远生物官网。
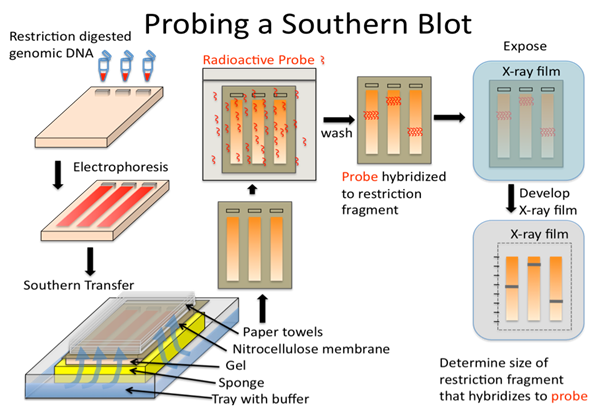
Southern blot是一种常用的DNA定量的分子生物学方法。其原理是将待测的DNA样品用限制性内切酶消化,并通过琼脂糖凝胶电泳分离,再固定在固相载体(硝酸纤维膜或尼龙膜)上,与标记的核酸探针进行杂交,在与探针有同源序列的固相DNA的位置上显示出杂交信号,通过检测信号的有无、强弱可以对样品定性、定量,从而计算出插入的拷贝数。
图13 Southern blot的实验过程。图片来源:LibreTexts。
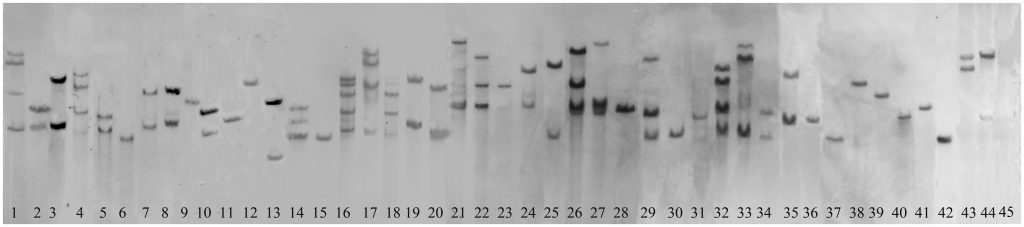
表1 使用∆∆Ct法计算拷贝数 (Bubner et al., 2004)。
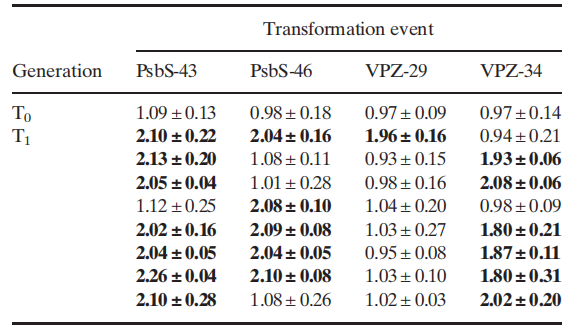
表2 分析T0和T1植株的拷贝数,粗体数字表示由相应的T0、T1植株中的拷贝数鉴定得到的纯合植株 (Głowacka et al., 2016)。
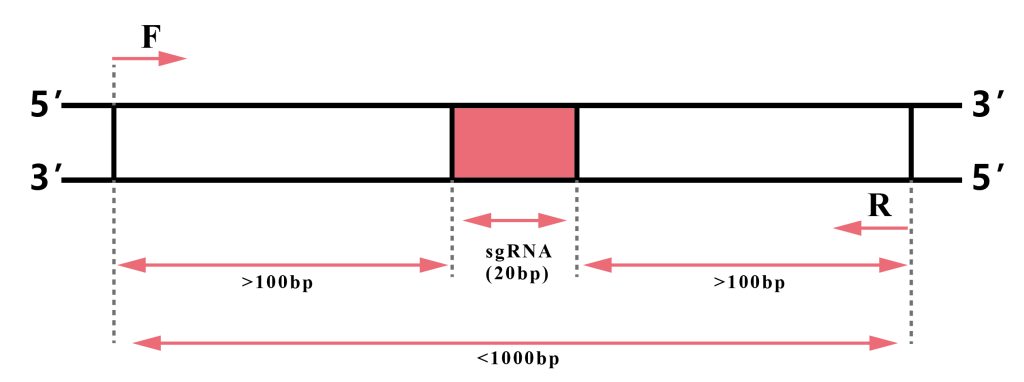
图15 在基因组靶位点附近1000bp范围内设计引物进行扩增、检测,最好控制在500-700bp范围内。图片来源:伯远生物官网。
植物细胞中有两种途径可以修复DNA双链断裂(DSBs):同源重组(Homologous recombination,HR)和非同源末端连接(Non-homologous end joining,NHEJ)。HR在植物中的修复效率非常低,NHEJ在DNA双链断裂处容易产生少数碱基的插入或缺失,导致基因失活,这也是目前CRISPR/Cas基因编辑系统的主要修复途径。
对于二倍体植物来说,NHEJ修复之后会产生5种突变类型:
(1)没有产生任何突变;
(2)单等位基因突变,只有一个等位基因发生突变,也称杂合突变;
(3)双等位基因杂合突变,两个等位基因都发生突变,但是突变的类型不一样,该突变形式常导致软件无法区分,需要继续做克隆及测序来进行鉴定;
(4)双等位基因纯合突变,两个等位基因发生相同的突变,这是研究者们最想要的突变类型;
(5)嵌合体,同一个植株上有3种或3种以上的突变类型。
多倍体产生的基因编辑类型同上。
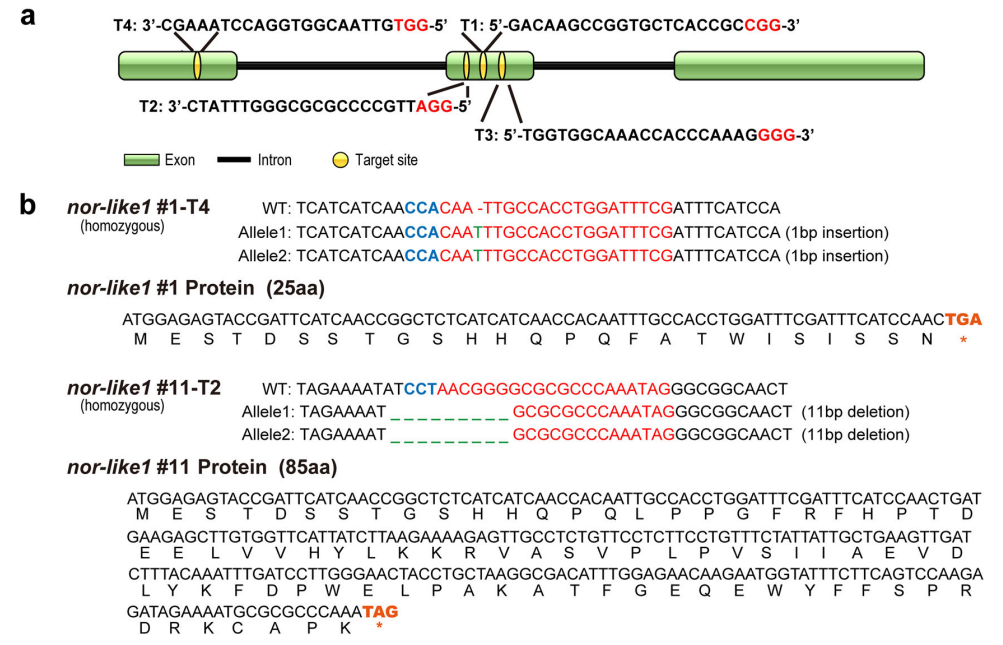
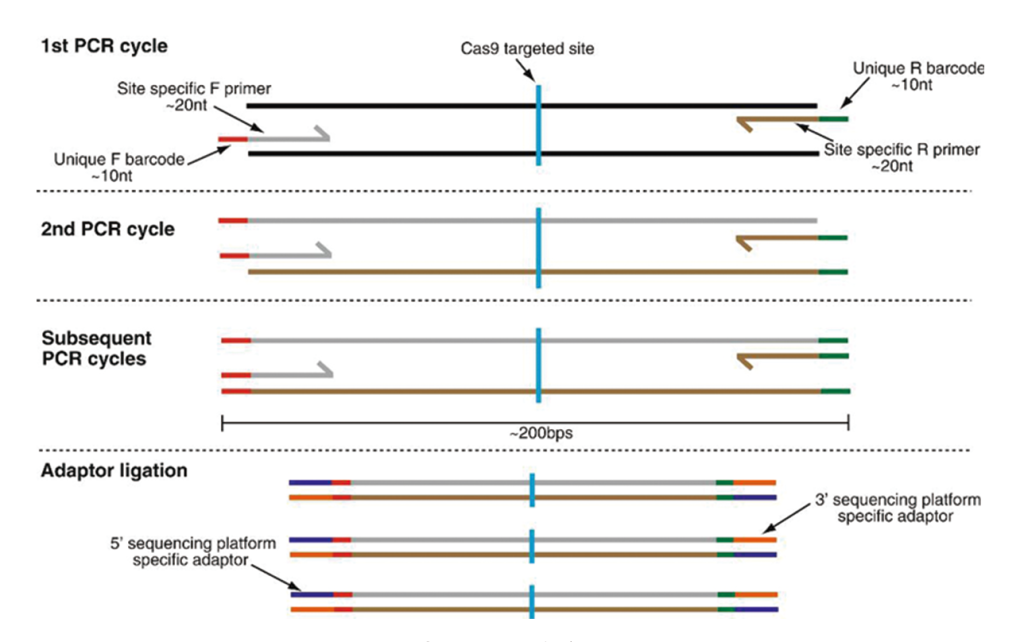
图18 基于二代测序技术的高通量基因编辑检测流程图。基本的实验步骤如下:(1)针对靶位点区间设计特异性引物,可以在上下游特异性引物带上barcode序列,方便多样品混合;(2)利用步骤1针对靶位点设计特异性引物,对目的片段进行首轮PCR,获得目的片段;(3)以步骤2中扩增获得片段为基础,进行第二轮PCR扩增,加上测序接头;(4)混样后,即可进行二代测序;(5)对测序结果进行比对、分析。
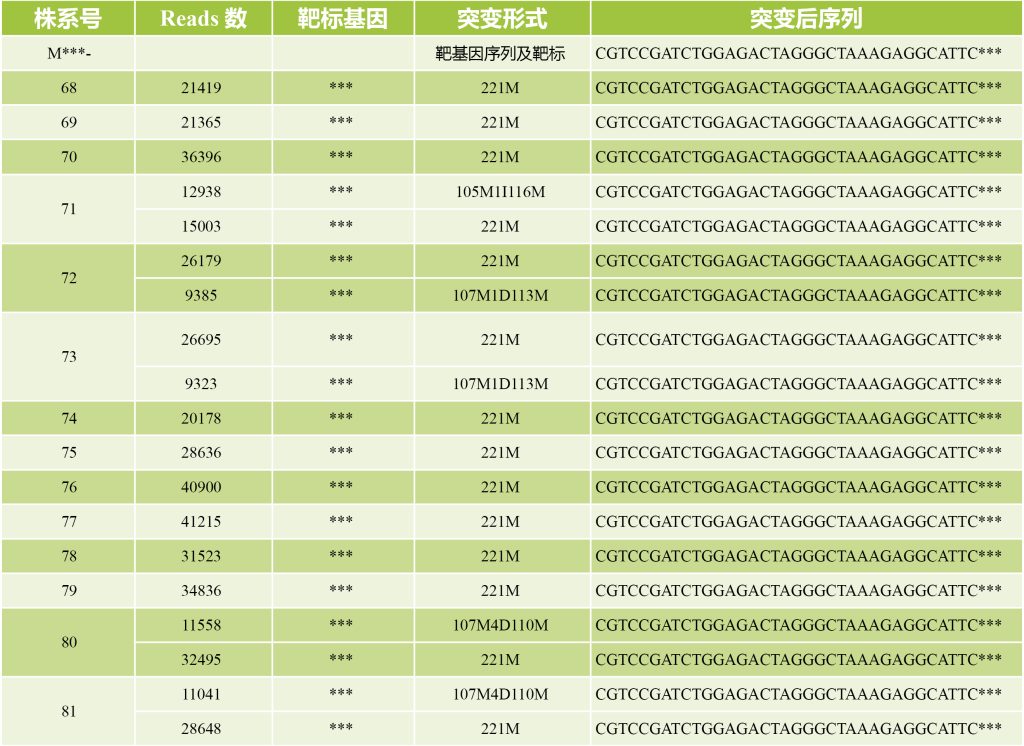
表3 基于二代测序技术的高通量基因编辑检测的结果展示。表格来源:伯远生物检测部。我司提供给客户的表格包含株系号、基因号、靶标序列、Reads数、突变类型、突变后的序列等信息。
对于突变体库的检测,大致可分为以下检测过程:(1)经过遗传转化出苗后PCR进行阳性鉴定;(2)确认每株苗中的sgRNA,二代测序检测;(3)确认突变类型,扩增靶标,二代测序检测。
对于(1)和(3),咱都比较熟悉了,所以只讲一下(2),因为创建突变体库一般采用的是混转策略,将含有不同基因编辑载体(即含有不同sgRNA的载体)的农杆菌混在一起,再对外植体进行侵染、共培、分化出苗等步骤,所以在这个过程中,阳性植株中的sgRNA具体是哪一个我们是不清楚的,并且还可能会同时插入多个sgRNA,因此,我们需要根据插入的sgRNA的情况来确定需要检测的靶标。
对于转化阳性率很高的物种,可以把(1)省略,直接进行(2)和(3)即可,因为未检测到sgRNA,则可认为是阴性苗,直接舍弃即可。
表4 鉴定突变体库的sgRNA(部分展示)。表格来源:伯远生物检测部。
等温扩增技术也是近年发展起来的一种快速核酸检测技术,因其扩增过程不需要特殊仪器、不需要温度循环,即可实现扩增。其中环介导等温扩增技术(LAMP)已经得到较多的应用,通过设计的引物能特异性地识别靶标序列的6个区域,在链置换性聚合酶(Bst酶)作用下,在恒温(60-65℃)条件下,大约40-60min即可完成核酸扩增 (张晓磊等, 2020)。
当然,还有其他很多检测方法,篇幅所限,伯小远就不再多写啦。
References:
Bubner B, Baldwin IT. Use of real-time PCR for determining copy number and zygosity in transgenic plants. Plant Cell Rep. 2004;23(5):263-271. doi:10.1007/s00299-004-0859-y
Gao Y, Wei W, Zhao X, et al. A NAC transcription factor, NOR-like1, is a new positive regulator of tomato fruit ripening. Hortic Res. 2018;5:75. Published 2018 Dec 21. doi:10.1038/s41438-018-0111-5
Głowacka K, Kromdijk J, Leonelli L, Niyogi KK, Clemente TE, Long SP. An evaluation of new and established methods to determine T-DNA copy number and homozygosity in transgenic plants. Plant Cell Environ. 2016 Apr;39(4):908-17. doi: 10.1111/pce.12693. Epub 2016 Jan 21. PMID: 26670088; PMCID: PMC5021166.
Karunarathna NL, Wang H, Harloff HJ, Jiang L, Jung C. Elevating seed oil content in a polyploid crop by induced mutations in SEED FATTY ACID REDUCER genes. Plant Biotechnol J. 2020;18(11):2251-2266. doi:10.1111/pbi.13381
Liu Q, Wang C, Jiao X, et al. Hi-TOM: a platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Sci China Life Sci. 2019;62(1):1-7. doi:10.1007/s11427-018-9402-9
Liu YG, Mitsukawa N, Oosumi T, Whittier RF. Efficient isolation and mapping of Arabidopsis thaliana T-DNA insert junctions by thermal asymmetric interlaced PCR. Plant J. 1995;8(3):457-463. doi:10.1046/j.1365-313x.1995.08030457.x
Liu W, Xie X, Ma X, Li J, Chen J, Liu YG. DSDecode: A Web-Based Tool for Decoding of Sequencing Chromatograms for Genotyping of Targeted Mutations. Mol Plant. 2015;8(9):1431-1433. doi:10.1016/j.molp.2015.05.009
Lv F, Wang P, Zhang E, et al. Efficient Transformation of Catalpa bungei Shows Crystal Genes Conferring Resistance to the Shoot Borer Omphisa plagialis. Front Plant Sci. 2021;12:777411. Published 2021 Dec 24. doi:10.3389/fpls.2021.777411
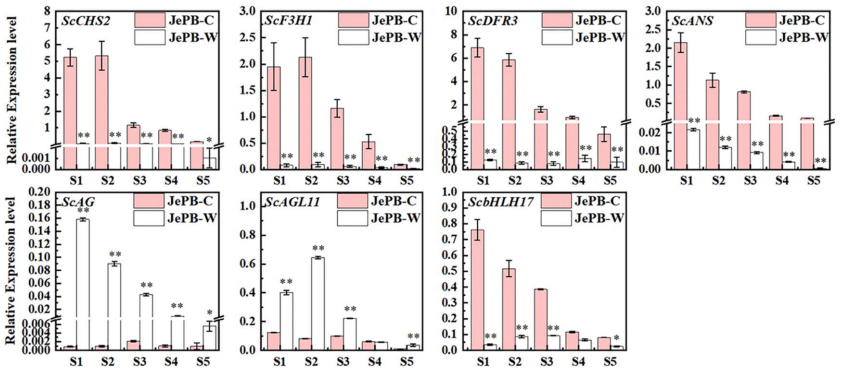
Qi F, Liu Y, Luo Y, et al. Functional analysis of the ScAG and ScAGL11 MADS-box transcription factors for anthocyanin biosynthesis and bicolour pattern formation in Senecio cruentus ray florets. Hortic Res. 2022;9:uhac071. Published 2022 Mar 23. doi:10.1093/hr/uhac071
Singer T, Burke E. High-throughput TAIL-PCR as a tool to identify DNA flanking insertions. Methods Mol Biol. 2003;236:241-272. doi:10.1385/1-59259-413-1:241
Wood CC, Robertson M, Tanner G, Peacock WJ, Dennis ES, Helliwell CA (2006) The Arabidopsis thaliana vernalization response requires a polycomb-like protein complex that also includes VERNALIZATION INSENSITIVE 3. Proc Natl Acad Sci U S A 103 (39):14631-14636. doi:10.1073/pnas.0606385103
Wu, Jinxia; Zhang, Zhiguo; Zhang, Qian; Liu, Yayun; Zhu, Butuo; Cao, Jian; et al. (2015): Detection of the T-DNA copy number in T0 FOX lines by Southern blotting using HPT as a probe.. PLOS ONE. Figure. https://doi.org/10.1371/journal.pone.0132314.g003
夏启玉, 李美英, 杨小亮, 肖苏生, 贺萍萍, 郭安平. 免疫层析试纸条技术及其在转基因检测中的应用[J]. 中国生物工程杂志, 2017, 37(2): 101-110.
张晓磊, 章秋艳, 熊炜,等. 转基因植物检测方法及标准化概述[J]. 中国农业大学学报, 2020, 25(9):12.
| 官网链接:plant.biorun.com |