本文内容速览:
在植物基因功能研究中,作为“主角”的编码区(Coding DNA sequence,CDS)一直是研究所关注的重点,而作为“配角”的非翻译区(Untranslated region,UTR)往往被忽视,受到的关注也相对较少,甚至有时被认为是可有可无的序列。随着基因功能的研究越来越深入与全面,UTR在基因表达调控中的重要作用逐渐被揭示,尤其是在应答特定环境和发育过程中的重要作用。因此,UTR不仅仅只是简单的非编码序列,它也包含着丰富的调控信息,这些信息对基因的转录后调控起着至关重要的作用。本文旨在分享UTR的相关内容,尤其是UTR中的一些重要调控序列如开放阅读框(Open reading frame,ORF)的功能,希望完善大家对UTR的认知。
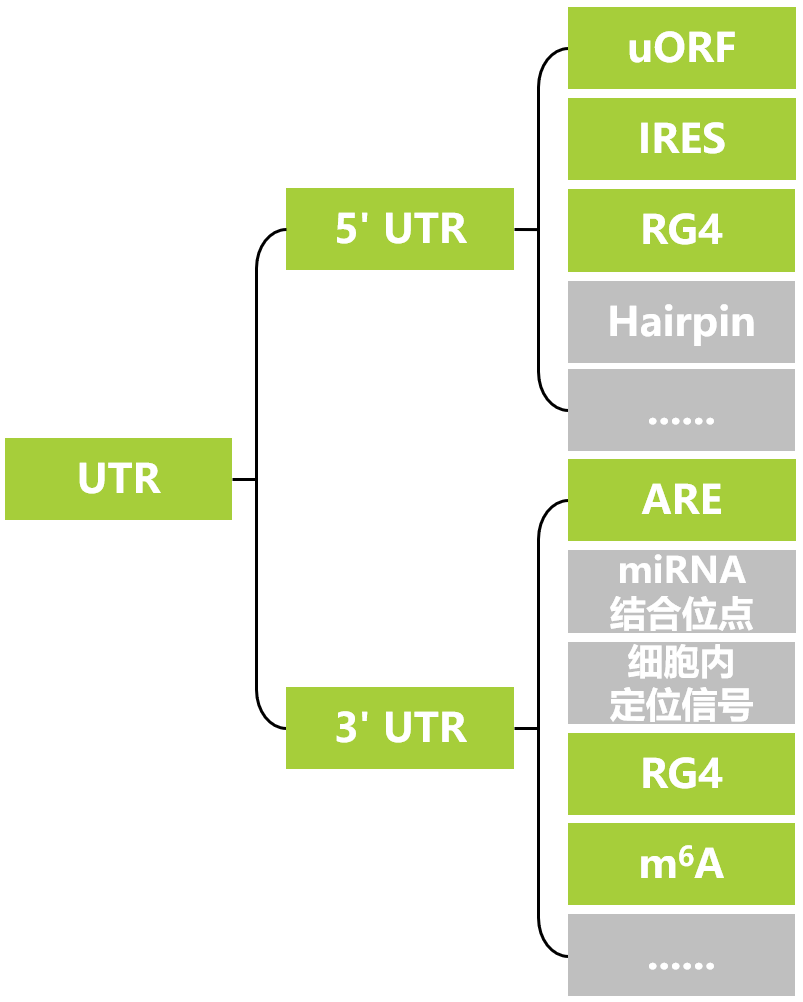
UTR是指位于mRNA两端的非编码序列,分为5′ UTR和3′ UTR。5′ UTR位于起始密码子之前,而3′ UTR则位于终止密码子之后。总体来说,UTR的功能主要体现在其对mRNA的翻译调控、稳定性及亚细胞定位等方面。在特定条件下,UTR中的调控元件可以通过影响核糖体的加载、mRNA的降解和转运等途径,精细调控基因表达的时间和空间模式。具体来说,5′ UTR通常包含影响翻译效率的顺式作用元件,如上游开放阅读框(Upstream open reading frame,uORF)、内部核糖体进入位点(Internal ribosome entry site,IRES)、G-四链体(RNA G-quadruplex,RG4)及具有复杂二级结构的发卡环(Hairpin)等。这些结构通过调控核糖体的结合效率、翻译起始以及mRNA的局部稳定性,精细调节下游编码区的蛋白质合成(图1B-E)。3′ UTR则更侧重于对mRNA的翻译调控、稳定性及亚细胞定位的调控。3′ UTR中通常包含AU富集元件(AU-rich element,ARE)、miRNA结合位点、细胞内定位信号以及RG4等调控元件,这些元件通过调节mRNA的降解速率、翻译抑制和亚细胞定位,在基因表达的精细调控中发挥重要作用(图1F-I)(Hardy et al., 2024)。接下来,小远将列举相关文献案例介绍5′ UTR和3′ UTR中重要元件的调控机制。

图1 UTR中的重要调控元件(Hardy et al., 2024)。(A)mRNA模式图及其UTR上的重要调控元件;(B-I)UTR上重要调控元件所参与分子调控过程的示意图。
5′ UTR中的调控元件
uORF广泛分布于植物基因组上,18~57%的基因都含有uORF。由于非常规起始密码子(非AUG)存在等原因,实际uORF的存在比例可能更高。uORF的种类共有三种,分别是不与mORF(uORF下游的主开放阅读框,有时可理解为CDS)重叠的uORF、与mORF部分重叠的uORF以及包含mORF的uORF(图2)(Niu et al., 2020)。这三种中最主要的还是第一种uORF。对于这种广泛存在的序列,其功能主要是抑制mORF的翻译或引起mRNA的降解,此外也可以促进mORF翻译以及翻译成小肽(Wang et al., 2023)。对于uORF翻译成小肽的过程在往期推文“个头小,作用大——植物中的小肽”中有所介绍,对小肽感兴趣的读者可以阅读回顾。近年来,随着基因编辑的发展,这使得对uORF的深入研究成为可能,此外鉴于uORF在育种中的潜力和前景,uORF在植物中的研究也愈来愈火热,相关文章也不断增多。接下来就给大家分享一篇关于植物中uORF的研究论文。
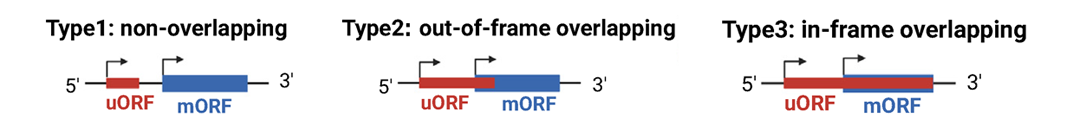
图2 uORF的三种类型(Niu et al., 2020)。
在往期推文“基因敲高——通过基因编辑改善作物农艺性状”中介绍了对uORF进行编辑实现基因敲高的目的。相反地,是否可以人为地通过基因编辑引入uORF实现基因敲低的目的呢?
2023年3月,中国科学院遗传与发育生物学研究所高彩霞课题组在Nature Biotechnology杂志上发表了一篇题为“Tuning plant phenotypes by precise, graded downregulation of gene expression”的研究论文。因为已有研究发现OsBRI1的突变会导致水稻叶片夹角变小等表型(Morinaka et al., 2006),所以这篇论文中以OsBRI1(植物激素油菜素内酯受体编码基因)为研究对象。通过引导编辑的方法在OsBRI1的5′ UTR区域定向突变生成了一个ATG,相当于人为引入了一个uORF(图3A)。后续实验发现,相较于野生型,uORFOsBRI1(-99bp, 28 aa,uORF起始于OsBRI1 mORF起始密码子-99bp的位置,肽段长度为28个氨基酸)突变体中OsBRI1的表达量没有差异,但是蛋白丰度明显降低(图3B、C),这说明人为引入uORF确实导致了OsBRI1翻译水平降低。进一步观察表型发现uORFOsBRI1(−99bp, 28 aa)突变体与已报道的OsBRI1突变后表型类似(图3D)(Xue et al., 2023)。这些结果说明,可以通过引入uORF可以抑制基因的翻译。
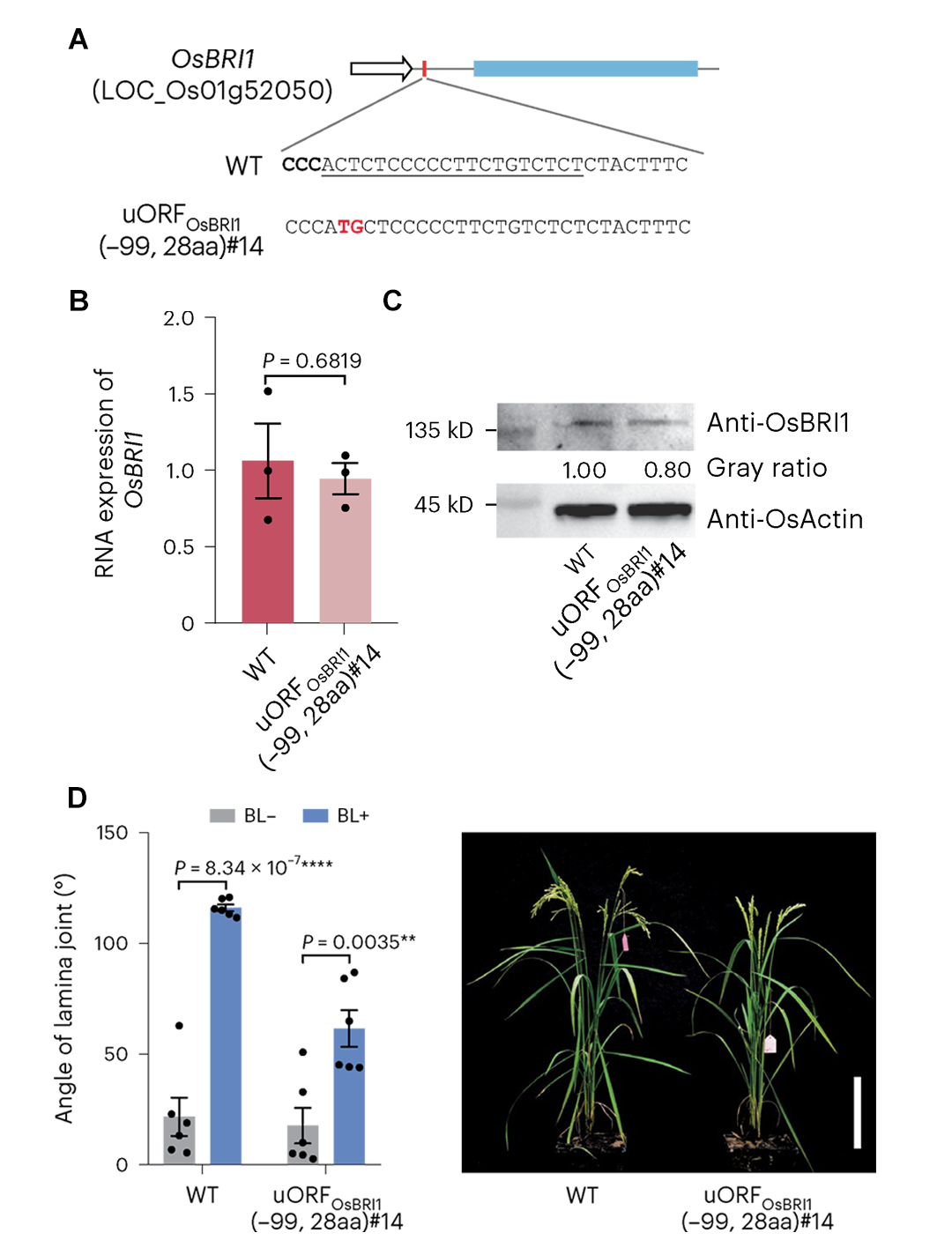
图3 通过引导编辑引入uORF实现基因敲低(Xue et al., 2023)。(A)在OsBRI1的5′ UTR区域引入ATG;(B)野生型和uORFOsBRI1(−99bp, 28 aa)突变体中OsBRI1的mRNA水平比较;(C)野生型和uORFOsBRI1(−99bp, 28 aa)突变体中OsBRI1的蛋白水平比较;(D)野生型和uORFOsBRI1(−99bp, 28 aa)突变体叶夹角表型观察。
在介绍IRES之前,先给大家分享一下真核生物mRNA翻译的起始过程。翻译的起始过程非常复杂,涉及一系列蛋白及复合体,其中两个十分重要的复合体分别是eIF4F复合体和43S PIC复合体。在mRNA准备翻译时,eIF4F复合体中的eIF4E(mRNA 5′ -帽结合亚基)结合在mRNA的m7G帽子上,随后eIF4F复合体招募43S PIC复合体。43S PIC复合体紧接着向mRNA 3’端扫描起始密码子,扫描到起始密码子后具有翻译能力的核糖体正式形成,这也标志着翻译的起始的结束和延伸的开始(Bhat et al., 2015)。此外值得注意的是,这个过程中poly(A)结合蛋白(Poly(A)-binding protein,PABP)能与相关复合物结合以稳定转录起始。
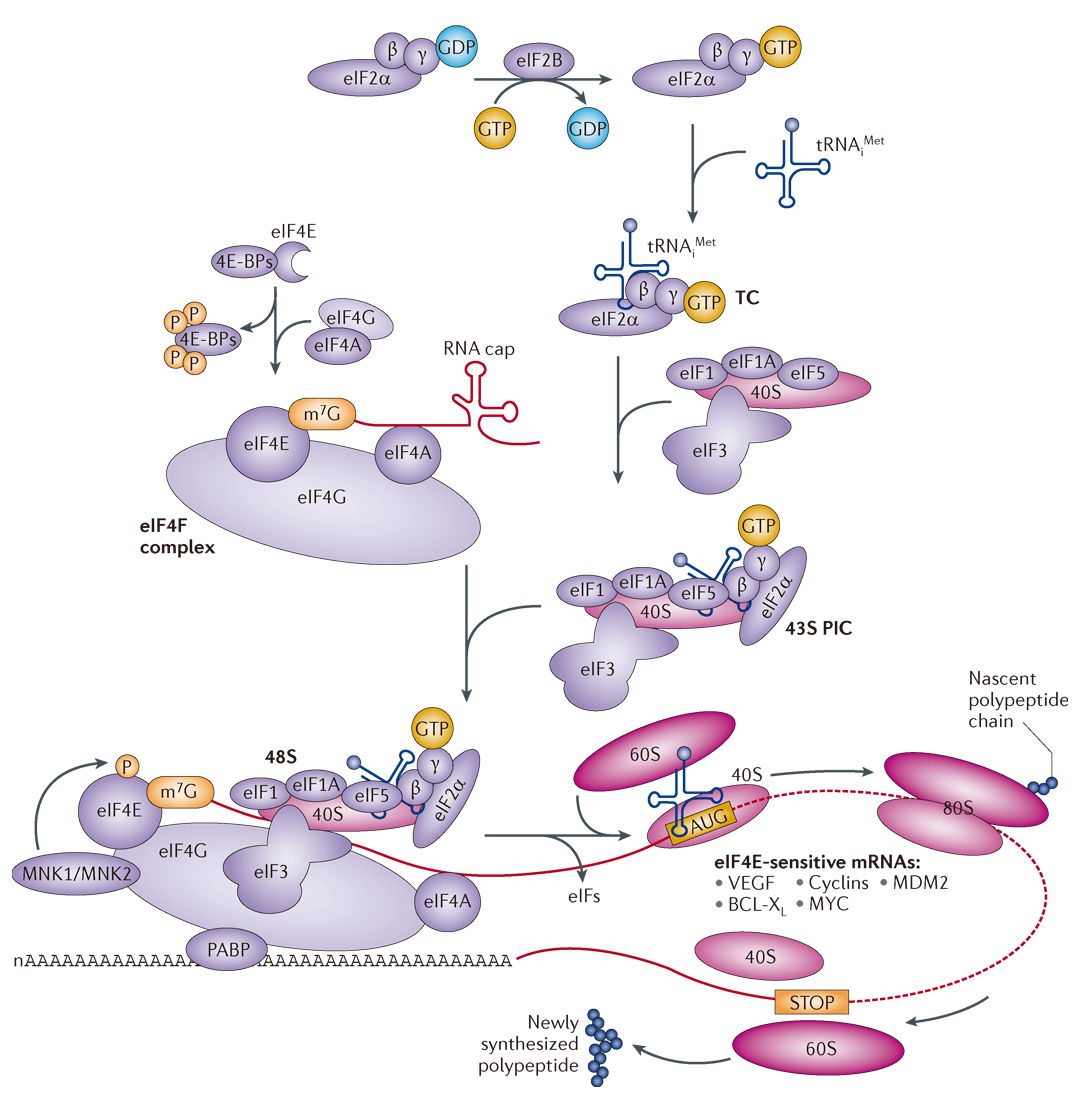 图4 翻译起始过程(Bhat et al., 2015)。
从上面的描述中可以发现,对于真核生物翻译起始过程来说m7G帽子必不可少。那么真核生物是否所有翻译过程都需要m7G帽子呢?2022年7月,杜克大学董欣年课题组在Cell杂志上发表了一篇题为“PABP/purine-rich-motif as an initiation module for cap-independent translation in pattern-triggered immunity”的研究论文,该研究发现在病原菌侵染条件下,脱帽蛋白(Decapping protein,DCP)会去除mRNA上的m7G帽子,这会导致mRNA的降解。正常来说mRNA的降解会导致抗病相关蛋白的丰度下降,从而使植物表现出感病。然而有研究发现DCP 的过表达并不会导致植物感病,这说明在生物胁迫时某些抗病基因的翻译并不会受到mRNA脱帽的影响(Yu et al., 2019)。作者进一步探索发现,在这些不受脱帽影响的mRNA的5′ UTR存在富含嘌呤的R基序(R-motif),通过实验证明R-motif招募翻译起始的必须组分(PABP、eIF4G和植物中eIF4G的异构体eIFiso4G)后正常启动抗病基因的翻译过程,因此这些R-motif可以充当IRES的作用(简单来说就是不需要m7G帽子也可以招募核糖体等翻译的必须组分从而启动翻译过程)。这一过程涉及病原体侵染信号的转递以及PABP、eIF4G和eIFiso4G的磷酸化(Wang et al., 2022)(图5),对详细过程感兴趣的读者可以阅读原文。
图4 翻译起始过程(Bhat et al., 2015)。
从上面的描述中可以发现,对于真核生物翻译起始过程来说m7G帽子必不可少。那么真核生物是否所有翻译过程都需要m7G帽子呢?2022年7月,杜克大学董欣年课题组在Cell杂志上发表了一篇题为“PABP/purine-rich-motif as an initiation module for cap-independent translation in pattern-triggered immunity”的研究论文,该研究发现在病原菌侵染条件下,脱帽蛋白(Decapping protein,DCP)会去除mRNA上的m7G帽子,这会导致mRNA的降解。正常来说mRNA的降解会导致抗病相关蛋白的丰度下降,从而使植物表现出感病。然而有研究发现DCP 的过表达并不会导致植物感病,这说明在生物胁迫时某些抗病基因的翻译并不会受到mRNA脱帽的影响(Yu et al., 2019)。作者进一步探索发现,在这些不受脱帽影响的mRNA的5′ UTR存在富含嘌呤的R基序(R-motif),通过实验证明R-motif招募翻译起始的必须组分(PABP、eIF4G和植物中eIF4G的异构体eIFiso4G)后正常启动抗病基因的翻译过程,因此这些R-motif可以充当IRES的作用(简单来说就是不需要m7G帽子也可以招募核糖体等翻译的必须组分从而启动翻译过程)。这一过程涉及病原体侵染信号的转递以及PABP、eIF4G和eIFiso4G的磷酸化(Wang et al., 2022)(图5),对详细过程感兴趣的读者可以阅读原文。
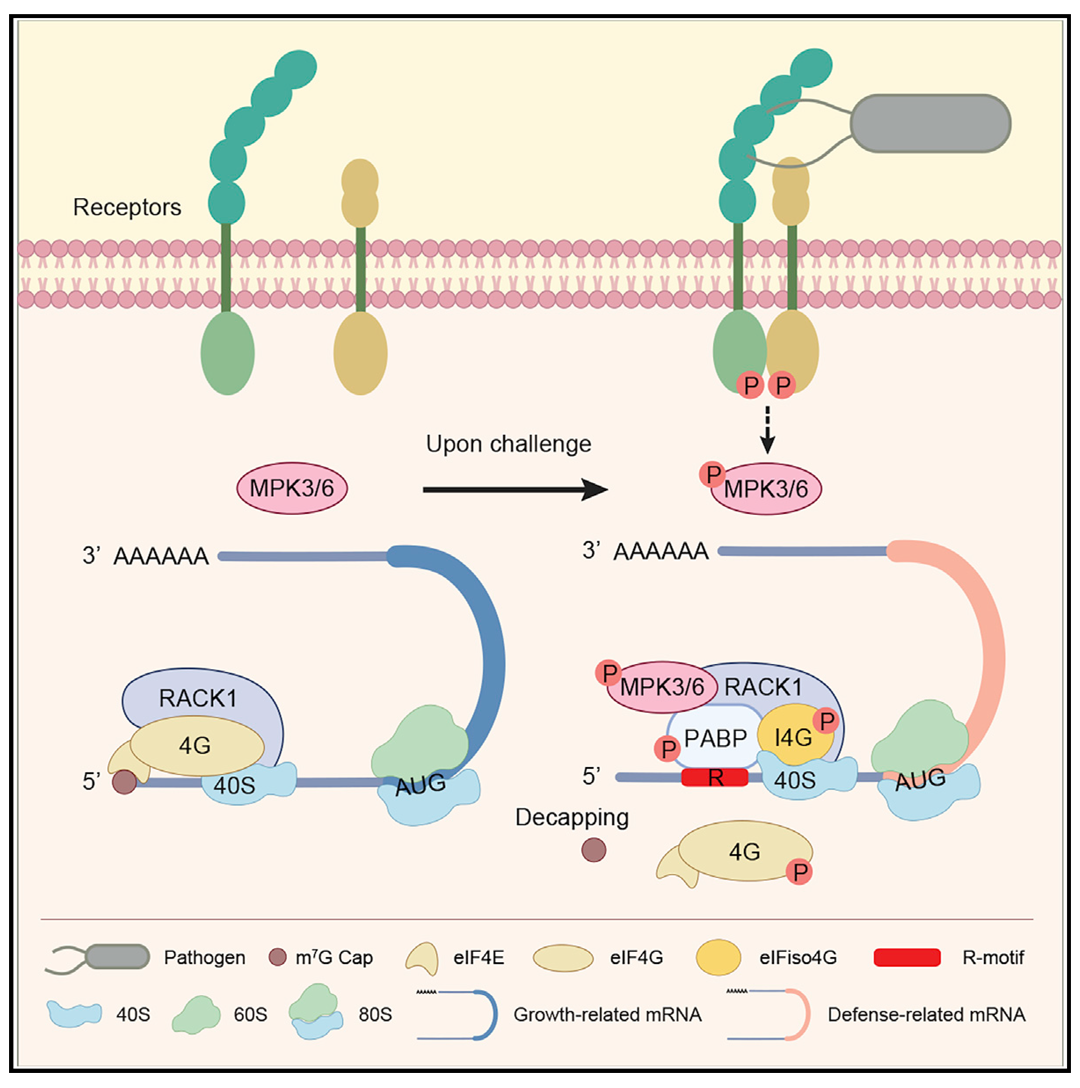 图5 IRES调控mRNA翻译过程的模式图(Wang et al., 2022)。
图5 IRES调控mRNA翻译过程的模式图(Wang et al., 2022)。
对于3′ UTR中的调控元件,伯小远本想首先给大家讲述miRNA的结合位点,因为研究miRNA对mRNA的调控,一般会关注miRNA在3′ UTR上的结合位点,该内容在往期推文“基因敲高——通过基因编辑改善作物农艺性状”有所涉及,在此就不重复分享。因此,这里就首先给大家分享ARE这种“低调”但也十分重要的调控元件。
ARE是一种位于mRNA 3′ UTR中的调控元件。ARE由富含腺嘌呤(A)和尿嘧啶(U)的短序列组成,典型的ARE通常包含多个AUUUA重复序列。这些元件的长度和确切序列可能有所不同,但它们通常富含AU两种核苷酸。ARE通常通过与特定的RNA结合蛋白相互作用,形成RNA-蛋白复合物从而调节mRNA稳定性和翻译等过程(Goss and Domashevskiy, 2016)。下面是一篇关于植物中ARE的文章。
2023年1月,福建农林大学马留银课题组联合苏州大学吴小惠课题组在Plant Physiology杂志上发表了一篇题为“Alternative 3′ UTRs regulate high salt tolerance of Spartina alterniflora”的研究论文,该研究前期发现盐胁迫下离子转运相关基因的3′ UTR会延长,这提高了这些基因mRNA的稳定性和蛋白的丰度。为了深入探索这一现象的内在分子机制,作者以这些离子转运相关基因中的SaHKT1为突破口,分析SaHKT1 3′ UTR发现,盐胁迫下SaHKT1延长的3′ UTR中发现了ARE,而未胁迫下SaHKT1的3′ UTR未见ARE,这说明ARE可能对SaHKT1提高mRNA稳定性以响应盐胁迫至关重要。通过实验证明29nt ARE的缺失确实会导致SaHKT1蛋白丰度的下降(Wang et al., 2023)(图6)。
图6 互花米草中ARE提高mRNA稳定性以响应盐胁迫的模式图(Wang et al., 2023)。
RNA的G-四链体(RNA G-quadruplex,RG4)是一种由富含鸟嘌呤(G)的序列形成的特殊二级结构。在RNA分子中,G-四链体通过碱基堆积和配位离子的稳定,形成由四个G通过氢键连接成的平面结构。多个这样的平面可以堆叠在一起,形成稳定的四链结构(Kwok and Merrick, 2017)。3′ UTR中的RG4参与调控mRNA的定位以及稳定性等,值得一提的是该结构也存在于5′ UTR(Fay et al., 2017)。相比于ARE,RG4更加“冷门”,接下来与各位读者分享一篇有关植物中G-四链体的文章。
2022年10月,中科院分子植物科学卓越创新中心杨小飞课题组、东北师范大学张铧坤课题组联合约翰英纳斯中心丁一倞课题组在Nature Communications杂志上发表了一篇题为“RNA G-quadruplex structure contributes to cold adaptation in plants”的研究论文,该论文前期研究发现冷胁迫诱导了RG4的形成,所以作者猜测RG4可能在低温感知中发挥特别的作用,这种作用很可能是抑制mRNA的降解。为了进一步探索,作者选择了一个受低温显著诱导的基因CORG1为研究对象,将CORG1 RG4中的G突变为A后获得mutRG4-CORG1突变体。实验后发现,冷胁迫条件下,mutRG4-CORG1突变体中CORG1 mRNA的降解速率高于野生型,此外,mutRG4-CORG1突变体的生长抑制弱于野生型。这些说明RG4结构通过抑制mRNA的降解从而提高了植物对冷胁迫敏感度(Yang et al., 2022)。
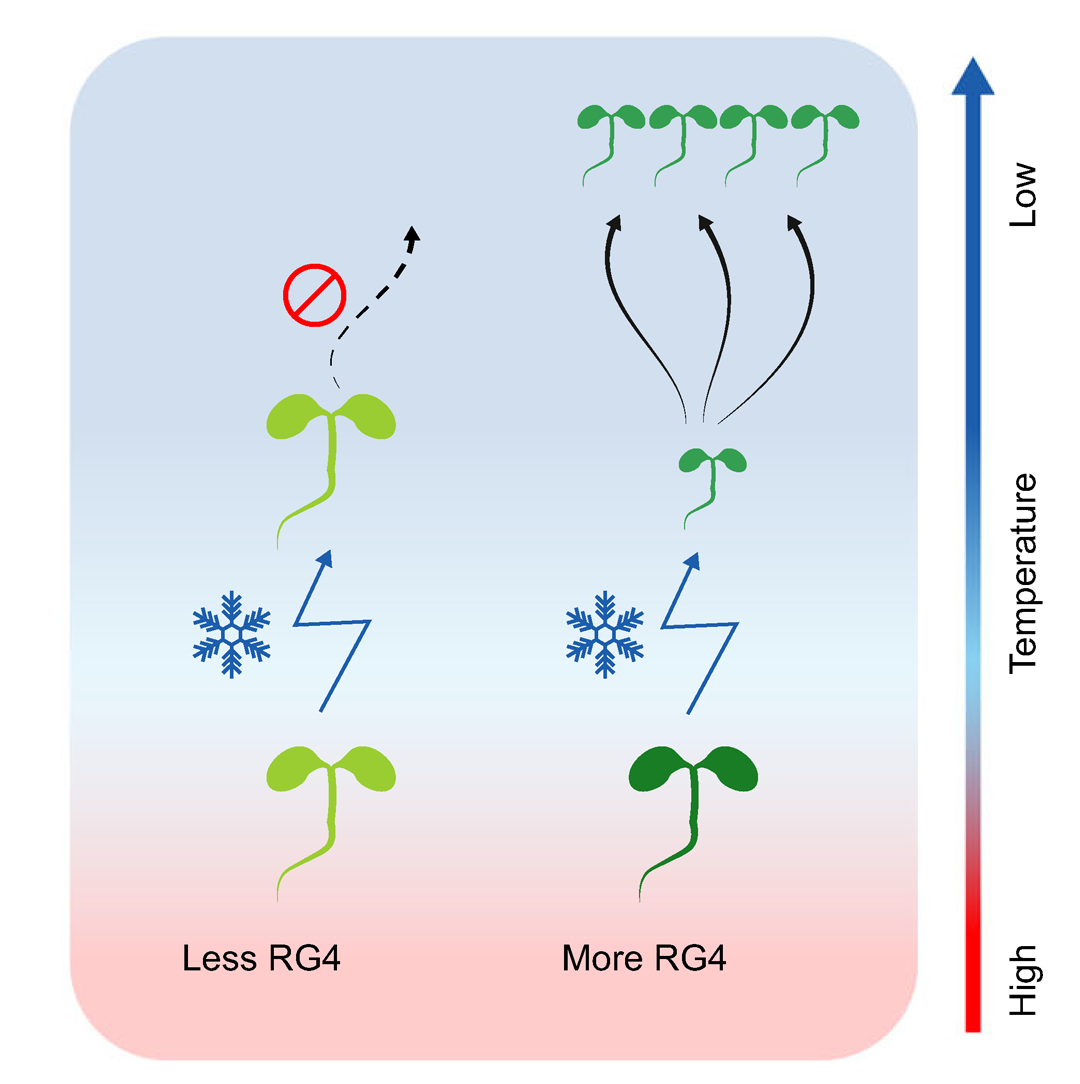
图7 RG4通过抑制mRNA的降解提高植物对冷胁迫敏感度的模式图(Yang et al., 2022)。
3′ UTR上的重要调控元件除了上面介绍的两种之外,比较常见的还有m6A修饰位点。具体来说,m6A指的是RNA分子中的腺苷(A)碱基的N6位点上附加一个甲基团(-CH3)。m6A是真核生物mRNA上含量最高的化学修饰,在基因表达调控中起着重要作用,包括转录和转录后调控(Tang et al., 2023)。相比于动物,植物中m6A修饰位点主要位于3′ UTR,而5′ UTR和CDS上较少(Wang et al., 2024)。3′ UTR及CDS上的m6A修饰主要与mRNA的稳定性有关,而5′ UTR上的m6A修饰是否与翻译效率有关还存在争议。最近有研究发现,单个m6A在5′ UTR中对翻译效率没有明显影响。由于3′ UTR上的m6A修饰比较常见,在此处讨论植物上的m6A的修饰还是主要把目光集中在3′ UTR上。
2018年4月,北京大学贾桂芳课题组在Plant Cell杂志上发表了一篇题为“The m6A Reader ECT2 Controls Trichome Morphology by Affecting mRNA Stability in Arabidopsis”的研究论文,作者发现m6A结合蛋白ECT2主要富集在mRNA的3′ UTR上,进一步探究发现3′ UTR的m6A修饰可以提高毛状体形态发生相关基因mRNA的稳定性(Wei et al., 2018)。
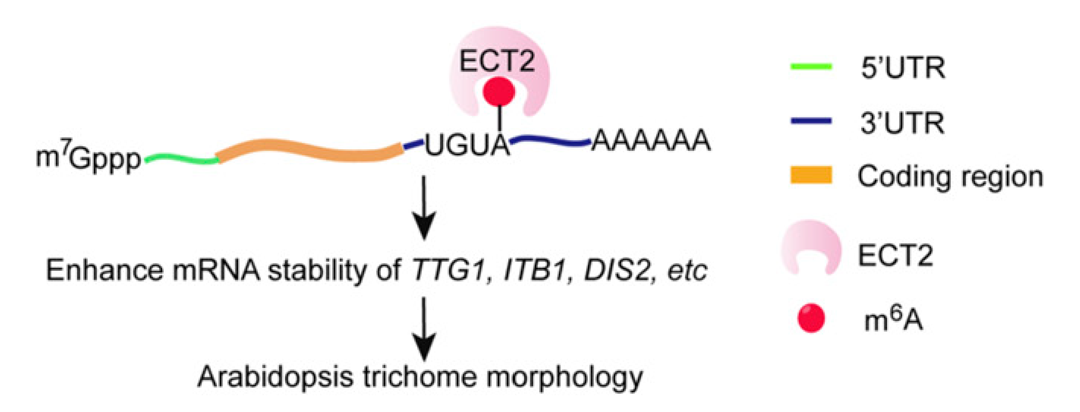
图8 3′ UTR的m6A修饰提高毛状体形态发生相关基因mRNA稳定性的模式图(Wei et al., 2018)。
本小节给大家分享了一些UTR上重要调控元件的相关研究,这些元件里有些已经逐渐走进了科研“舞台”的中心,如uORF等,而有些还徘徊在科研“舞台”的边缘,如RG4等。通过分享的文献案例大家可以了解到,这些元件在mRNA的翻译调控、稳定性及亚细胞定位相关过程中都起着重要的调控作用,由于篇幅有限的原因,并没有给大家列举所有调控元件的相关案例,例如,mRNA的定位可由RNA结合蛋白(RNA-binding proteins,RBPs)所介导,结合位置通常位于UTR和CDS(Tian et al.,2020),感兴趣的小伙伴可以自行查阅相关资料。
在植物基因功能研究过程中,研究人员通常通过基因敲除、基因沉默或过表达来调控特定基因的表达。然而,阅读本文后,相信大家也能意识到调控UTR区也是一种行之有效的方法。当实验仅需要基因敲除或过表达时,传统方法可能已经足够,但如果要求不改变编码序列且不引入外源片段,就需要考虑其他策略。通过改造UTR区中的重要调控元件,不仅可以满足要求,还可以实现基因的上调及下调,这对品种改良和实际推广具有重要意义。总之,随着基因编辑技术的发展和基础研究的进步,越来越多的优良基因被发现。为了将这些基因应用于实际育种中,UTR区中的重要调控元件将成为一个重要的突破口。
Bhat M, Robichaud N, Hulea L, et al. Targeting the translation machinery in cancer[J]. Nature reviews Drug discovery, 2015, 14(4): 261-278.
Fay M M, Lyons S M, Ivanov P. RNA G-quadruplexes in biology: principles and molecular mechanisms[J]. Journal of molecular biology, 2017, 429(14): 2127-2147.
Goss D J, Domashevskiy A V. Messenger RNA (mRNA): The link between DNA and protein[J]. Encyclopedia of Cell Biology, 2016.
Hardy E C, Balcerowicz M. Untranslated yet indispensable—UTRs act as key regulators in the environmental control of gene expression[J]. Journal of Experimental Botany, 2024: erae073.
Kwok C K, Merrick C J. G-quadruplexes: prediction, characterization, and biological application[J]. Trends in biotechnology, 2017, 35(10): 997-1013.
Morinaka Y, Sakamoto T, Inukai Y, et al. Morphological alteration caused by brassinosteroid insensitivity increases the biomass and grain production of rice[J]. Plant physiology, 2006, 141(3): 924-931.
Niu R, Zhou Y, Zhang Y, et al. uORFlight: a vehicle toward uORF-mediated translational regulation mechanisms in eukaryotes[J]. Database, 2020, 2020: baaa007.
Tang J, Chen S, Jia G. Detection, regulation, and functions of RNA N6-methyladenosine modification in plants[J]. Plant communications, 2023, 4(3).
Tian L, Chou H L, Fukuda M, et al. mRNA localization in plant cells[J]. Plant physiology, 2020, 182(1): 97-109.
Wang G, Li H, Ye C, et al. Quantitative profiling of m6A at single base resolution across the life cycle of rice and Arabidopsis[J]. Nature Communications, 2024, 15(1): 4881.
Wang J, Liu J, Guo Z. Natural uORF variation in plants[J]. Trends in Plant Science, 2023.
Wang J, Zhang X, Greene G H, et al. PABP/purine-rich motif as an initiation module for cap-independent translation in pattern-triggered immunity[J]. Cell, 2022, 185(17): 3186-3200. e17.
Wang T, Ye W, Zhang J, et al. Alternative 3′-untranslated regions regulate high-salt tolerance of Spartina alterniflora[J]. Plant Physiology, 2023, 191(4): 2570-2587.
Wei L H, Song P, Wang Y, et al. The m6A reader ECT2 controls trichome morphology by affecting mRNA stability in Arabidopsis[J]. The Plant Cell, 2018, 30(5): 968-985.
Xue C, Qiu F, Wang Y, et al. Tuning plant phenotypes by precise, graded downregulation of gene expression[J]. Nature Biotechnology, 2023, 41(12): 1758-1764.
Yang X, Yu H, Duncan S, et al. RNA G-quadruplex structure contributes to cold adaptation in plants[J]. Nature communications, 2022, 13(1): 6224.
Yu X, Li B, Jang G J, et al. Orchestration of processing body dynamics and mRNA decay in Arabidopsis immunity[J]. Cell Reports, 2019, 28(8): 2194-2205. e6.











