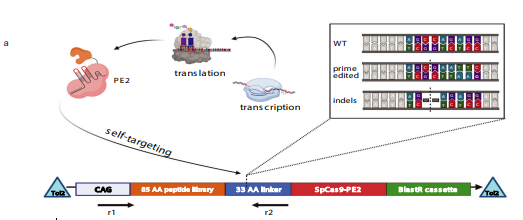
prime editing是一种“搜索和替换”的基因组编辑技术,可以介导靶向的插入、缺失以及所有的碱基替换,而且它可以将不同类型的编辑相结合。所有这些都可以在没有双链断裂(DSB)或供体DNA模板的情况下进行。它的原理如何?首先,将经过改造的向导RNA(pegRNA)与prime editor蛋白相结合,pegRNA具有双重功能,既能够将编辑蛋白引导到目标位点,又含有编辑的模板序列。prime editor蛋白则由Cas9切口酶和逆转录酶融合而成。在Cas9切割目标位点之后,逆转录酶以pegRNA作为模板进行逆转录,然后将DNA直接聚合到切口的DNA链上。prime editor不大受PAM序列的限制,可实现所有12种可能的碱基替换,扩展了CRISPR基因组编辑的范围,但该技术目前最重要的问题是编辑效率低。
2022年6月18日,Nature Communications杂志在线发表了题为“Peptide fusion improves prime editing efficiency”的研究论文,该研究提出了一种高通量方法,即肽自编辑测序测定法 (PepSEq),用来测量多个氨基酸肽的不同融合是如何影响主要编辑效率,该研究筛选出高效的双肽并证实双肽融合后的编辑器IN-PE2编辑效率显著,为基因编辑器效率的提升提供了一种新的研究思路。
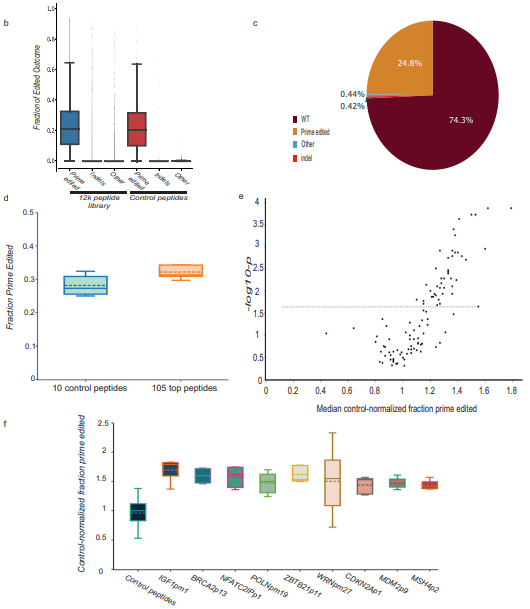
以往文献证实,肽和蛋白质融合是一种行之有效的调节基因组编辑结果的方法,源自 DNA 修复相关蛋白的肽的编码可能能够改变prime editing编辑效率的结构域。虽然可扩展、敏感的蛋白质融合筛选仍然具有挑战性,但高通量寡核苷酸文库合成能够筛选高度多样化的肽融合构建体。基于此,作者设计了一个包含 85 个氨基酸肽的文库, 417 个 DNA 修复相关蛋白和 29 个管家基因作为对照,以及 5458 个 DNA 修复相关的突变肽。将这个 12,000 个寡核苷酸的文库在 N 端克隆到 33 个氨基酸的 XTEN 接头并在载体中克隆 PE2,利用 Tol2 转座子介导的基因组整合,以筛选附加到 PE2 的 N 端时可提高编辑效率的肽。
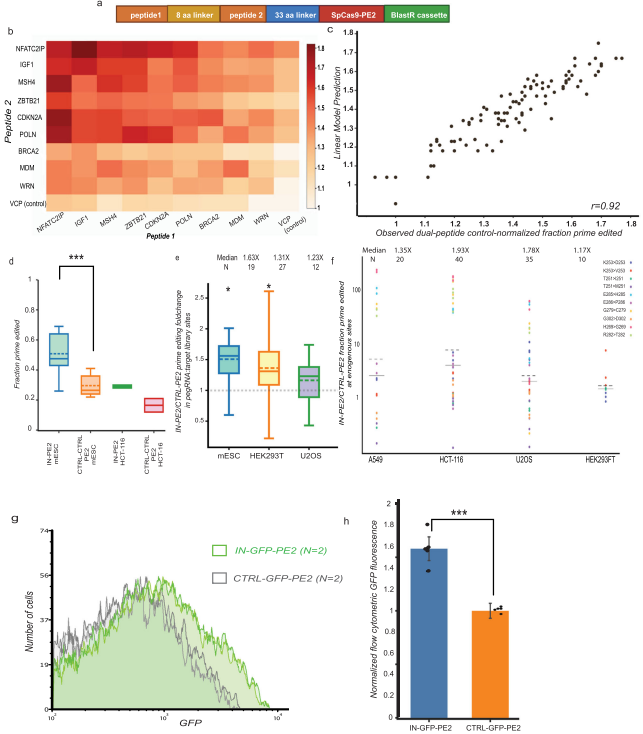
为了能够在高通量中定量评估肽-PE2 编辑效率,作者开发了肽自编辑测序测定法 (PepSEq),即通过一种自我靶向 pegRNA,它在肽相邻接头序列(sgPE-接头)中引入了一个 6 nt 突变(CCTCTG-> GAATTC)。为了评估汇集格式的prime编辑效率,作者进行了配对末端下一代测序(NGS)并利用 β-二项式模型分析出了具有独立肽-PE2 整合的三个组合重复,确定了 105个最佳候选prime editing的增强肽。
结果发现,有两种肽在单肽-PE2 筛选和双肽-PE2 筛选的线性模型中增强了prime editing的效率, 即IN-PE2。研究发现,在所有靶向基因座中,与 PE2 相比,IN-PE2 处理在所有细胞系中产生高效编辑。GFP 荧光强度与初始编辑率呈正相关,表明较高水平的 PE2 导致肽增强的初始编辑增加,这一结果表明IN肽增加了PE2酶的转录或翻译,并为IN-PE2活性增加提供了合理的解释。另一方面,作者克隆了一个构建体,其中 IN 肽通过自切割 2A 肽 (IN-P2A-PE2) 与 PE2 分离。在 mESCs 中使用 PepSEq 测定,我们发现与 CTRL-PE2 相比,IN-P2A-PE2 可显着改善初始编辑,编辑效率与 IN-PE2 相当。总而言之,作者推测IN 肽通过增加翻译起作用提高了编辑效率。但这些肽如何增加翻译仍然是一个悬而未决的问题,未来也可使用PepSEq扫描诱变来发现对 IN 肽功能至关重要的残基。