本文内容速览:
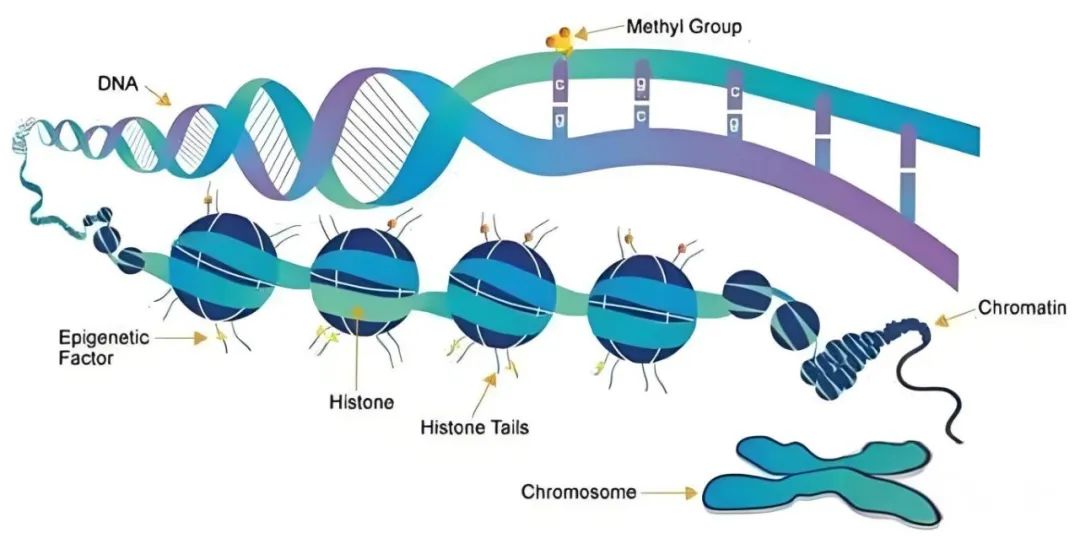
图1 染色质结构。图片来源:”What is epigenetics”网站。
常见的表观组学技术如下:
染色质免疫共沉淀测序技术(Chromatin immunoprecipitation and sequencing, ChIP-seq):在全基因组范围内检测与转录因子或组蛋白互作的DNA区段的测序分析技术,可用于寻找转录因子调控的下游基因、组蛋白修饰关联的基因、检测增强子或启动子的组蛋白修饰标志(H3K4me1、H3K4me3或H3K27ac)等,还可比较转基因与野生型材料中转录因子结合位点或组蛋白修饰位点的差异来研究目的基因功能。
靶向剪切及标签技术(Cleavage under targets and tagmentation, CUT&Tag):在全基因组范围内检测与转录因子或组蛋白互作的DNA区段的测序分析技术,是可替代ChIP-seq的技术,与ChIP-seq适用内容相同。
DNA亲和纯化测序技术(DNA affinity purification and high-throughput sequencin, DAP-seq):在全基因组范围内检测转录因子结合位点的体外测序分析技术,可用于寻找转录因子调控的下游基因。
全基因组亚硫酸氢盐测序技术(whole-genome bisulfite sequencing, WGBS/BS-seq):在全基因组范围内检测DNA甲基化位点的测序分析技术,可用于寻找甲基化修饰的关联基因,还可比较转基因与野生型材料中DNA甲基化修饰位点的差异来研究目的基因功能。
表观组学通常可以和转录组学进行联合分析以获得目的基因或寻找转录因子调控的下游基因,本文所说的转录组主要是指检测mRNA表达水平的RNA-seq,全转录组或者非编码RNA组(例如:miRNA组、lncRNA组、circRNA组等)和表观组学联合分析的思路与表观组学和转录组联合分析相似,不做详细介绍。
图2 ATAC-seq和RNA-seq数据Venn图(改编自Di et al., 2023)。
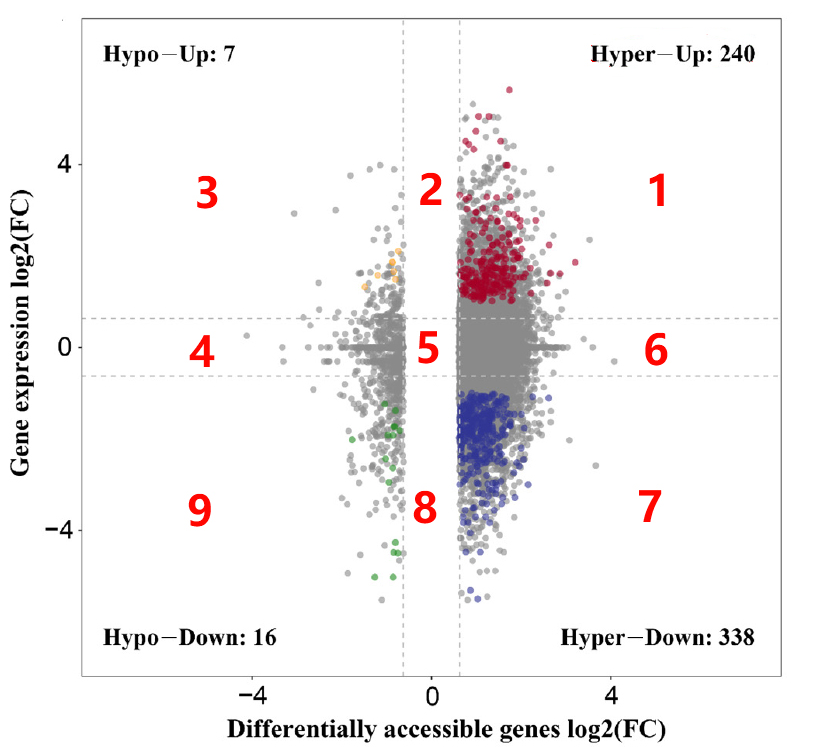
但Venn图展示的内容有限,如果两个组学都有差异比较组,想知道基因在两个组学中共上调或者共下调情况还需要分别对两个组学上调或下调的基因集取交集,但也有基因在两个组学中的上下调情况相反或者仅在一个组学中差异显著,这就需要四/九象限图(图3)。
在第1象限中基因的染色质可及性程度提高和表达水平上调,这表明转录因子可能激活这些基因的表达。在第9象限中基因的染色质可及性程度降低和表达水平下调,这说明基因在特定细胞类型或条件下复制或转录等过程都不活跃。而在第3象限和第7象限中基因的染色质可及性程度与表达水平的上下调情况相反,第3象限的结果可能意味着存在其他转录因子或调控机制能够激活这些基因的表达,第7象限的结果可能表明转录抑制因子抑制了这些基因的表达,或者需要其他协同因子共同调控才能激活基因的表达。在第2象限和第8象限中基因表达水平的变化显著而染色质可及性的程度变化不显著,在第4象限和第6象限中的基因与之相反。在第5象限中基因的染色质可及性程度和表达水平差异都不显著。其中第2/5/8象限中的基因(灰色)并没有展示出来,后续对第1/3/7/9象限中的基因进行了功能富集分析,并使用红/黄/绿/蓝四种不同颜色标记出了具有特定功能的基因。
图3 ATAC-seq和RNA-seq联合分析象限图(改编自Wang et al., 2022)。横坐标为基因的染色质可及性的差异倍数的log2值,纵坐标为基因表达水平的差异倍数的log2值。差异倍数(Fold Change, FC)>1。
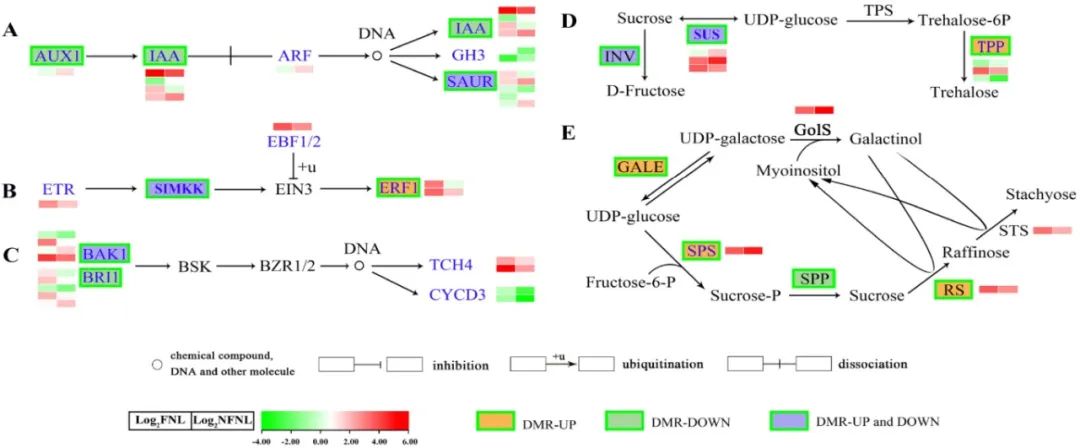
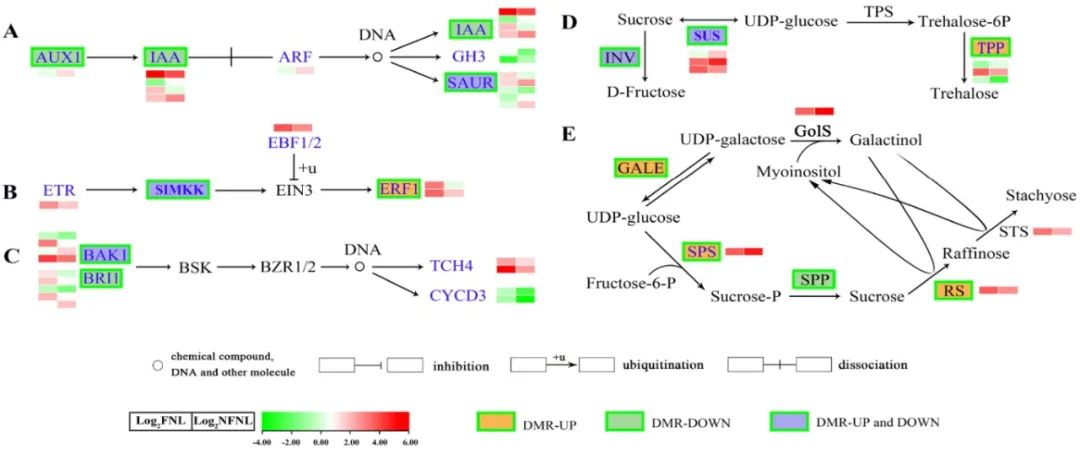
图4 作物产量取决于获得同化物(sink,库)的潜力和供给同化物(source,源)的能力。与“库-源”调控相关的代谢通路中差异甲基化区域和DEGs(Wang et al., 2023)。
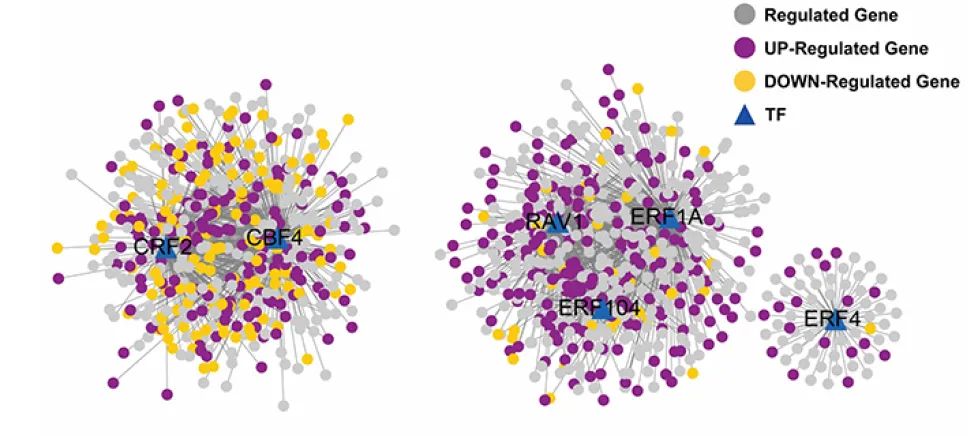
图6 基因共表达网络(Ren et al., 2021)。将ATAC-seq数据中获得的6个转录因子和RNA-seq数据中获得的DEGs构建成两个独立的调控网络(CBF4-CRF2和RAV1-ERFs)。转录因子用三角形表示,预测的转录因子调控的下游基因用圆形表示。DEGs分别以紫色(上调)和橙色(下调)表示。
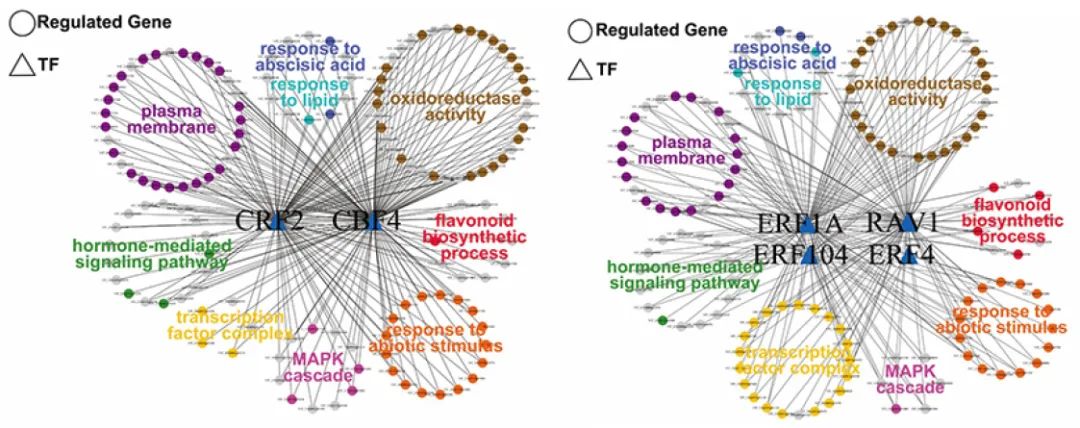
图7 CBF4-CRF2和RAV1-ERFs共表达网络中共有的GO富集分析结果(Ren et al., 2021)。
2020年8月,中国科学院华南植物园段学武课题组在New Phytologist上发表了一篇题为“Histone demethylase SlJMJ6 promotes fruit ripening by removing H3K27 methylation of ripening-related genes in tomato”的研究论文。在该文章中,作者发现番茄(Solanum lycopersicum)SlJMJ6编码一种组蛋白赖氨酸去甲基化酶,可以特异性地促使H3K27me3去甲基化,激活成熟相关基因的表达,从而促进番茄果实成熟。作者还揭示了组蛋白去甲基化和DNA去甲基化在调节果实成熟过程中的新联系,这是第一个报道关于组蛋白赖氨酸去甲基化酶参与果实成熟调节的文章。
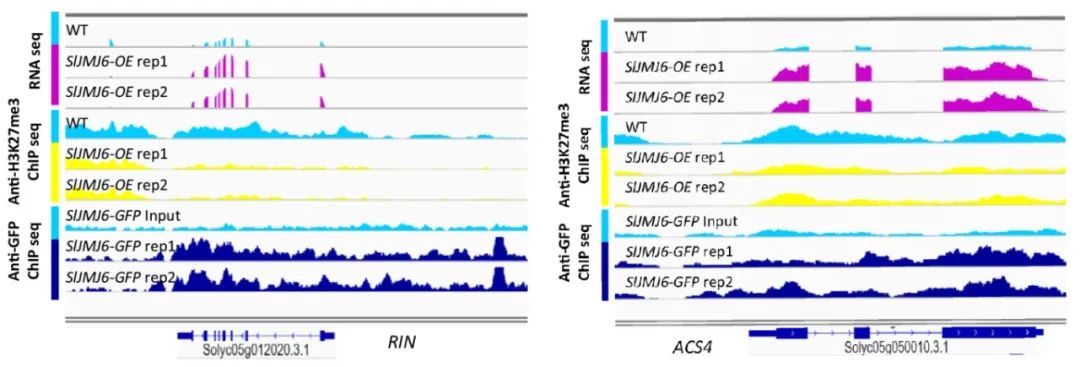
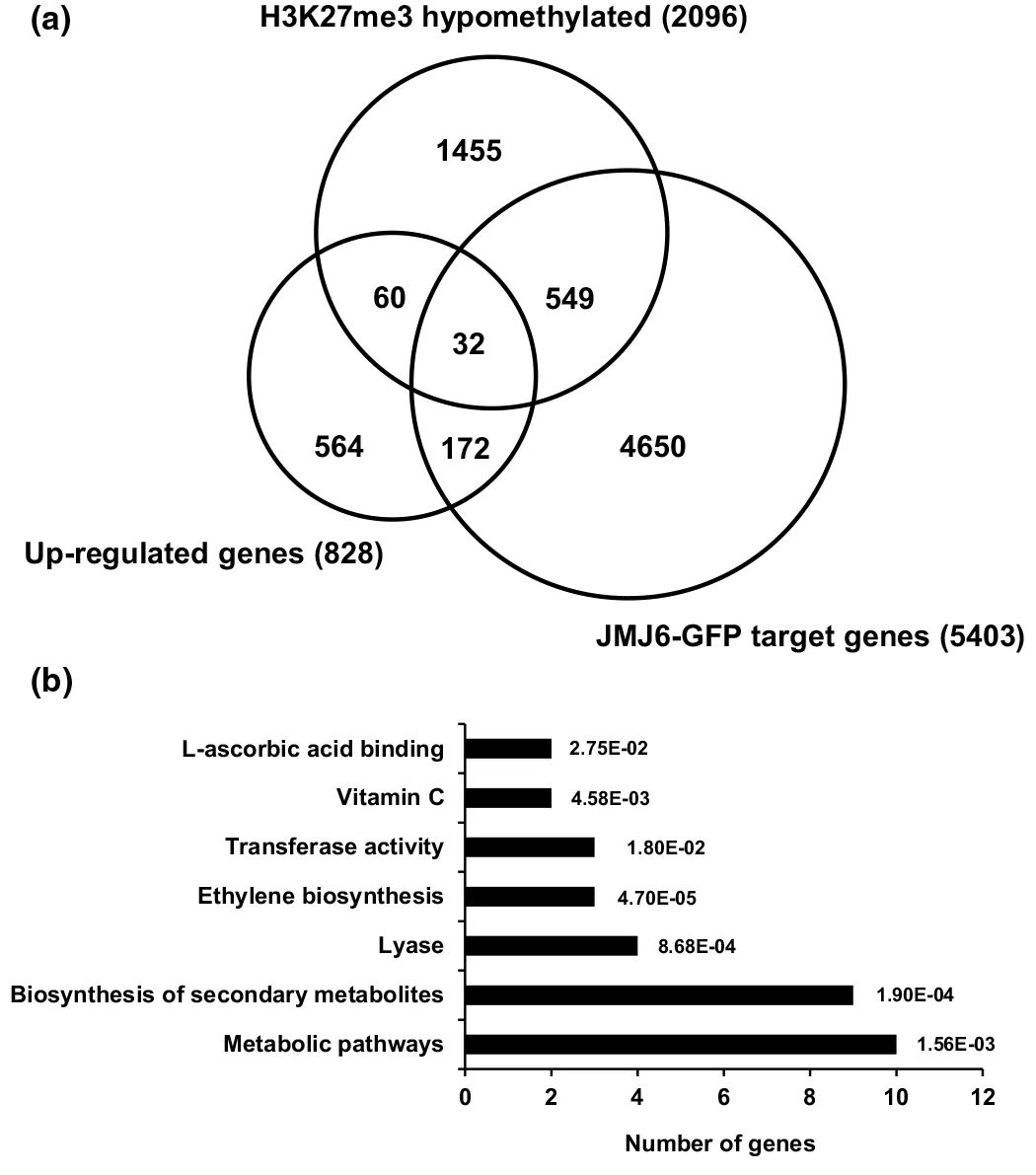
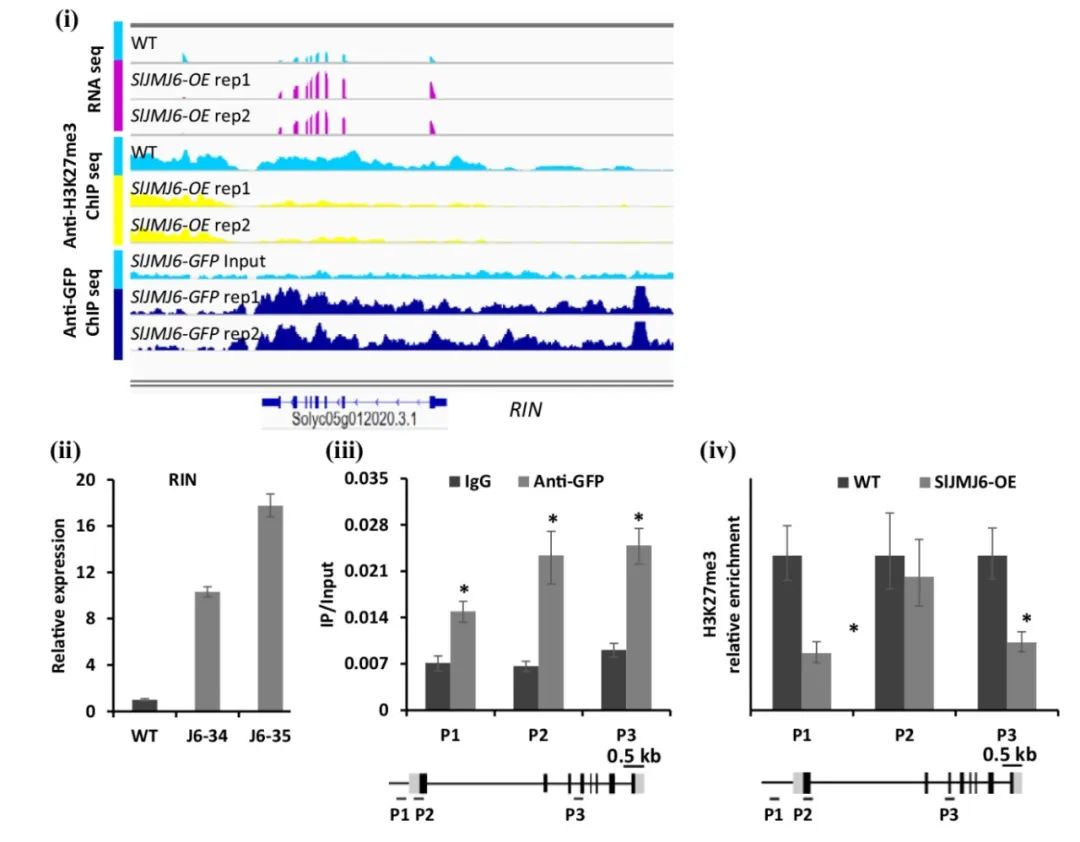
通过RNA-seq和ATAC-seq的联合分析,作者寻找到了32个SlJMJ6下游的调控基因,在SlJMJ6过表达的番茄果实中这些基因的转录上调,同时,这些基因座位上的H3K27m3修饰水平降低,它们主要参与转录调控、乙烯生物合成、细胞壁降解和激素信号传导等过程(图8)。RT-qPCR和ChIP-qPCR的结果进一步证实了11个与成熟相关的基因如RIN、ACS4、ACO1、PL和DML2被SIJMJ6通过H3K27me3去甲基化的方式直接调控(图9)。
图8 ChIP-seq与RNA-seq联合分析(Li et al., 2020)。(a)SlJMJ6结合位点的关联基因(即SlJMJ6下游的调控基因)、SlJMJ6过表达的番茄果实中转录上调的基因以及H3K27me3去甲基化的关联基因这三者的Venn图,图中显示出三者有32个共有基因;(b)32个共有基因的GO富集分析。
图9 H3K27me3的去甲基化与SlJMJ6介导的成熟相关基因的表达水平上调有关(Li et al., 2020)。(i)SlJMJ6介导的成熟相关基因RIN在SlJMJ6过表达和野生型植株中的表达水平、基因座位上的H3K27me3修饰水平以及SlJMJ6结合位点这三者的基因组可视化图谱;(ii)RT-qPCR分析RIN在野生型和过表达植株中的表达水平;(iii)ChIP-qPCR分析RIN座位上SlJMJ6的结合情况;(iv)ChIP-qPCR分析RIN座位上H3K27me3的修饰情况。
这是一篇典型的联合分析案例,ChIP-seq与RNA-seq联合分析不仅获得了SlJMJ6下游的调控基因,由于联合分析了H3K27me3的去甲基化情况,还说明了SlJMJ6通过H3K27me3的去甲基化激活了成熟相关基因的表达,从而促进番茄果实成熟。看来联合分析不仅可以获得基因,还可以用来研究基因的功能。
2021年8月,中国科学院植物研究所梁振昌课题组在Plant & cell physiology发表了一篇题为“Characterization of Chromatin Accessibility and Gene Expression upon Cold Stress Reveals that the RAV1 Transcription Factor Functions in Cold Response in Vitis Amurensis”的研究论文。在该文章中,作者使用ATAC-seq和RNA-seq来寻找具有高耐寒性葡萄品种Vitis amurensis的寒冷响应性转录因子,共找出CBF4、RAV1和ERF104等9个转录因子。其中,VaRAV1在葡萄细胞中的过表达提高了其耐寒性。该工作为植物对寒冷响应提供了新的见解,并证明了ATAC-seq和RNA-seq快速鉴定葡萄寒冷响应性转录因子的实用性。VaRAV1转录因子可能在植物适应寒冷环境过程中起重要作用。
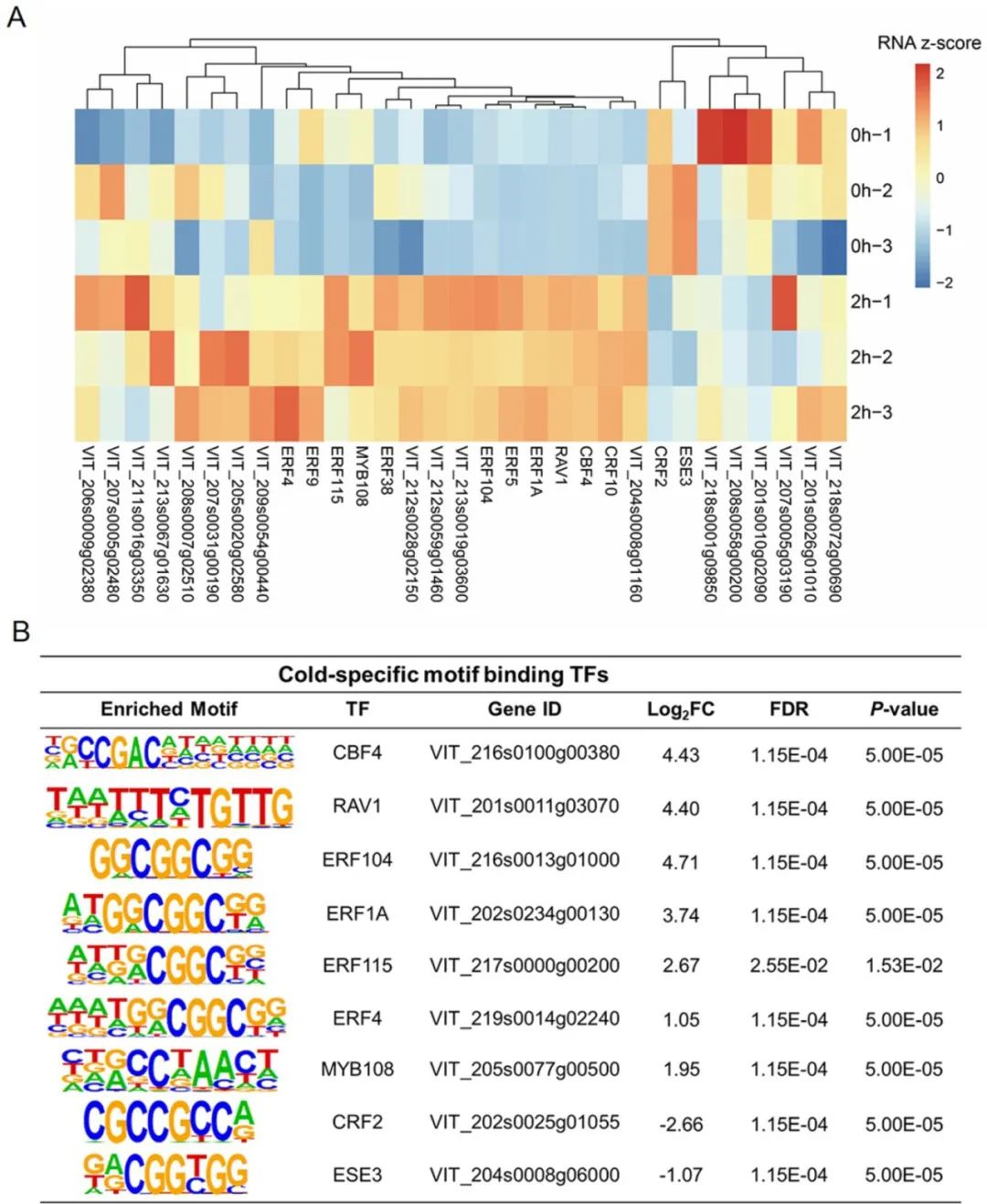
首先,作者利用ATAC-seq绘制了葡萄的染色质可及性图谱,Peak或者是转座酶超敏位点(Transposase hypersensitive sites, THSs)表示富集的染色质可及性区域,在寒冷处理2h后的葡萄样品中共鉴定了1376个正富集和189个负富集的THSs(“正富集”的THSs可以理解为寒冷处理2h与未处理相比某些染色质区域的可及性程度提高,“负富集”的THSs可以理解为寒冷处理2h与未处理相比某些染色质区域的可及性程度降低)。对这些THSs进行了motif(转录因子碱基偏好性)分析并搜索植物转录因子数据库,在77个motif中寻找出了CBF家族转录因子、MYB家族转录因子和AP2/ERF超家族转录因子的motif,这些转录因子可能在葡萄的寒冷响应中发挥重要作用。结合RNA-seq数据结果,最终获得了31个寒冷处理后染色质可及性改变并且表达水平发生改变的转录因子(图10A),其中有9个转录因子表现出显著的表达变化(图10B)。寒冷处理明显降低了CRF2和ESE3的表达,而其他基因在寒冷处理下表达水平上调。
图10 候选的转录因子对寒冷处理的响应(Ren et al., 2021)。(A)冷处理后TF基因表达热图。对寒冷处理后差异显著的THSs进行motif分析获得的转录因子,进一步根据其在冷处理后的表达进行筛选。对RNA-seq获得的转录因子表达数据采用Z-score归一化处理;(B)最有可能在早期寒冷响应中起作用的9个转录因子。
为了获得这些转录因子的可能靶基因,对RNA-seq结果进行了权重基因共表达网络分析(Weighted gene co-correlation network analysis, WGCNA)。根据结果,高度相关的基因富集在相同的模块中。作者将9个转录因子模块中的6个成功地分为两个代表性共表达网络:CBF-CRF2依赖性以及RAV1-ERFs依赖性调节网络(图11)。在第一个网络中,CBF4和CRF2作为核心基因。在二个网络中,RAV1、ERF1A、ERF104和ERF4作为核心基因,表明这些转录因子可能通过至少两种不同的信号通路参与葡萄寒冷响应过程。
图11 转录因子及其靶基因的共表达网络以及GO富集分析结果(Ren et al., 2021)。(A)CBF4-CRF2和RAV1-ERFs共表达网络中共有的GO富集结果;(B)CBF4-CRF2和RAV1-ERFs共表达网络中特异的GO富集结果。转录因子用三角形表示,预测的转录因子调控的下游基因用圆形表示。
由于CBFs在寒冷响应中的作用已在包括葡萄在内的许多植物物种中得到很好的验证,作者最终选择RAV1、ERF1A和CRF2作为候选基因来验证它们在葡萄耐寒中的功能,其中VaRAV1在葡萄细胞中的过表达提高了其耐寒性。
这篇论文没有直接寻找ATAC-seq和RNA-seq的共有基因,而是先对ATAC-seq结果中染色质可及性显著变化的转录因子进行注释,再根据RNA-seq数据有针对性地选择其中表达水平有显著变化的转录因子进行功能验证,是ATAC-seq和RNA-seq联合分析来获得目的基因非常好的案例哦!
3.3 BS-seq与RNA-seq联合分析案例
作物产量取决于获得同化物(sink,库)的潜力和供给同化物(source,源)的能力。“库-源”关系的优化对作物产量调控具有重要意义。黄瓜是一种典型的棉子糖家族低聚糖(Raffinose family oligosaccharide, RFO)转运作物。DNA甲基化是植物中一种常见的表观遗传修饰,但其在“库-源”调控中的作用尚未在RFO转运物种中得到证实。
2023年12月,扬州大学缪旻珉课题组在Plants上发表了一篇题为“The Sink-Source Relationship in Cucumber (Cucumis sativus L.) Is Modulated by DNA Methylation”的研究论文。研究结果基于BS-seq和RNA-seq为DNA甲基化在“库-源”关系中的潜在作用提供了最新的推断,这将为进一步探索DNA甲基化在提高RFO转运植物产量中的分子机制提供重要参考。
研究通过BS-seq和RNA-seq比较了两种处理下的叶片:第12节点的非结果节叶(Nonfruiting-Node Leaves,NFNL)和坐果叶(leaves of fruit setting,FNL)。BS-seq结果显示大量甲基化差异显著的关联基因在光合作用和碳水化合物代谢过程中富集。BS-seq与RNA-seq联合分析表明,许多具有差异甲基化区域的DEGs参与生长素,乙烯和油菜素内酯的代谢、蔗糖代谢、以及与“库-源”调控相关的RFO合成通路(图12)。
图12 与库源调控相关的代谢通路中差异甲基化区域和DEGs(Wang et al., 2023)。
该文章虽然没有选择目的基因进行功能验证,但是联合分析结果为获得目的基因提供了线索,完全可以往下进行基因功能验证哦!
Di X, Xiang L, Jian Z. YAP-mediated mechanotransduction in urinary bladder remodeling: Based on RNA-seq and CUT&Tag[J]. Frontiers in Genetics, 2023, 14: 1106927.
Li Z, Jiang G, Liu X, et al. Histone demethylase SlJMJ6 promotes fruit ripening by removing H3K27 methylation of ripening‐related genes in tomato[J]. New Phytologist, 2020, 227(4): 1138-1156.
Ren C, Li H, Wang Z, et al. Characterization of chromatin accessibility and gene expression upon cold stress reveals that the RAV1 transcription factor functions in cold response in Vitis amurensis[J]. Plant and Cell Physiology, 2021, 62(10): 1615-1629.
Wang S, He J, Deng M, et al. Integrating ATAC-seq and RNA-seq reveals the dynamics of chromatin accessibility and gene expression in apple response to drought[J].International Journal of Molecular Sciences, 2022, 23(19): 11191.
Wang Y, Zhang H, Gu J, et al. The Sink-Source Relationship in Cucumber (Cucumis sativus L.) Is Modulated by DNA Methylation[J]. Plants, 2023, 13(1): 103.