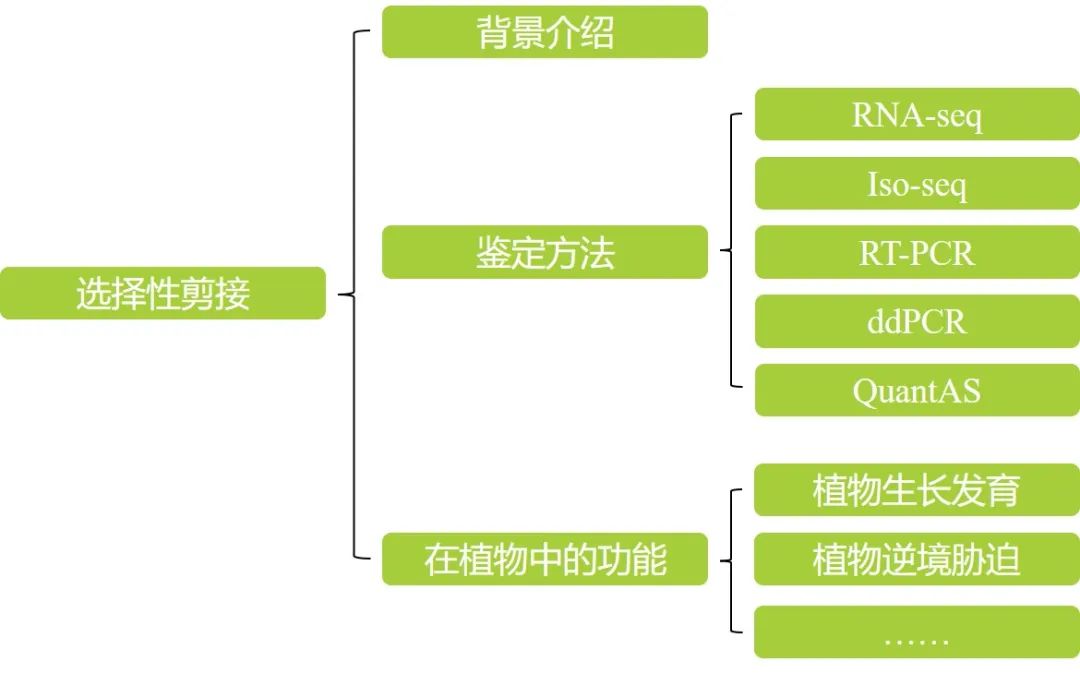
本文内容速览:
选择性剪接(可变剪接)是一种转录后调控机制,它通过在剪接过程中选择不同的剪接位点,使单个基因能够产生多种不同的mRNA亚型,从而产生多种蛋白质亚型。这种复杂的调控机制在植物生长发育、生物和非生物胁迫响应等各种生命活动中发挥着至关重要的作用。今天就让我们紧跟伯小远的步伐,一同去探索基因选择性剪接的奥秘吧!
剪接可分为组成性剪接(Conventional splicing)和选择性剪接(Alternative splicing, AS)两种类型。组成性剪接是指编码蛋白质的不连续基因通过RNA剪接将内含子从mRNA前体(pre-mRNA)中依次除去,规范地将外显子连接起来形成成熟mRNA的过程。这种剪接方式只产生一种成熟的mRNA,一般也只产生一种蛋白质。选择性剪接是指用不同的剪接方式从一个pre-mRNA中产生不同的成熟mRNA,由此产生的不同的mRNA可能被翻译成不同的蛋白质。因此,一个基因可能编码多种蛋白质。
剪接过程是由一种高分子量的剪接体介导完成的。剪接体是由U1、U2、U4、U5和U6五种小核糖核酸蛋白(Small nuclear ribonucleoprotein, snRNPs)和多种非snRNP蛋白质组成的一种复合体。snRNPs和其他复合体蛋白质相互作用,形成功能复杂的结构,确保剪接体能够正确地定位和识别剪接位点,完成剪接过程。
pre-mRNA的剪接是一个复杂而精密的过程,不同的剪接方式可以产生不同的mRNA亚型,从而增加了蛋白质的多样性。下面我们来看看pre-mRNA剪接的主要过程。
(1)U1 snRNP的识别:U1 snRNP与pre-mRNA的5’端外显子结合,并以ATP依赖性方式识别5’剪接位点。在这一过程中丝氨酸/精氨酸富集蛋白(Serine/Arginine-rich proteins, SR蛋白)与异质核核糖核蛋白(Heterogeneous nuclear ribonucleoproteins, hnRNPs)相互作用,SR蛋白识别并结合在外显子剪接增强子(Exonic splicing enhancer, ESE)上促进U1 snRNP识别5’剪接位点,而hnRNPs识别并结合在外显子剪接沉默子(Exonic splicing silencer, ESS)上阻碍U1 snRNP识别5’剪接位点。
(2)U2 snRNP的结合:U2 snRNP结合到内含子的分支位点形成剪接体前体。这是剪接的关键步骤,导致内含子的分离。
(3)U4/U5/U6 snRNP的组装:U4/U5/U6 snRNP与剪接体前体复合物相互作用组装成完整的剪接体。
(4)剪接体的催化活化:剪接体的催化活化通过两步酯交换反应完成。U2 snRNP招募十九号复合物(Nineteen Complex, NTC)和十九号复合物相关蛋白(Nineteen-Related Complex, NTR),进而释放U1和U4,然后打破剪接位点处的磷酸二酯键,产生两个相邻的外显子以及一个套索结构的内含子。
(5)剪接体的解除和剪接完成:内含子和snRNPs从剪接体复合物中释放出来,5’方向上的外显子攻击套索结构,去除内含子,并将相邻外显子连接起来完成剪接,最终生成成熟的mRNA。
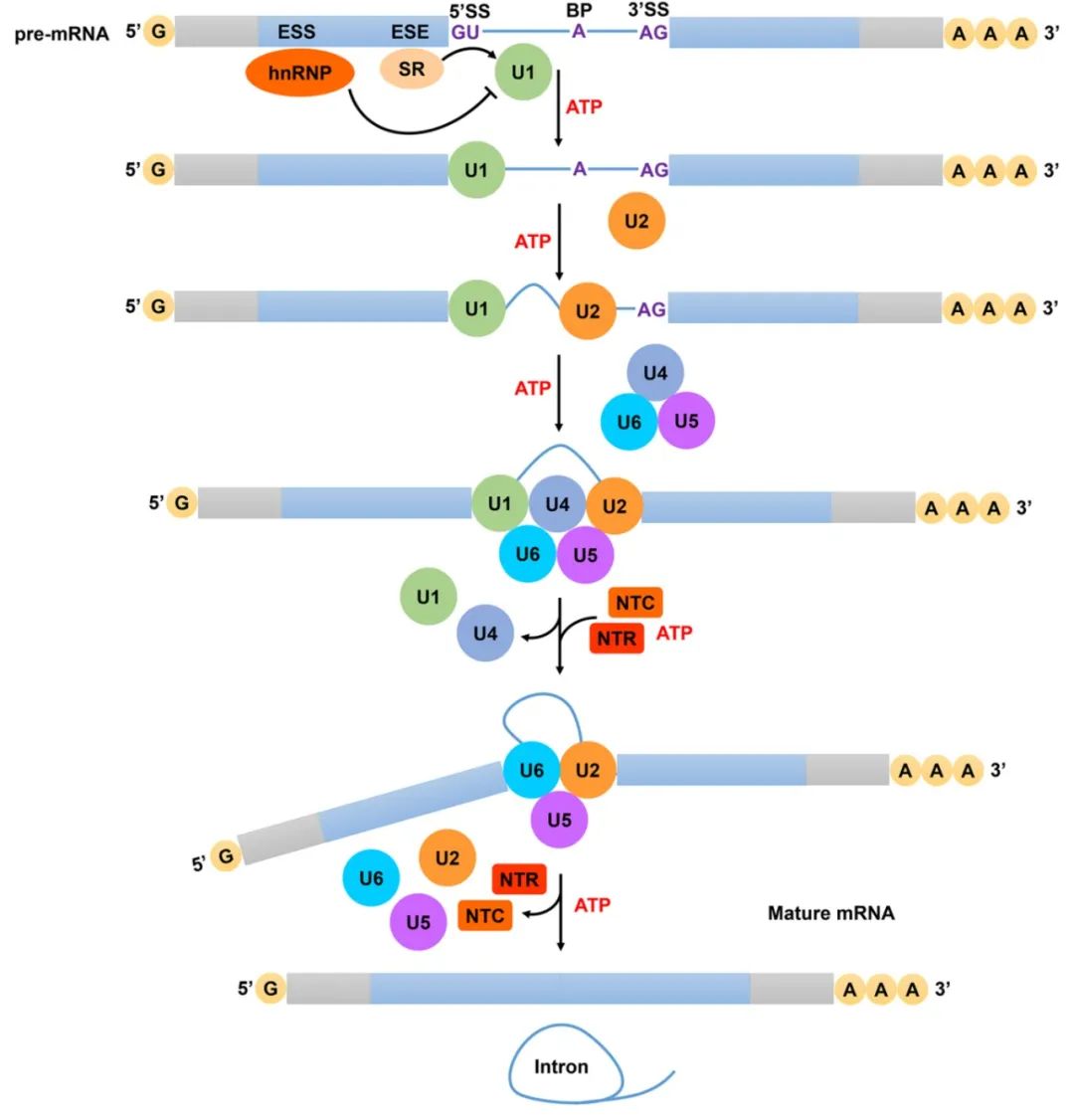
图1 pre-mRNA剪接过程(Lin and Zhu, 2021)。
选择性剪接根据外显子、内含子的去留以及剪接位点的不同可以分为8种常见类型。在植物中以内含子保留的选择性剪接方式居多(Reddy, et al., 2013)。
(1)外显子跳跃类型(Exon skipping, ES):将其中一个外显子以及其两侧的内含子直接剪接掉,使其缺失一个外显子,导致成熟mRNA变短;
(2)内含子保留类型(Intron retention, IR):剪接体选择性地保留一个或多个内含子,导致成熟mRNA中含有内含子序列;
(3)选择性5’剪接类型(Alternative 5′ splicing, A5SS):在5′端外显子序列上具有一个剪接位点,导致该外显子的剪接位点前的序列保留下来,与下一个外显子完成连接;
(4)选择性3’剪接类型(Alternative 3′ splicing, A3SS):在3′端外显子序列上具有一个剪接位点,导致该外显子的剪接位点后的序列保留下来,与上一个外显子完成连接;
(5)选择性5’和3’剪接类型(Alternative 5′ and 3′ splicing):在某一个完整外显子的5’端序列上具有一个剪接位点,在相邻的完整外显子的3’端序列上具有一个剪接位点,导致两个剪接位点中间的外显子和内含子序列均被剪接掉;
(6)外显子互斥类型(Mutually exclusive exon, MXE):两个或者多个外显子之间相互排斥,不能存在同一个转录本中;
(7)选择性起始外显子剪接(Alternative first exon, AFE):基因的两个转录本的区别在于第一个外显子不同;
(8)选择性末端外显子剪接(Alternative last exon, ALE):基因的两个转录本的不同之处于最后一个外显子不同。
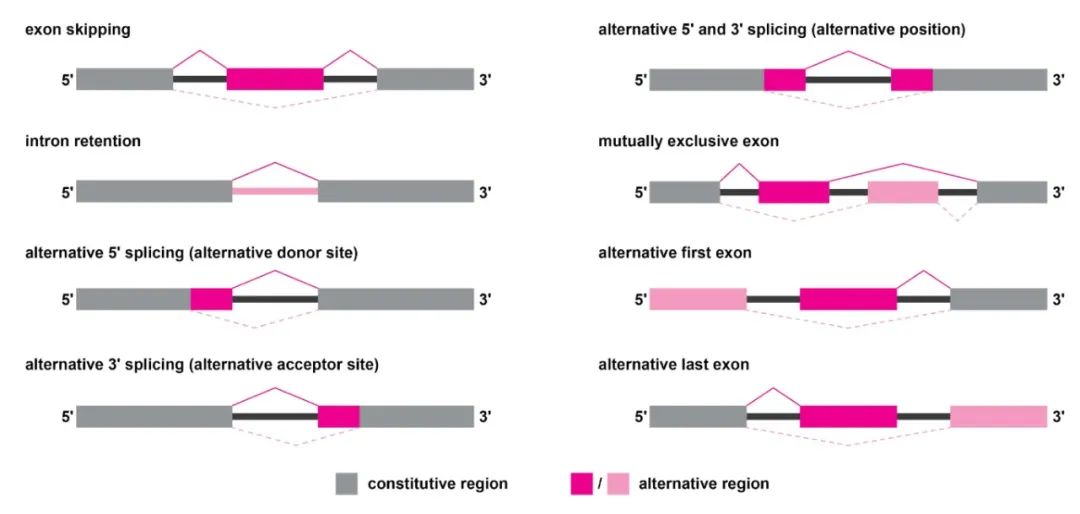
图2 选择性剪接的类型(Lam et al., 2022)。方框代表外显子,线条代表内含子。
通过选择性剪接鉴定,可以深入了解基因转录调控的复杂性和多样性,以及它们在生物体内的功能和调节网络。目前常用的选择性剪接的鉴定方法包括RNA-seq和Iso-seq等。对于以上方法鉴定出的选择性剪接事件,可以使用RT-PCR、ddPCR和QuantAS等实验方法进行验证。
转录组测序技术(RNA sequencing, RNA-seq)是最常用的鉴定选择性剪接事件的方法之一,通过对样本中的mRNA进行高通量测序,然后使用专门的选择性剪接分析软件(例如rMATS等)对测序数据进行选择性剪接分析。
2024年1月,浙江农林大学张进课题组和中国农科院生物技术所王欢课题组在Plant, Cell & Environment杂志上发表了一篇题为“Profiling of the gene expression and alternative splicing landscapes of Eucalyptus grandis”的研究论文。作者以巨桉GL1为材料,对不同组织、发育阶段和胁迫处理的201个样品开展了全面的基因表达谱和选择性剪接分析,研究发现10416个基因发生了选择性剪接,约占桉树总基因的33.49%(图3c),并且发现IR是占比最多的选择性剪接类型,在7538个基因中发现(图3d)。
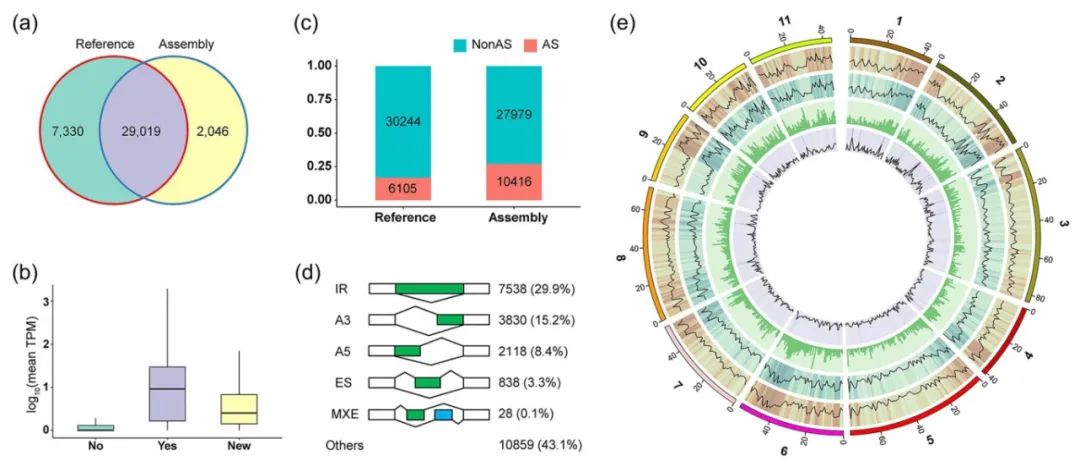
图3 桉树RNA-seq数据特征(Fan et al., 2024)。
(a)通过RNA-seq数据鉴定到的已注释基因和新基因比例;(b)已注释基因和新基因的每百万读取次数(TPM)值的分布情况;(c)发生选择性剪接的基因数量;(d)不同选择性剪接类型统计;(e)选择性剪接和基因组特征在基因组中的分布。
作者进一步通过RT-PCR对巨桉次生生长、组织特异性表达、盐胁迫响应、不定根诱导、磷饥饿反应和硼饥饿反应相关基因的选择性剪接事件进行验证,发现这些关键转录因子和调控基因确实发生了差异选择性剪接。
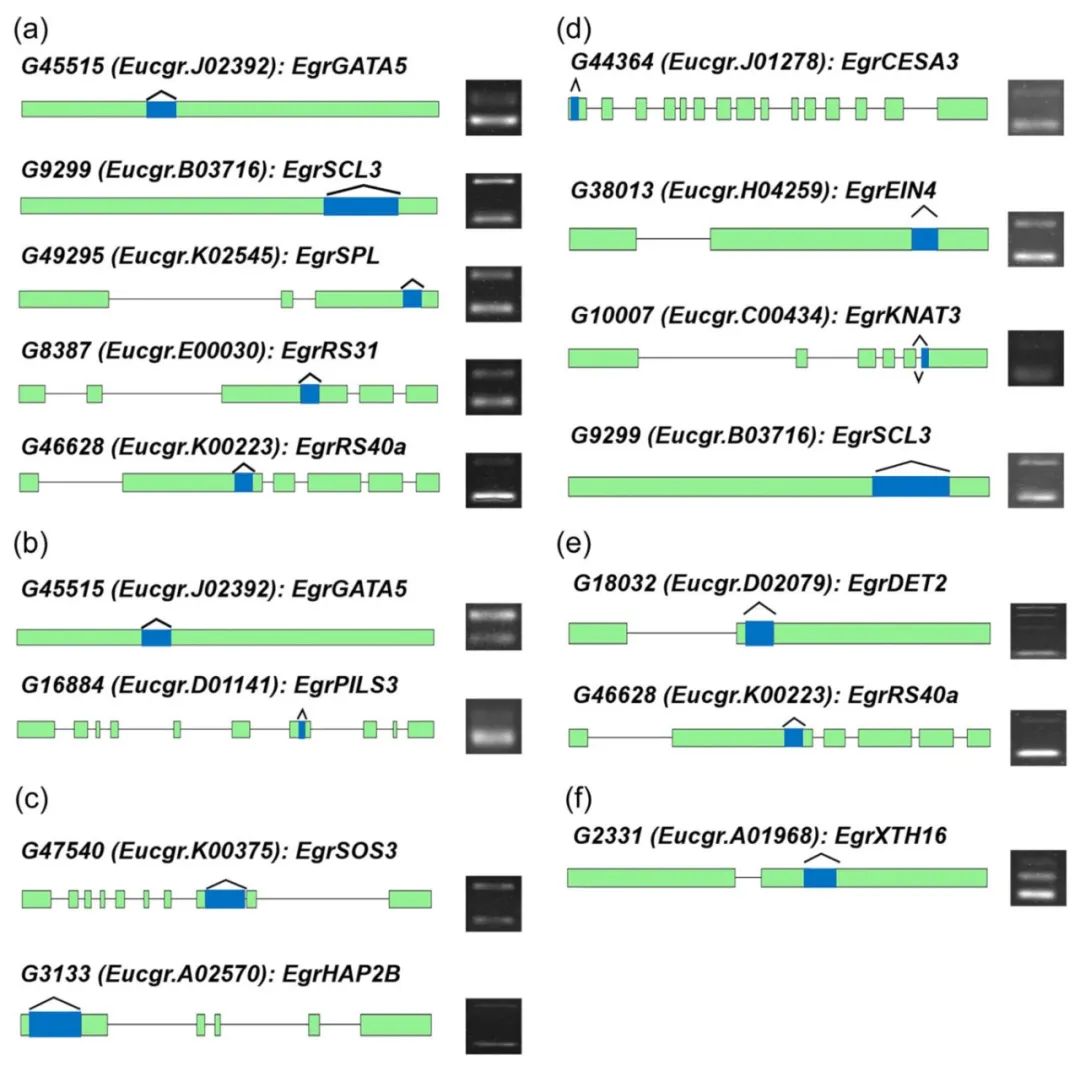
图4 利用RT-PCR验证选择性剪接事件(Fan et al., 2024)。
(a)植物次生生长相关基因的选择性剪接事件;(b)参与不同组织或器官发育相关基因的选择性剪接事件;(c)参与盐胁迫反应相关基因的选择性剪接事件;(d)参与诱导产生不定根相关基因的选择性剪接事件;(e)参与磷饥饿反应相关基因的选择性剪接事件;(f)参与硼饥饿反应相关基因的选择性剪接事件。浅绿色方块表示外显子;蓝色方块表示选择性剪接区域;灰色线条表示内含子。
全长RNA测序(Isoform-sequencing, Iso-seq)是一种通过单分子测序来获得RNA全长信息的技术,这种方法通过直接测定RNA分子的全长,能够检测完整的mRNA或lncRNA序列,包括各种选择性剪接转录本。相对于RNA-seq,Iso-seq具有直接测序全长RNA的优势,特别适用于探索复杂的选择性剪接模式。
2020年1月,中国热带农业科学院彭明课题组在Journal of Experimental Botany杂志上发表了一篇题为“Large-scale analysis of the cassava transcriptome reveals the impact of cold stress on alternative splicing”的研究论文。作者利用Iso-seq技术鉴定木薯在对照和低温处理下的转录本亚型,结果表明在对照和低温处理样本中分别从4708和5373个基因座位中鉴定了总共14868和19275个选择性剪接事件(图5B、C),并且发现IR是木薯在对照和低温条件下最丰富的选择性剪接事件类型(占53-55%),A3SS是第二丰富的选择性剪接事件类型(22-25%)(图5D-F)。
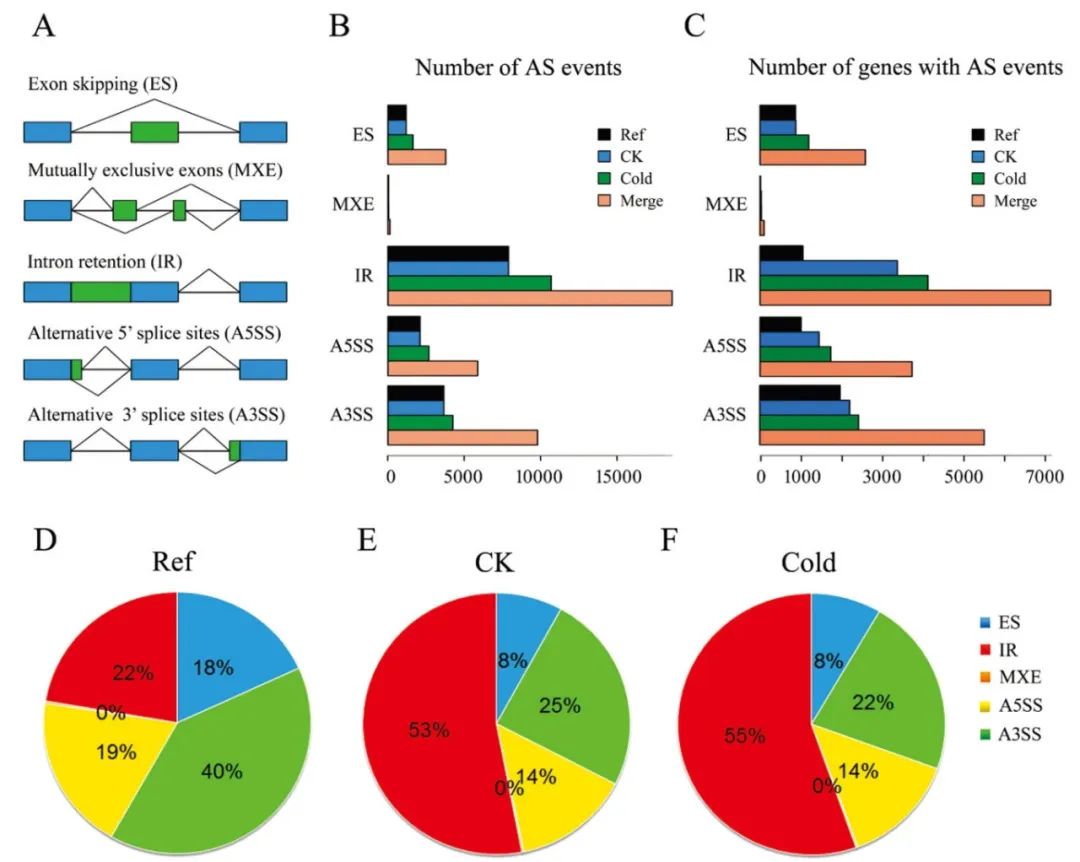
图5 基于参考基因组信息和Iso-seq数据鉴定木薯的选择性剪接(Li et al., 2020)。(A)选择性剪接的类型;(B、C)基于Iso-seq数据,在参考基因组、对照和低温处理样本以及合并样本中鉴定的各种类型选择性剪接事件的数量(B)及其对应的基因数量(C);(D-F)参考基因组(D)、对照(E)和低温处理(F)样本中不同选择性剪接类型出现的频率。
作者进一步通过RT-PCR对七个冷应激相关基因的选择性剪接事件进行验证,发现所验证的七个基因显示出与RNA-seq数据结果一致的选择性剪接模式,这些结果表明,冷胁迫总体上促进了选择性剪接事件。
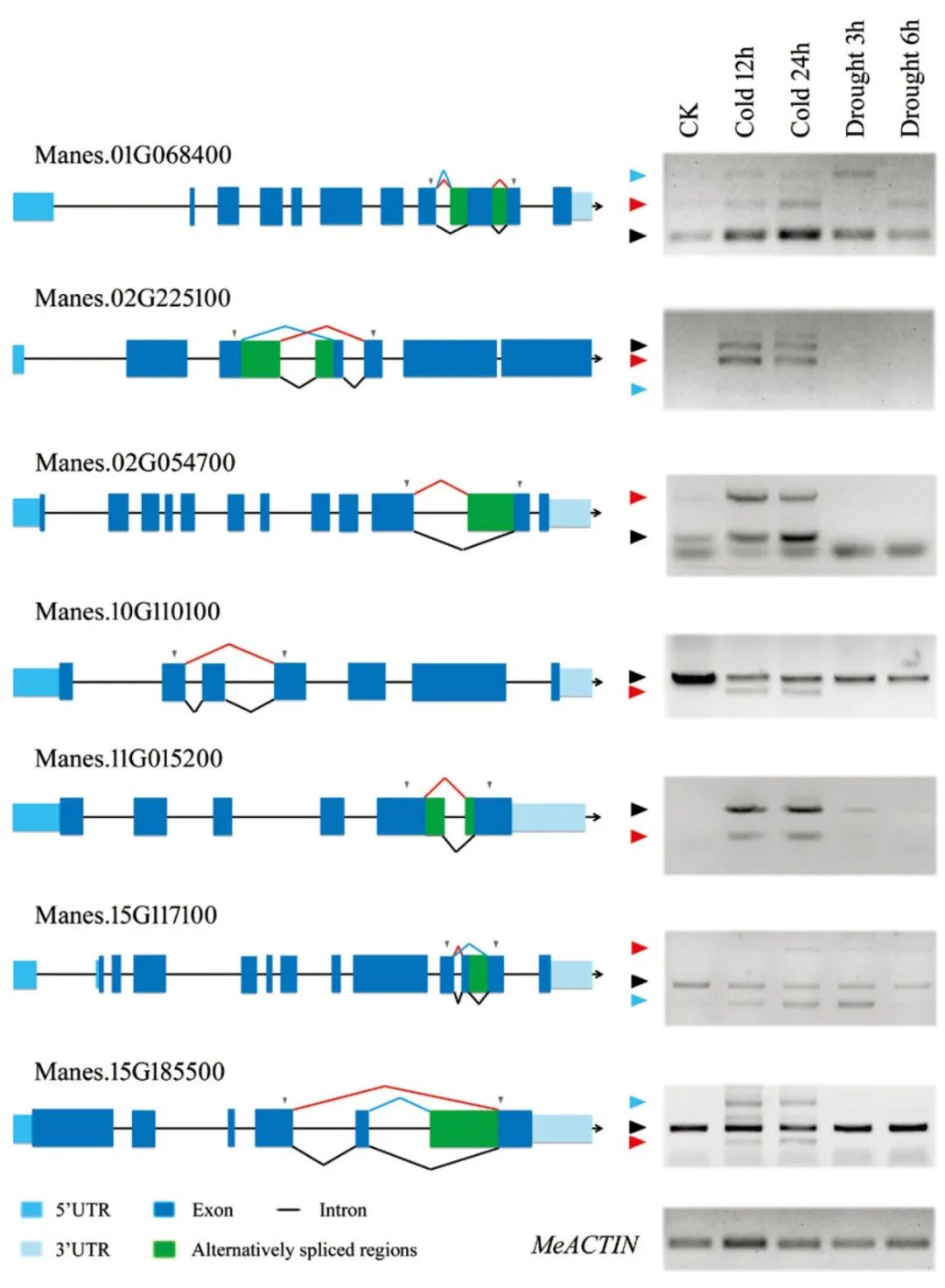
图6 利用RT-PCR检测七个随机选择的基因的选择性剪接事件(Li et al., 2020)。
实时逆转录PCR技术(Reverse Transcription-Polymerase Chain Reaction, RT-PCR)是一种根据选择性剪接产生的剪接转录本设计特异性引物,通过PCR克隆基因,电泳检测产物的大小来初步确定基因是否发生选择性剪接的方法。该方法可以初步鉴定基因有几个剪接转录本,通过测序还可以精确鉴定剪接情况。以上两篇文献案例已经介绍了利用RT-PCR去验证选择性剪接事件,这里就不再举例啦。
微滴式数字PCR(Droplet Digital PCR, ddPCR)可以用于鉴定在特定选择性剪接事件中不同剪接转录本的相对表达水平。与传统的PCR技术相比,ddPCR将样品分成了大量微滴,每个微滴作为一个独立的反应单元,可以提供非常准确的定量结果,尤其对低丰度的剪接转录本更为敏感,这使得ddPCR成为研究选择性剪接事件中不同剪接转录本表达水平的有效方法。
2021年3月,加拿大萨斯喀彻温大学Raju Datla课题组和加拿大国家研究委员会Daoquan Xiang课题组在Plant Biotechnology Journal杂志上发表了一篇题为“Alternative splicing dynamics and evolutionary divergence during mbryogenesis in wheat species”的研究论文。作者分析了小麦胚胎发生和籽粒发育过程中选择性剪接的进化,揭示了基因水平和转录水平调控之间的差异。在小麦中,同源基因的序列相似性很高,因此很难对选择性剪接进行验证。为了解决这一问题,作者开发了一种经济有效的方法,可在一次ddPCR反应中检测三个同源基因的剪接转录本,大大降低了选择性剪接验证的成本。
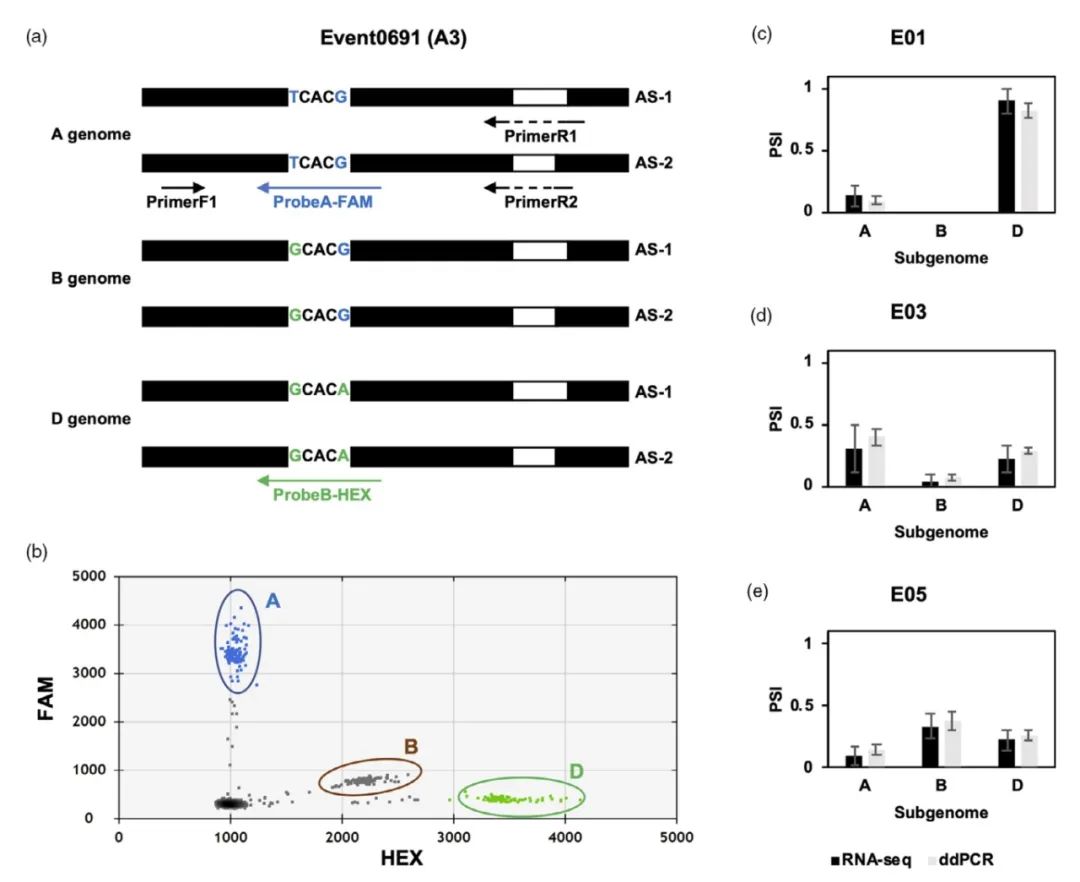
图7 利用ddPCR验证选择性剪接三联体(Gao et al., 2021)。(a)针对A3类型选择性剪接三联体-Event0691的验证策略示例。显示了三个亚基因组中包含两个不同选择性剪接位点(AS-1和AS-2)的PCR产物的结构;(b)AC Barrie在E03阶段AS-1转录本的2-D液滴幅度图;(c-e)RNA-seq分析和ddPCR分析之间的剪接转录本表达水平的一致性比较(PSI)。
2023年11月,南京林业大学曹福亮课题组和朱福远课题组在New Phytologist杂志上发表了一篇题为“QuantAS: a comprehensive pipeline to study alternative splicing by absolute quantification of splice isoforms”的研究论文,基于对各种剪接转录本准确定量的迫切需求,作者开发了一种快速鉴定植物选择性剪接事件的新方法QuantAS,该方法基于绝对定量PCR技术,通过设计特异性引物对不同剪接转录本进行精确定量。QuantAS根据剪接本的蛋白编码区结构,进行剪接本的重新分类,减少了重复鉴定,并且可以在一个反应中检测多个转录本,降低了实验成本,也极大地提高了鉴定的准确性。
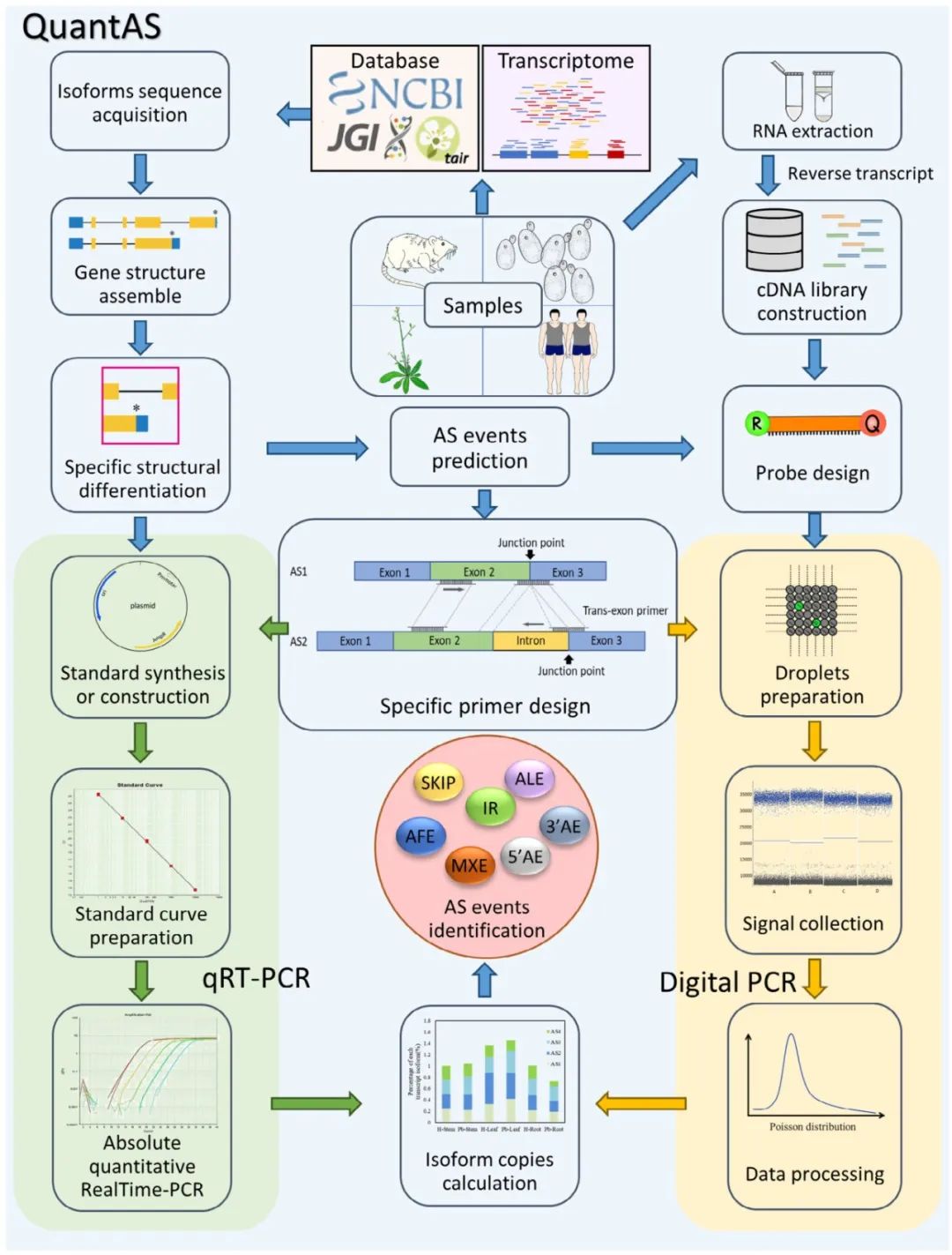
图8 QuantAS的一般实验流程(Song et al., 2023)。首先,通过对数据库和转录组数据的筛选,获得基因各剪接本的序列数据。然后,组装基因结构,寻找不同剪接本之间的结构差异,分析可能的选择性剪接事件,并针对结构差异设计特异性引物。通过qRT-PCR和Digital PCR实验,采用两种方法对每种剪接转录本拷贝数进行绝对定量,以验证选择性剪接事件。
选择性剪接对于生物体是有益的,在不同物种中普遍存在,因为它使得同一个基因可以产生多种功能不同的蛋白质,增加了遗传信息的多样性,提高了生物体的适应性和复杂性。
2022年6月,河北农业大学申书兴课题组在The Plant Journal杂志上发表了一篇题为“Discovery of a DFR gene that controls anthocyanin accumulation in the spiny Solanum group: roles of a natural promoter variant and alternative splicing”的研究论文,作者从乙基甲磺酸(EMS)诱导的突变体库和茄子的自然突变体中发现了一个无花青素的突变体efc1,该突变体的花朵为白色,果实为绿色(图9a、b)。通过进一步鉴定,发现efc1突变体显示了与野生型不同的DFR剪接模式,其中第二个内含子完全保留,导致efc1突变体中的DFR转录本存在一个过早终止密码子(PTC),最终导致较长的转录本无法正常翻译成完整的DFR蛋白(图9e、f)。并且在efc1突变体中过表达DFR转录本恢复了efc1突变体无花青素的表型(图9g)
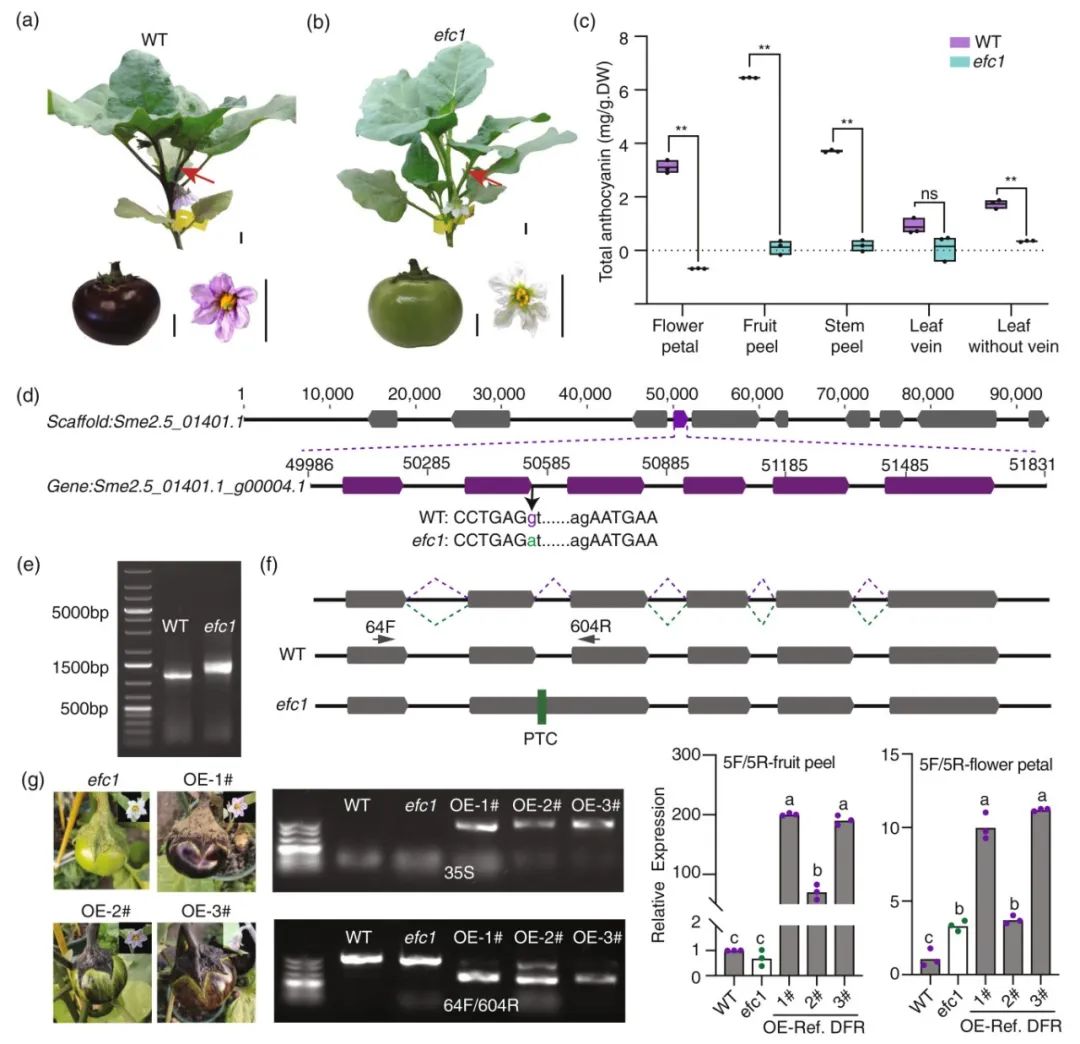
图9 DFR的选择性剪接导致了efc1突变体的无花青素表型(Wang et al., 2022)。(a-b)野生型和efc1突变体的表型;(c)野生型和efc1突变体不同组织中的总花青素含量;(d)候选基因Sme2.5_01401.1_g00004.1在基因组上的位置以及其外显子和内含子结构;(e)RT-PCR扩增野生型和efc1突变体的DFR转录本;(f)efc1突变体中DFR基因发生选择性剪接;(g)过表达DFR转录本恢复了efc1突变体的表型。
作者进一步检测了来自不同种属的17份种质资源的DFR基因第二个内含子的剪接情况,发现茄子种质资源CGN18774显示出与efc1相似的剪接模式,该种质资源也表现出无花青素的表型(图10)。表明DFR基因第2个内含子的剪接与花青素在茄子花瓣和果皮中的积累有很强的相关性,揭示了DFR基因的选择性剪接调控花青素合成的新机制。
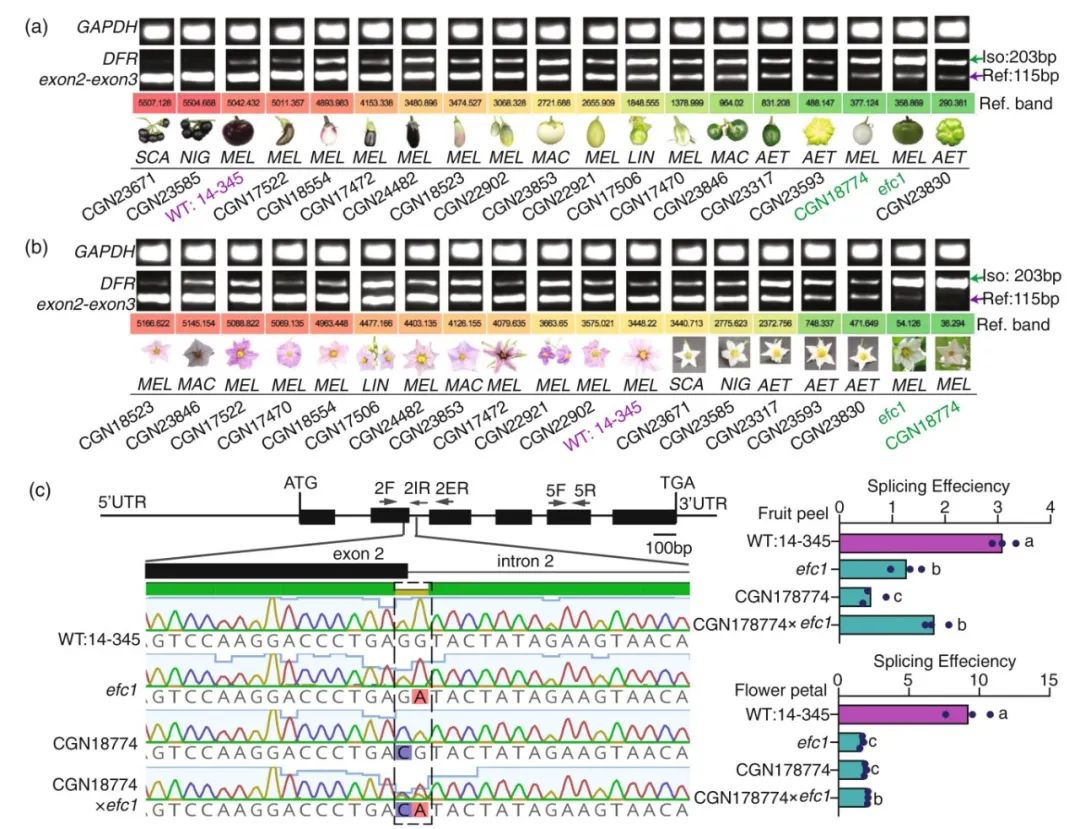
图10 DFR基因第二内含子的选择性剪接与自然群体中花色素积累的关系(Wang et al., 2022)。(a、b)利用RT-PCR对17个茄子种质资源中DFR基因第二内含子的选择性剪接进行验证;(c)来自WT:14-345、efc1、CGN18774、CGN18774×efc1的DFR基因序列比对。
2024年3月,安徽农业大学韦朝领和朱俊彦课题组在Plant Physiology and Biochemistry杂志上发表了一篇题为“Alternative splicing of CsWRKY21 positively regulates cold response in tea plant”的研究论文,作者发现CsWRKY21在5′ UTR区域发生选择性剪接(图11A)。在冷处理下,CsWRKY21-a和CsWRKY21-b的表达模式不同,CsWRKY21-a转录本的表达量没有明显变化,选择性剪接转录本CsWRKY21-B的表达水平在低温下逐渐升高,恢复到正常温度后CsWRKY21-B的表达水平又迅速恢复到对照水平(图11B)。进一步检测CsWRKY21的蛋白表达水平,发现CsWRKY21-b的5′ UTR区域保留内含子后,CsWRKY21-b的蛋白表达水平增加(图12)。综上,CsWRKY21的选择性剪接通过调节其蛋白表达水平进而参与茶树的抗寒性,为揭示选择性剪接介导茶树低温胁迫防御的分子机制提供了理论依据。
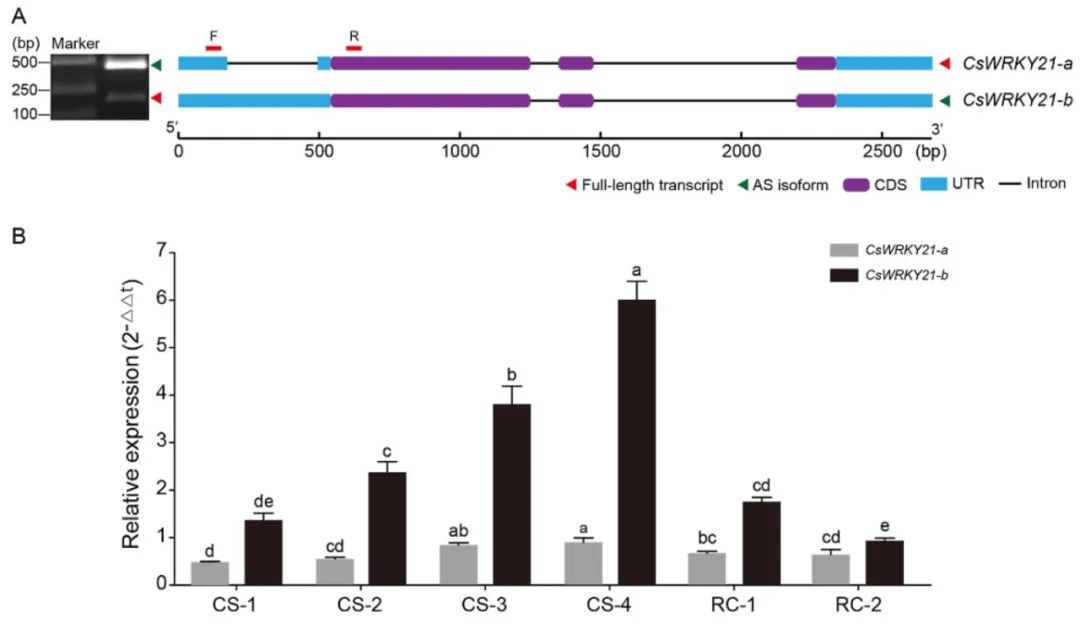
图11 CsWRKY21选择性剪接产物的序列分析和表达分析(Mi et al., 2024)。(A)CsWRKY21选择性剪接的鉴定和序列分析;(B)低温下CsWRKY21-a和CsWRKY21-b不同转录本的qRT-PCR分析。
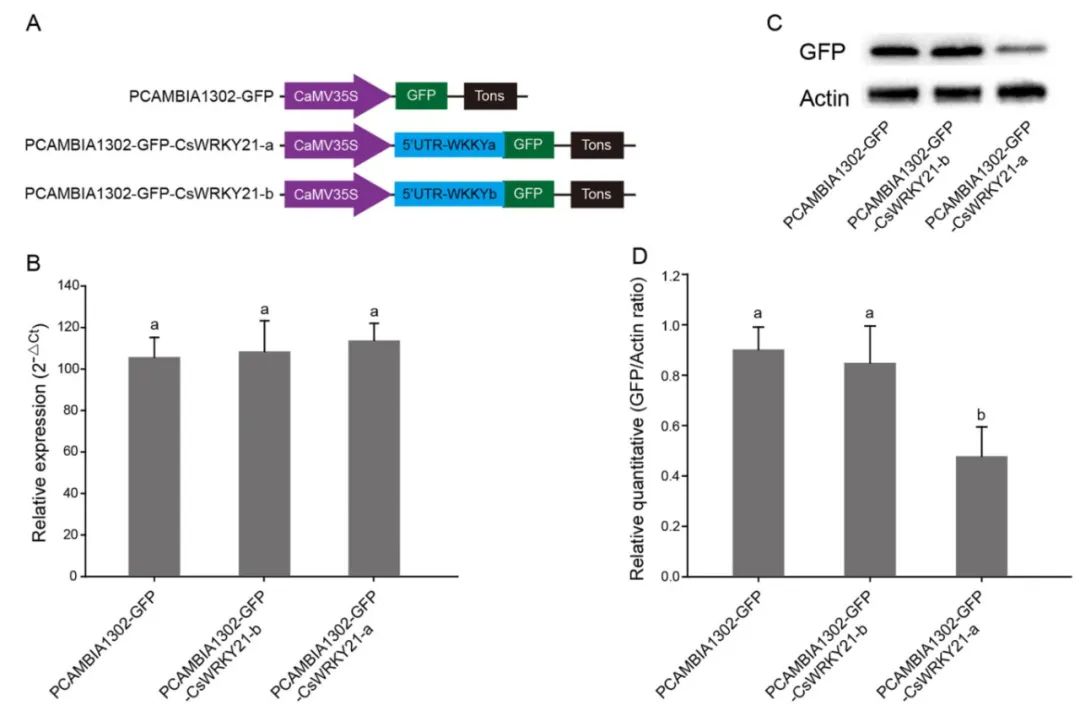
图12 验证选择性剪接对蛋白翻译的影响(Mi et al., 2024)。(A)GFP报告基因载体的构建;(B)烟草中GFP的相对表达水平;(C)GFP蛋白的Western blot分析;(D)GFP蛋白的相对含量。
之前有老师问伯小远,所有基因都会发生选择性剪接吗?对于基因发生选择性剪接后产生的不同转录本,应该选择哪一个转录本进行实验?这里伯小远简要回答一下噢。不是所有基因都会发生选择性剪接,有些基因可能只在特定条件下发生选择性剪接。对于基因发生选择性剪接后产生的不同转录本可能在组织特异性、功能和调控方面存在差异。首先,可以根据已有文献或基因表达谱数据,选择在所研究组织中主要表达的转录本,这将有助于我们更准确地了解基因在特定组织中的功能和调控机制。其次,对各个转录本进行序列分析,比如结构域分析,推测各个转录本可能存在的功能差异。最后,可以考虑同时研究多个转录本,这有助于全面了解基因的功能和调控机制,以及不同转录本之间的相互作用。
在这篇推文中,伯小远首先给大家介绍了关于选择性剪接的一些基础知识,包括剪接的概念、剪接过程以及一些常见的选择性剪接类型。接着介绍了选择性剪接的鉴定方法,包括RNA-seq、Iso-seq、RT-PCR、ddPCR以及QuantAS。最后介绍了选择性剪接在植物中的功能,包括参与植物生长发育、参与植物胁迫响应。希望通过这篇文章,可以给大家的研究提供一些参考。但是选择性剪接的研究远不止如此,转录因子、剪接因子、染色质结构或者组蛋白修饰都有可能会影响选择性剪接的发生,关于这部分的研究有机会再跟大家分享噢。
References:
Lin J, Zhu Z. Plant responses to high temperature: a view from pre-mRNA alternative splicing[J]. Plant Molecular Biology, 2021, 105: 575-583.
Lam P Y, Wang L, Lo C, et al. Alternative splicing and its roles in plant metabolism[J]. International Journal of Molecular Sciences, 2022, 23(13): 7355.
Fan C, Lyu M, Zeng B, et al. Profiling of the gene expression and alternative splicing landscapes of Eucalyptus grandis[J]. Plant, Cell & Environment, 2024.
Li S, Yu X, Cheng Z, et al. Large-scale analysis of the cassava transcriptome reveals the impact of cold stress on alternative splicing[J]. Journal of experimental botany, 2020, 71(1): 422-434.
Gao P, Quilichini T D, Zhai C, et al. Alternative splicing dynamics and evolutionary divergence during embryogenesis in wheat species[J]. Plant biotechnology journal, 2021, 19(8): 1624-1643.
Song Y C, Chen M X, Zhang K L, et al. QuantAS: a comprehensive pipeline to study alternative splicing by absolute quantification of splice isoforms[J]. The New phytologist, 2023, 240(3): 928-939.
Wang X, Chen X, Luo S, et al. Discovery of a DFR gene that controls anthocyanin accumulation in the spiny Solanum group: roles of a natural promoter variant and alternative splicing[J]. The Plant Journal, 2022, 111(4): 1096-1109.
Mi X, Tang M, Zhu J, et al. Alternative splicing of CsWRKY21 positively regulates cold response in tea plant[J]. Plant Physiology and Biochemistry, 2024: 108473.
Reddy A S N, Marquez Y, Kalyna M, et al. Complexity of the alternative splicing landscape in plants[J]. The Plant Cell, 2013, 25(10): 3657-3683.














