单核苷酸多态性基因分型芯片(SNP 芯片)在推进动物和植物群体遗传变异研究中发挥着重要作用。它们可以在一次测定中鉴定和分析多达数十万个 SNP,从而提供高通量且经济有效的方法来分析遗传多样性。因此,它们已被广泛用于生成遗传连锁图、研究进化关系、揭示功能基因组学和支持保护工作。它们还被证明在育种计划中具有很高的价值,可用于基因组选择,标记辅助选择(MAS),全基因组关联研究(GWAS)和 QTL 作图等。
基因分型芯片在六倍体面包小麦的基因分型中发挥着关键作用,使研究人员能够快速筛选小麦品种,识别与重要性状相关的遗传变异,并开发用于育种计划的标记。对于小麦,已经开发了几种 SNP 芯片。这些芯片包含大量的SNP,并已被证明是用于连锁分析、重要性状的QTL作图和全基因组关联分析的有效工具。
然而,之前开发的小麦基因分型芯片由于连锁不平衡(LD)而存在标记分布不均匀和标记冗余的问题。除此之外,较旧的基因分型芯片还受到用于开发它们的群体遗传多样性的限制。面包小麦是一种高度多样化的作物,在不同种群之间和内部具有显着的遗传变异。所以,使用有限的小麦品系开发的基因分型芯片可能无法捕获作物中存在的全部遗传多样性。由于这些限制,科学家和育种者呼吁开发新一代小麦基因分型芯片,以克服这些技术和生物学挑战,提供更全面的基因组视图并捕获面包小麦全基因组中存在的全部遗传多样性。
2024年3月24日,Plant Biotechnology Journal在线发表了一篇题为“Development of a next generation SNP genotyping array for wheat”的研究论文。研究人员描述了一种新的小麦 SNP 基因分型芯片 TaNG 芯片的开发,该芯片旨在克服其中的几个问题,从而提供比以前版本更全面的基因组覆盖范围。
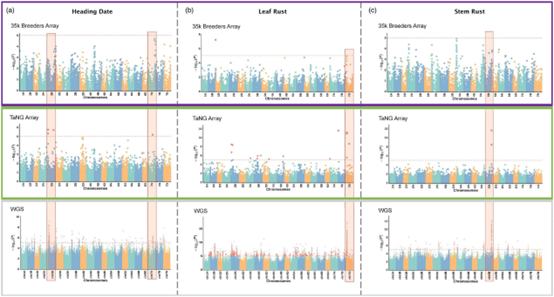
研究者开发了一种新颖的 Axiom 基因分型芯片,即‘Triticum aestivum Next Generation’ array (TaNG), 主要源自对沃特金斯‘Core Collection’中的 204 个优良小麦品系和 111 个小麦地方品种的全基因组测序。研究者使用一种新颖的单倍型优化方法来选择具有最高组合品种辨别力的 SNP,并使用设计迭代步骤来测试和替换未能转换为可靠标记的 SNP。最终设计包含 43372 个 SNP,包含单倍型优化的新型 SNP 和传统跨平台标记的组合。TaNG具有改进的 SNP 分布,并且可用于生成具有比我们以前的阵列明显更多数量的不同箱的遗传图谱。研究者进一步展示了 TaNGv1.1 在全基因组关联研究 (GWAS) 中的性能改进及其在拷贝数变异 (CNV) 分析中的实用性。
综上所述,SNP 基因分型芯片提供了一种高通量且经济高效的方法来分析植物种群的基因组成,从而实现更有针对性和更高效的研究和育种工作。随着技术的不断进步,SNP 基因分型芯片将继续成为植物科学的基石,为可持续农业、作物安全和我们对植物生物学的理解做出贡献。
原文链接:https://doi.org/10.1111/pbi.14341
文章来源:植物生物技术Pbj