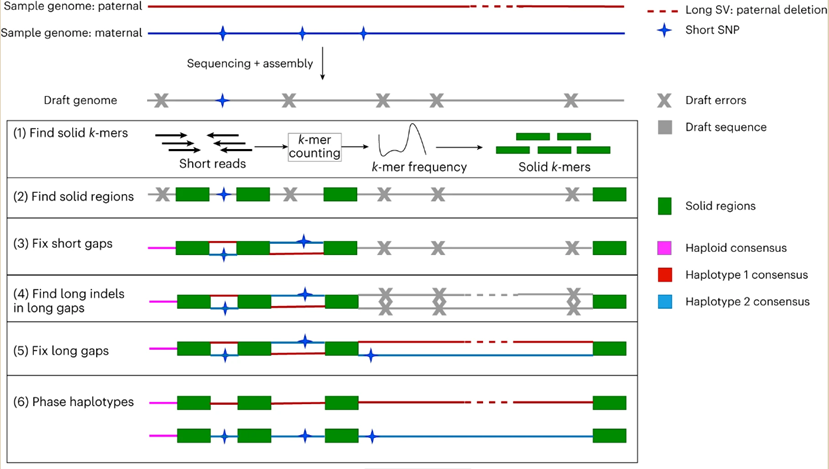
Hypo-short 主要利用 Illumina reads来降低基于 ONT 的草图组装,Hypo-hybrid使用短reads和长reads来减少组装结果中的错误,并获得高质量的二倍体基因组。基于hyper-hybrid,作者进一步开发了hyper-assembler的二倍体基因组组装流程。Hypo-assembler 使用 ONT 长reads、Illumina 短reads和可选的 Hi-C reads来自动的进行高度准确、连续且几乎完整的二倍体基因组的组装,并最终获得端粒到端粒完整二倍体基因组。
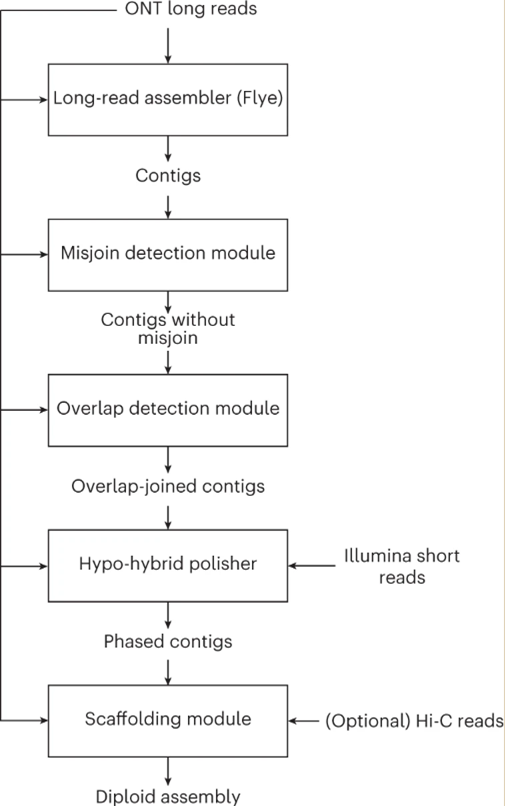
图2 hyper-assembler 组装的流程
研究结果为其他植物端粒到端粒的组装、二倍体基因组的组装提供了新的思路和工具。在弥补错误率问题的同时还能组装高质量的基因组,为今后研究植物基因组组装领域的发展奠定基础或提供技术支持。
原文链接:
https://www.nature.com/articles/s41592-023-02141-1
文章来源:植物生物技术Pbj