本文内容速览:
首先我们回忆一下什么是转录因子(Transcription factor, TF)和转录因子结合位点(Transcription factor binding site, TFBS)。转录因子是指能够与顺式作用元件发生特异性相互作用,并对基因的转录具有激活或抑制作用的DNA结合蛋白。在植物生长发育和逆境应答等关键过程中,转录因子起着非常重要的调控作用。转录因子结合位点是转录因子调节基因表达时,与基因的模板链发生结合的特定区域,即与转录因子结合的DNA片段,长度通常在5~20bp之间。一个转录因子通常可以同时调控多个基因,其在不同基因上的结合位点具有一定的保守性,但又并非完全相同。
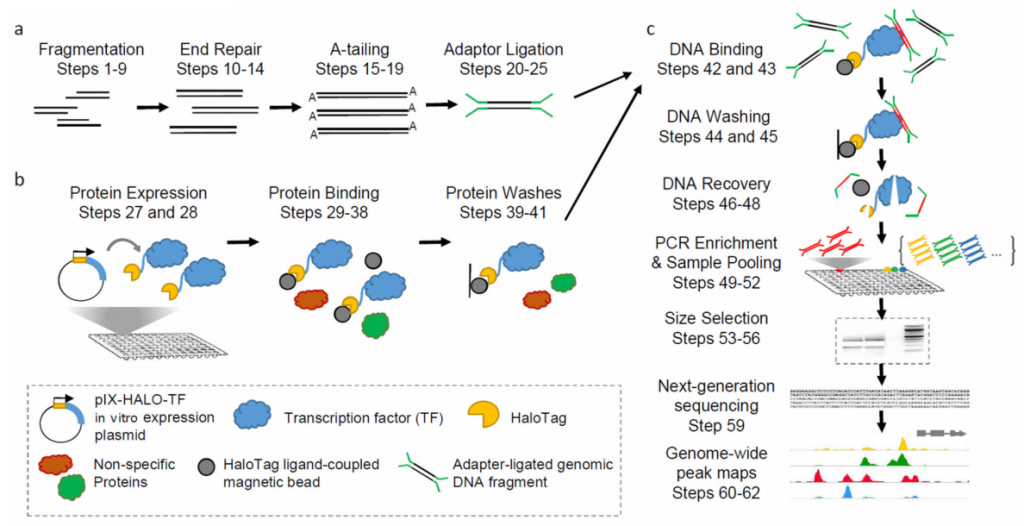
(2)体外表达蛋白:将编码目的转录因子的CDS序列构建到带有亲和标签(Halo Tag)的载体中,构建蛋白表达载体,进行体外蛋白表达,形成转录因子和亲和标签的融合蛋白;
(3)亲和纯化及测序:将纯化后的转录因子和亲和标签融合蛋白与基因组DNA文库共同孵育,融合蛋白能与基因组DNA片段上的特定序列结合。通过使用Halo Tag特异性磁珠,将目的蛋白和DNA复合物进行提取和纯化,然后对捕获的DNA片段进行PCR扩增,并利用Illumina测序平台进行测序;
(4)数据分析:将测序产生的序列读数与相应的参考基因组进行比对,从而确定感兴趣转录因子的潜在结合位点。
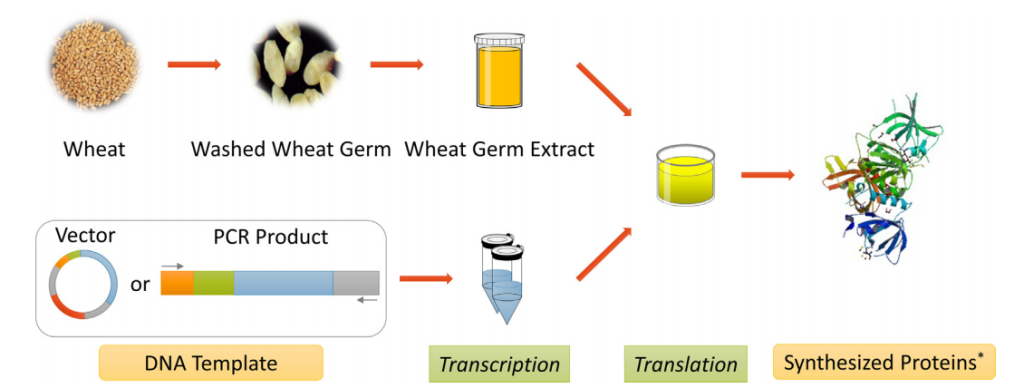
小麦胚芽(麦胚)被认为是小麦籽粒中生命活动最旺盛的部分,它含有蛋白质合成所必需的各种要素,包括蛋白质合成场所、蛋白因子以及相关酶系等。正因如此,科学家们已经建立了以小麦胚芽为原料的无细胞蛋白表达系统。与大肠杆菌无细胞蛋白表达系统相比较,WGCF更适宜用于表达真核基因,能够高效地在体外合成复杂的真核蛋白并进行正确地折叠(Madono et al., 2011)。
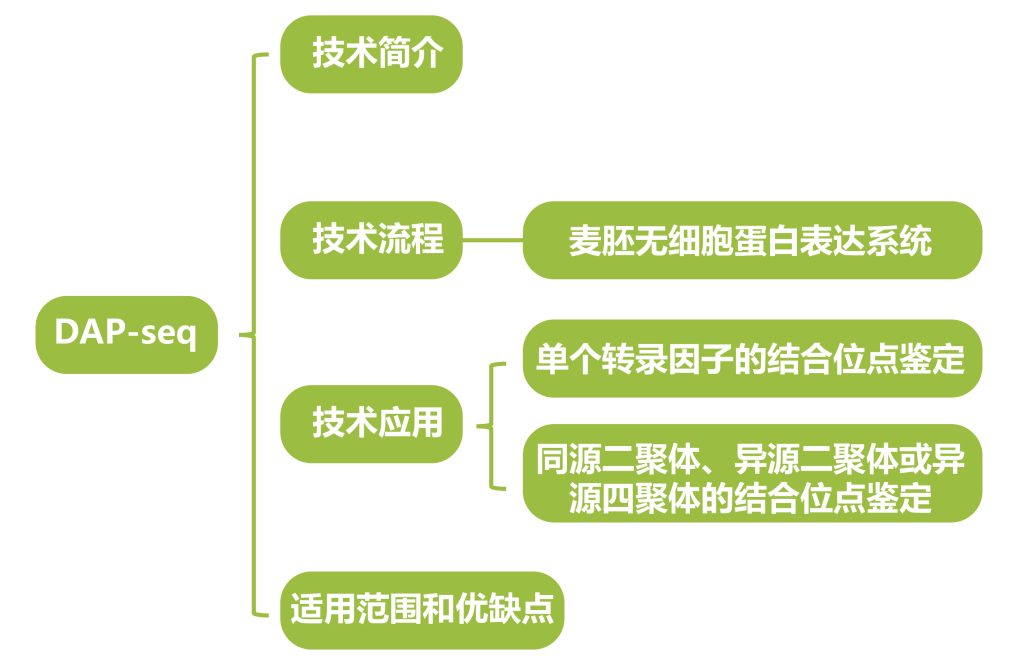
DAP-seq技术应用
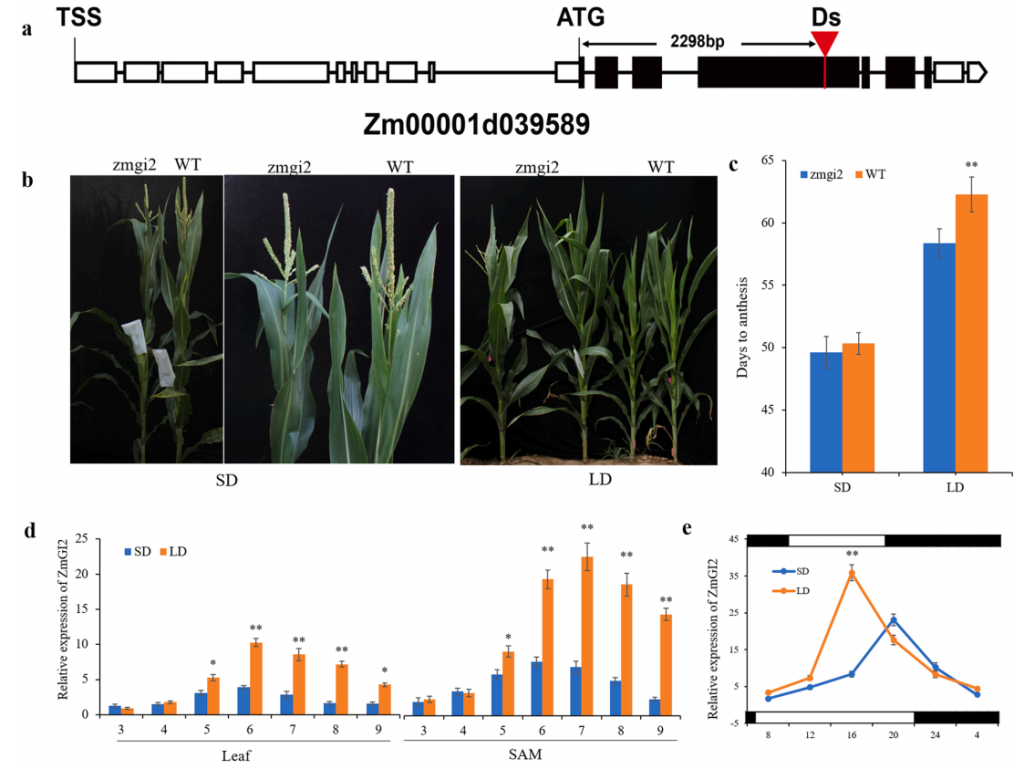
GIGANTEA(GI)编码一种新的核蛋白,是控制植物生理节律和开花的关键调控因子,在植物长日照途径中GI发挥着重要的调控作用,然而关于玉米中GI对开花时间的调控途径目前还不清楚。
图3 ZmGI2调控玉米开花时间(Li et al., 2023)。(a)zmgi2突变体中Ds转座子插入位置的示意图;(b)在LD和SD条件下zmgi2突变体和WT植株的表型;(c)在LD和SD条件下zmgi2突变体和WT植株开花的天数;(d)ZmGI2在第3至第9片完全展开的叶片和茎尖分生组织(SAM)中的表达模式;(e)ZmGI2在SAM中的表达显示出昼夜节律。
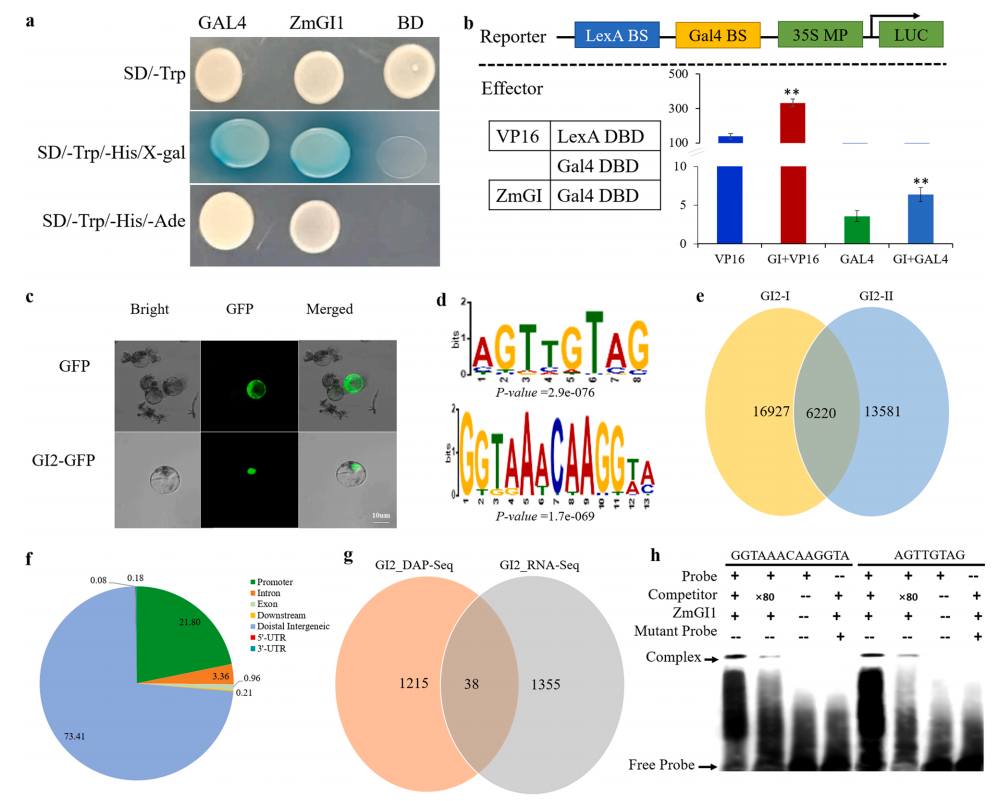
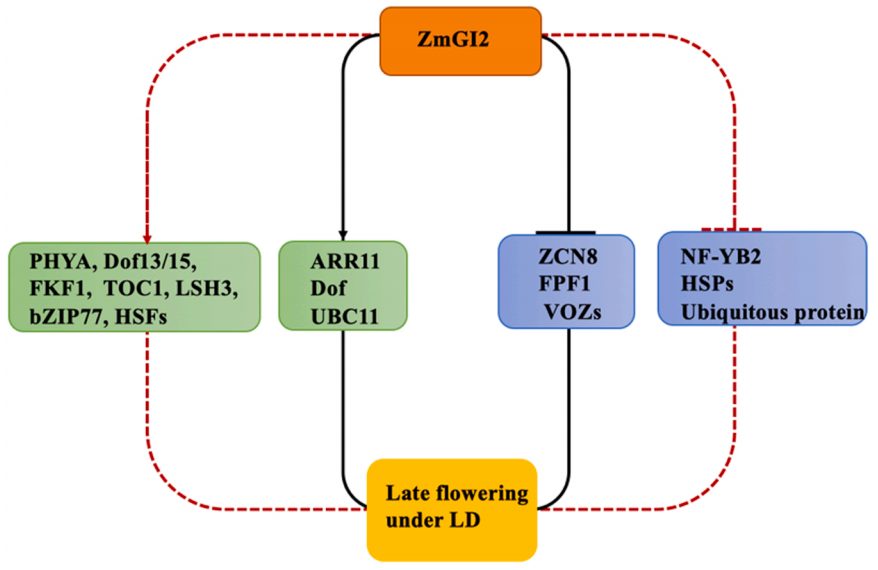
接着作者利用RNA-seq和DAP-seq技术探究ZmGI2的靶基因,发现ZmGI2可以与AGTTGTAG和GGTAAACAAGGT这两个基序(Motif)特异性结合(图4),进一步利用凝胶迁移实验(EMSA)和双荧光素酶报告基因实验(Dual-LUC)证明ZmGI2通过直接结合ZmVOZs、ZmZCN8和ZmFPF1的上游区域来抑制这些基因的表达,并直接结合ZmARR11、ZmDOF和ZmUBC11的上游区域来促进这些基因的表达,从而延缓开花(图5)。
图4 DAP-seq技术分析ZmGI2的结合位点(Li et al., 2023)。(a)ZmGl2在酵母中的转录活性分析;(b)使用Gal4/UAS系统对ZmGI2的转录活性进行瞬时测定;(c)在玉米原生质体中瞬时表达ZmGI2-GFP融合蛋白的核定位;(d)ZmGI2的两个结合基序;(e)两个生物学重复的结合峰数量;(f)ZmGI2结合位点在玉米基因组中的分布;(g)DAP-seq和RNA-seq结果之间的重叠基因;(h)EMSA结果证实ZmGI2与GGTAAACAAGGTA和AGTTGTAG体外结合。
综上所述,作者绘制了ZmGI2调控开花时间的工作模型,为全面解析GI调控玉米花期的分子机制和调控网络提供了帮助。
通过上面这篇文献案例,我们知道可以利用DAP-seq技术来研究单个转录因子的结合位点,但是许多真核生物转录因子是通过形成同源二聚体、异源二聚体或异源四聚体来共同调控基因表达的,是否有一种技术可以用来研究同源二聚体、异源二聚体或异源四聚体的DNA结合特异性呢?答案是肯定的,让我们一起往下看吧。
seq-DAP-seq
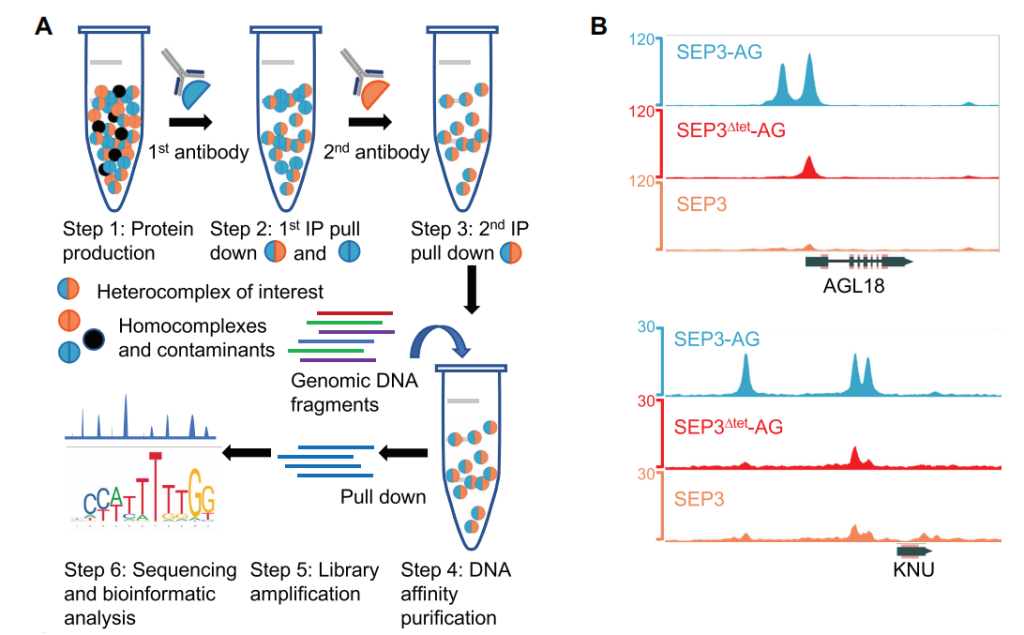
2020年9月,格勒诺布尔-阿尔卑斯大学Chloe Zubieta课题组在Nucleic Acids Research杂志上发表了一篇题为“Genome-wide binding of SEPALLATA3 and AGAMOUS complexes determined by sequential DNA-affinity purification sequencing”的研究论文,作者开发了一种改进的DAP-seq方法(seq-DAP-seq)(图6A),并利用该方法研究了SEP3同源二聚体、SEP3-AG异源二聚体和SEP3Δtet-AG异源四聚体在全基因组范围内的结合情况(图6B)。
图6 seq-DAP-seq实验流程与数据分析(Lai et al., 2020)。(A)seq-DAP-seq实验流程。每个TF或TF组合都用标签表达,并用特异性抗体依次拉下并纯化。纯化的TF复合物用于DNA亲和纯化测序;(B)利用seq-DAP-seq方法测定的SEP3-AG、SEP3Δtet-AG、SEP3在全基因组范围内的结合峰。
此外,一些MADS转录因子还可以调控温度介导的芽休眠,这些TFs被称为DORMANCY-ASSOCIATED MADS-BOX(DAM),类似于拟南芥的SHORT VEGETATIVE PHASE(SVP)。MADS转录因子以二聚体、四聚体等复合体形态发生作用,然而,MADS转录因子形成的多聚体复合物如何控制树木的休眠周期尚不清楚。
2021年12月,蒙彼利埃大学Fernando Andrés课题组在New Phytologist杂志上发表了一篇题为“Unraveling the role of MADS transcription factor complexes in apple tree dormancy”的研究论文,作者发现DAM-、FLC-和SVP-like复合体通过响应环境信号和激素信号通路来调节苹果的休眠周期。
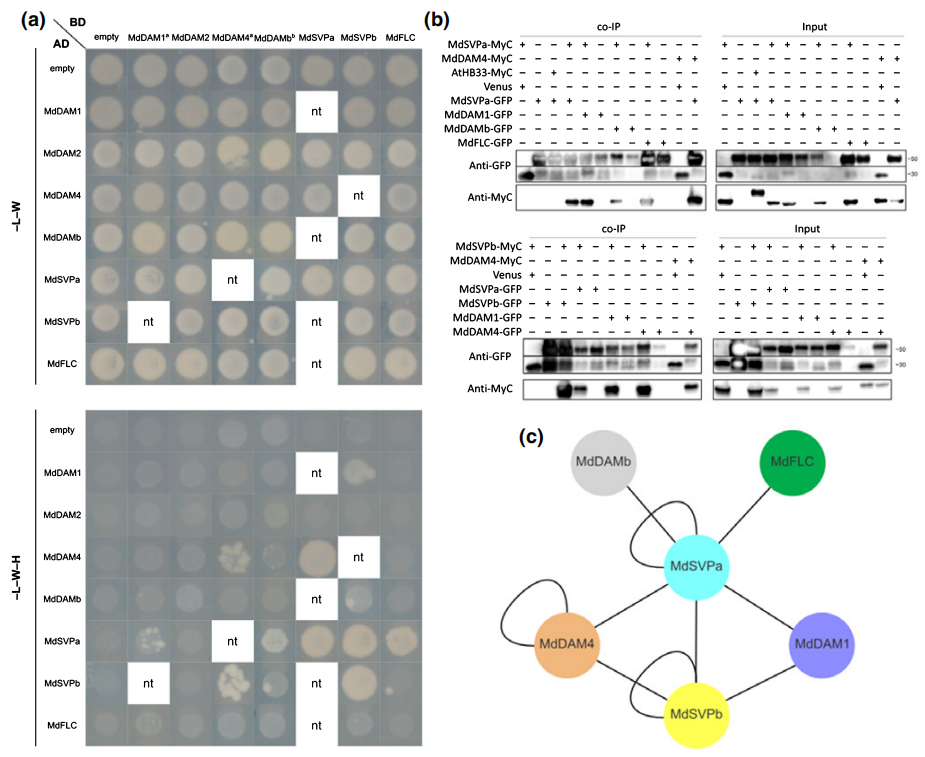
作者通过酵母双杂交实验(Y2H)和免疫共沉淀实验(Co-IP)证明苹果DAM-、FLC-和SVP-like蛋白之间存在一个以MdSVPa为中心的相互作用网络(图7)。
图7 苹果DAM-、FLC-和SVP-like蛋白之间的相互作用网络(da et al., 2021)。(a)Y2H实验验证MADS蛋白之间的相互作用;(b)Co-IP实验验证Y2H的结果;(c)本研究中发现的MADS蛋白之间的相互作用网络。
进一步通过EMSA实验发现只有含有MdSVPa的复合体才表现出DNA结合,随后利用seq-DAP-seq技术来研究这些复合体在苹果中的全基因组结合位点从而阐明它们的功能,结果表明四种MADS复合体(MdDAM1-MdSVPa、MdDAM4-MdSVPa、MdFLC-MdSVPa、MdSVPa-MdSVPa)共同结合了219个位点,其中包括重要的开花进程调控因子,如MdSPL9和MdCDF2(图8)。
dDAP-seq
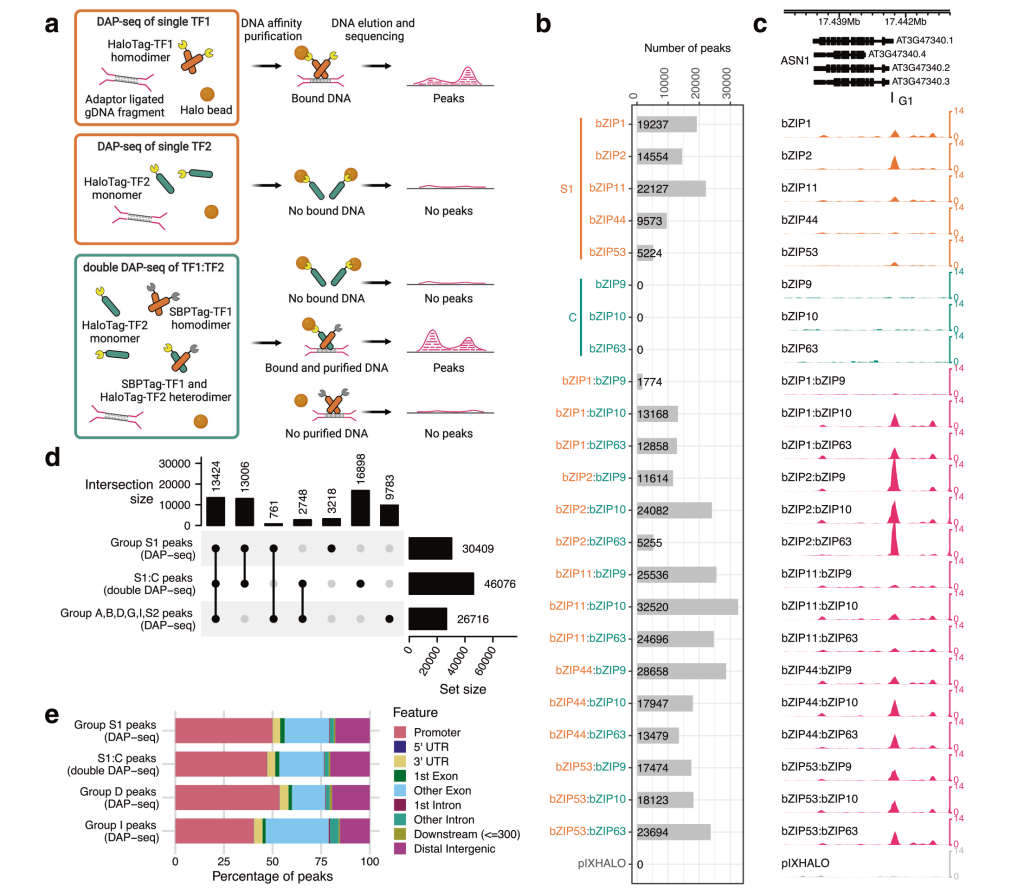
2023年5月,美国纽约大学Shao-shan Carol Huang课题组在Nature Communications杂志上发表了一篇题为“Double DAP-seq uncovered synergistic DNA binding of interacting bZIP transcription factors”的研究论文,作者开发了一种新方法Double DAP-seq(dDAP-seq),该方法可以用来研究同源二聚体或异源二聚体在全基因组范围内的结合位点。作者利用dDAP-seq鉴定了拟南芥中20对C/S1 bZIP异源二聚体和S1同源二聚体的全基因组结合位点(图9),结果表明C/S1 bZIP异源二聚体显著扩展了bZIP转录因子的DNA结合偏好,进一步对C/S1 bZIP异源二聚体结合位点的分析揭示了bZIP9在ABA响应中的功能以及bZIP53异源二聚体特异性结合在种子成熟中的作用。
转录因子结合位点鉴定方法
的适用范围和优缺点
(1)ChIP-seq技术不仅可以用于研究转录因子结合位点,还可用于研究组蛋白的修饰情况、核小体的定位图谱等。在植物中,由于转录因子特异性抗体制备较为缺乏,并且ChIP-seq实验需要107个细胞的样本,因此,在进行ChIP-seq实验之前,需要获得转录因子融合标签蛋白的稳定遗传转化植株,再利用标签抗体进行免疫沉淀,从而解决较难获得植物蛋白特异性抗体和ChIP-seq所需样本量多的问题。但这样就导致ChIP-seq实验周期较长,并且ChIP-seq并不适用于一些没有稳定遗传转化体系的物种。满足样本量需求的原生质体转化ChIP-seq技术已被成功应用到模式植物中,能够高效鉴定体内DNA的结合位点,但该技术需要建立高效的原生质体制备技术和交联方法。
(2)CUT&Tag技术也可以用于研究转录因子结合位点以及组蛋白的修饰情况。与ChIP-seq相比,CUT&Tag处理流程更快、分辨率更高、背景信号更低、所需的样本量更少。但是,CUT&Tag技术也需要获得稳定的遗传转化植株,因此不适用于一些没有稳定遗传转化体系的物种。除此之外,基于原生质体体系的CUT&Tag技术(pCUT&Tag)不需要转基因,可以节约样品准备周期和建库成本,且与ChIP-seq数据相比具有更高的数据质量和信号分辨率以及更低的测序深度,但是原生质体制备是该技术的一个难点。
(3)DAP-seq技术主要用于鉴定转录因子结合位点,包括单个转录因子结合位点的鉴定以及同源二聚体、异源二聚体或异源四聚体的结合位点鉴定。DAP-seq技术采用体外表达标签蛋白,将表达产物与目标基因组序列进行结合,不需要特异性抗体,不需要转基因,可用于没有稳定遗传转化体系的物种,并且DAP-seq技术实验时间更短。相比于pCUT&Tag,没有制备植物原生质体的技术限制,对构建转录因子调控网络方面具有很好的应用前景。但是DAP-seq是体外实验,因此无法真实反映体内转录因子与DNA的互作。
伯小远将ChIP-seq、CUT&Tag、DAP-seq三种方法的优缺点和适用范围总结如下(表1)。
表1 鉴定转录因子结合位点的表观组学技术比较。
其实目前还有一些用于鉴定转录因子结合位点的方法,比如CUT&RUN、DAMID-seq、RDSA、DiMeLo-seq以及PER-seq,并且这些方法在植物转录因子结合位点的研究中也有广泛应用,如果大家感兴趣的话,伯小远将会在后面的文章中详细介绍这几种方法噢。
Katzen F, Chang G, Kudlicki W. The past, present and future of cell-free protein synthesis[J]. Trends in Biotechnology. 2005;23(3):150-156.
Madono M, Sawasaki T, Morishita R, et al. Wheat germ cell-free protein production system for post-genomic research[J]. New Biotechnology. 2011, 28(3): 211-217.
Harbers M. Wheat germ systems for cell-free protein expression[J]. FEBS Letters. 2014, 588(17): 2762-2773.
da Silveira Falavigna V, Severing E, Lai X, et al. Unraveling the role of MADS transcription factor complexes in apple tree dormancy. New Phytologist. 2021, 232(5): 2071-2088.
Bartlett A, O’Malley R C, Huang S C, et al. Mapping genome-wide transcription-factor binding sites using DAP-seq[J]. Nature Protocols, 2017, 12(8): 1659-1672.
O’Malley R C, Huang S C, Song L, et al. Cistrome and epicistrome features shape the regulatory DNA landscape[J]. Cell, 2016, 165(5): 1280-1292.
Li Z, Gao F, Liu Y, et al. ZmGI2 regulates flowering time through multiple flower development pathways in maize. Plant Science. 2023, 332: 111701.
Lai X, Stigliani A, Lucas J, et al. Genome-wide binding of SEPALLATA3 and AGAMOUS complexes determined by sequential DNA-affinity purification sequencing[J]. Nucleic Acids Research. 2020, 48: 9637-9648.
Li M, Yao T, Lin W, et al. Double DAP-seq uncovered synergistic DNA binding of interacting bZIP transcription factors[J]. Nature Communications, 2023, 14(1): 2600.
Wang G, Li X, An Y, et al. Transient ChIP-Seq for genome-wide in vivo DNA binding landscape[J]. Trends in Plant Science, 2021, 26(5): 524-525.