本文内容速览:
小远在前面的推文中给大家介绍过“蛋白翻译后修饰——泛素化”,文中提到了泛素-蛋白酶体系统,知道了该系统可以降解蛋白质,那么生物体内除了该系统可以降解蛋白质之外,是否还存在其它的蛋白质降解机制呢?答案当然是有的,这里就引出了小远今天将要给大家介绍的主题——自噬。
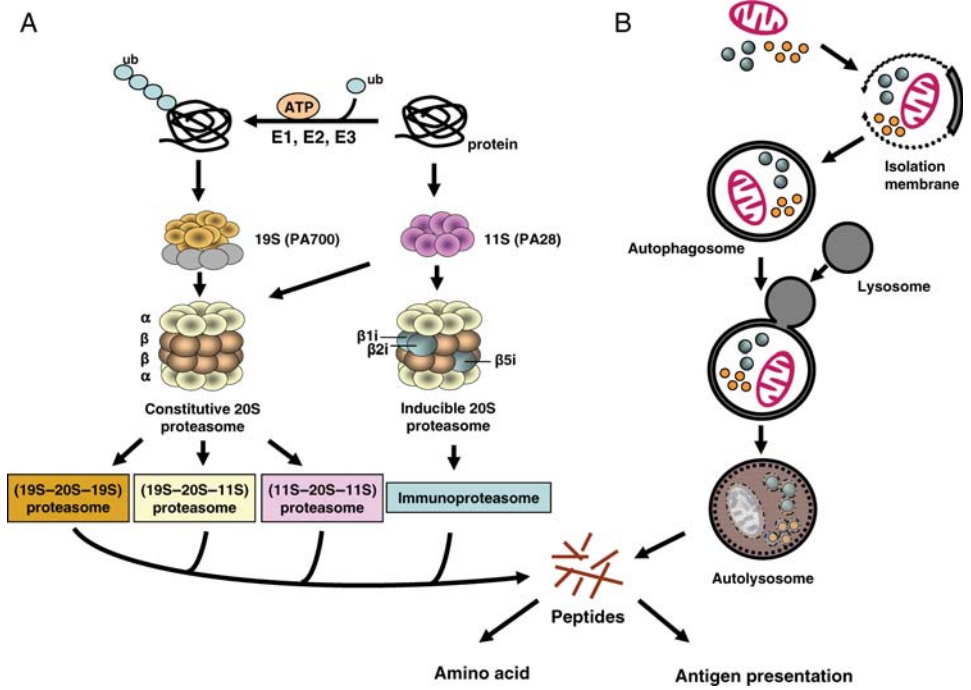
自噬(Autophagy)和泛素-蛋白酶体系统(Ubiquitin-proteasome system, UPS),是细胞内蛋白降解的两个主要途径(图1)。自噬,字面意思就是“自己吃自己”,它在降解细胞内细胞器和较稳定蛋白上起着重要的功能,而UPS则负责特异性地降解大多数细胞内蛋白,是一种蛋白高效降解途径,具有广泛的生物学作用。另外,自噬参与了许多重要的生理和病理过程,成为近年来分子生物学发展最快的领域之一。
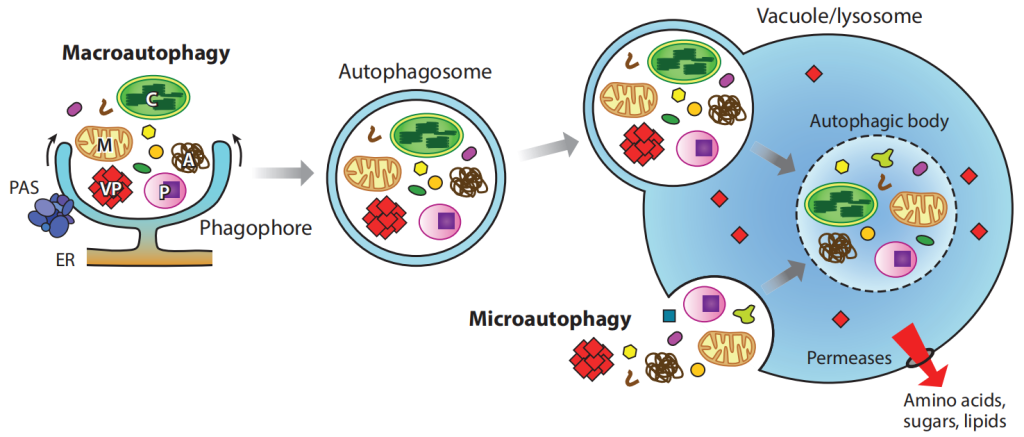
在微自噬过程中,细胞质蛋白或整个细胞器聚集在液泡附近,并被液泡膜的内陷或突出包裹起来,从而形成一个被称为自噬体的泡内囊泡,通过膜分裂释放蛋白或细胞器到空泡腔内进行降解(Van Doorn and Papini, 2013;Marshall Richard S. and Vierstra Richard D., 2018)。相比之下,在巨自噬中,自噬体组装位点(PAS)上形成了一个环状的双膜结构,称为吞噬细胞。一些研究表明,吞噬细胞起源于内质网、线粒体、质膜或内质网和线粒体所接触的部位(Hamasaki et al., 2013;Le et al., 2014;Zhuang et al., 2017)。吞噬细胞拉长并包围细胞质物质,形成双膜结构的自噬体,随后与液泡融合并且降解其所包裹的内容物(Yimo Liu and Diane C. Bassham, 2012)。
自噬的第三种途径——超级自噬,目前只在植物中有发现,并与程序性细胞死亡(PCD)同时发生。与微自噬和巨自噬不同,超级自噬将大分子成分从液泡中回收到细胞质中,是一种大规模降解、导致细胞死亡的极端形式。
除了保守的微自噬和巨自噬途径外,酵母中还有一个细胞质-液泡靶向(Cvt)途径,该途径是生物合成的,并组成型地将驻留水解酶的前体传递到液泡。Cvt途径的运作机制与巨自噬的机制相似(Daniel J. Klionsky and Scott D. Emr, 2000)。在哺乳动物中,分子伴侣介导的自噬是第三种途径,该途径不使用单独的囊泡,直接将底物蛋白转移到溶酶体中(J. Fred Dice, 2007)。目前,没有证据表明植物中存在Cvt途径或分子伴侣介导的自噬。
植物的微自噬和巨自噬都有功能(Bassham et al., 2006)。这两个途径的机制与在其他模式生物中描述是相似的。
在植物微自噬中,目标物质直接被细胞膜内陷所吞噬。含有目标物质的囊泡被挤压,释放到液泡内并降解。在植物发育过程中,微自噬参与了储存蛋白、脂质和积累淀粉颗粒的降解过程(Van et al., 1980;Poxleitner et al., 2006)。
巨自噬,通常简称为自噬,自噬过程可分为不同的阶段:诱导、识别降解物质、吞噬细胞形成、吞噬细胞扩张和关闭以及自噬体融合和分解(Masclaux-Daubresse et al., 2017)。自噬的具体的过程见下图(图3)。
关于自噬的具体机制,这里不详细展开,主要介绍与自噬相关的基因与蛋白,具体如下:
关于自噬的研究技术就介绍这几种,当然还有一些其它的方法,由于篇幅的关系,这里就不做过多介绍,感兴趣的小伙伴可以自己去查阅相关资料噢!
上面简要介绍了一些关于植物蛋白自噬降解的背景知识,下面我们来看一篇文献案例,看看在研究中遇到蛋白降解这一块内容时,具体该如何研究。
2023年6月,武汉大学何光存课题组在Nature杂志上发表了一篇题为“A tripartite rheostat controls self-regulated host plant resistance to insects”的研究论文,该论文发现了首个被植物抗虫蛋白识别并激活抗性反应的昆虫效应子,并揭示了BISP-BPH14-OsNBR1互作系统精细调控抗性-生长平衡的新机制,为开发高产、抗虫水稻品种提供了重大理论和应用基础,也为其它粮食作物新型抗虫、抗病机理的研究提供了新思路。
相关名词解释:
BPHs:褐飞虱。
BPH14:水稻中分离到首个抗BPH的基因Bph14编码的蛋白。
BISP:BISP是BPH分泌的唾液蛋白,可被BPH14直接识别。
Bph14:RI35系(被命名为Bph14)是一个含有BPH抗性基因Bph14的重组自交系。
N14:日本晴(在原文中记为N14)是一个典型的粳稻品种,含有Bph14的N14易感等位基因,N14基因对应的蛋白记为N14蛋白。在早期实验中使用N14建立了BISP-BPH14相互作用的原理。
MH63:明辉63(MH63)是RI35的BPH易感亲本,是籼稻育种和基因组学研究的典范品种。
Bph14–Bisp:以Bph14 为受体材料,转入了pUBI::Bisp-Myc的转基因材料。
MG132:26S蛋白酶体抑制剂。
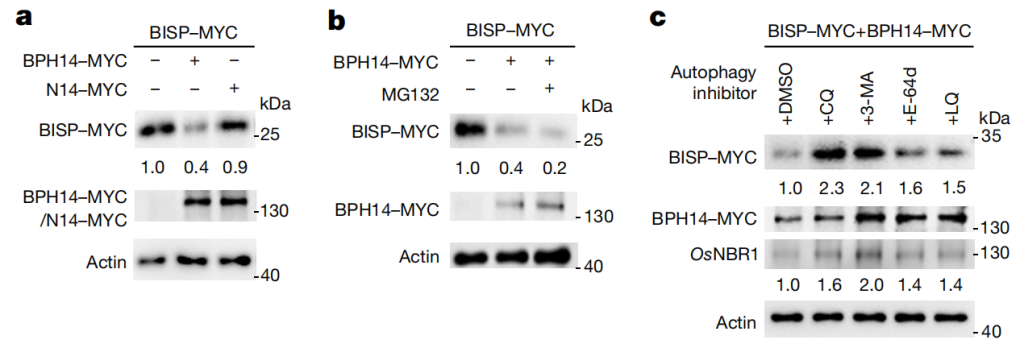
在本篇研究中,褐飞虱在取食含Bph14的水稻过程中,BISP作为昆虫效应子被BPH14识别并激活抗虫反应。植物抗性的持续激活往往会为生长发育带来不利影响,因此,水稻需要精细调控自身抗虫反应以兼顾自身的正常生长发育。那么如何平衡这两者的关系呢?作者研究后发现BISP被BPH14识别可以激活抗虫反应,同时BISP的蛋白水平也被BPH14严格控制,从而同时兼顾自身生长与抗虫反应。
图4 BISP以依赖BPH14的方式降解(Guo et al., 2023)。(a)水稻原生质体中BPH14和N14对BISP水平的影响;(b)26S蛋白酶体抑制剂MG132对水稻原生质体中BISP水平的影响;(c)自噬抑制剂对水稻原生质体中BISP蛋白水平的影响。3-MA:3-甲基腺嘌呤、CQ:氯喹、E-64d:阿洛司他丁、LQ:亮抑酶肽。以DMSO为溶剂溶解所有抑制剂。
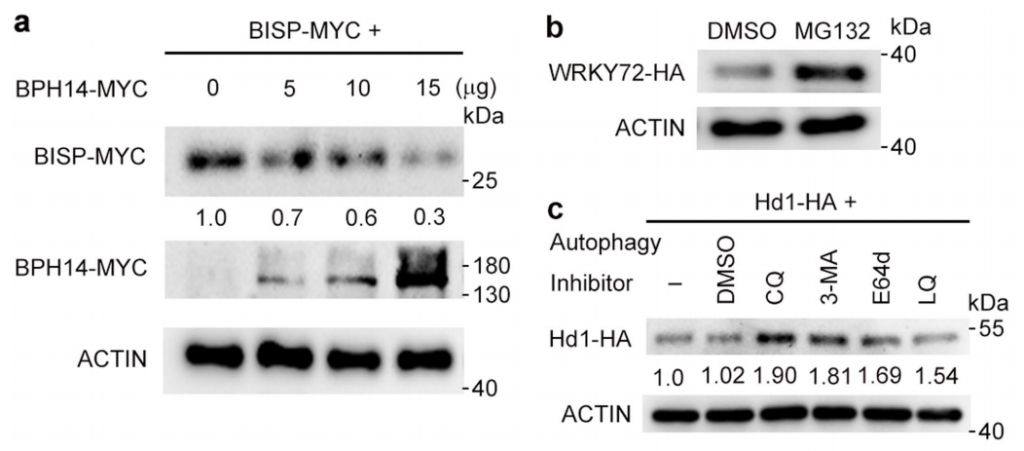
图5 (a)水稻原生质体中BPH14对BISP含量的影响。在水稻原生质体中BISP-MYC单独表达或与BPH14-MYC一起共表达。分别用抗MYC抗体和抗ACTIN抗体检测总蛋白。(b)免疫印迹显示,MG132处理阻断了WRKY72-HA的蛋白酶体降解。分别用抗HA和抗ACTIN抗体检测原生质体总蛋白。(c)免疫印迹显示,自噬抑制剂可阻断Hd1-HA的自噬降解。分别用抗HA和抗ACTIN抗体检测原生质体总蛋白(Guo et al., 2023)。
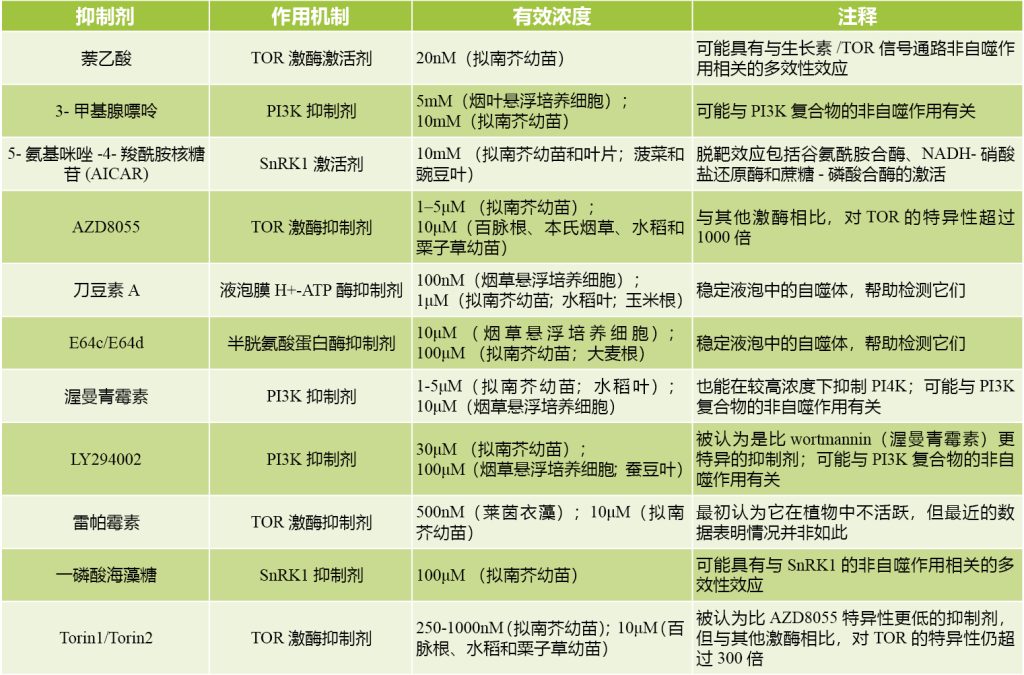
表1 植物自噬研究中常用的抑制剂和激活剂(Marshall Richard S. and Vierstra Richard D., 2018)。
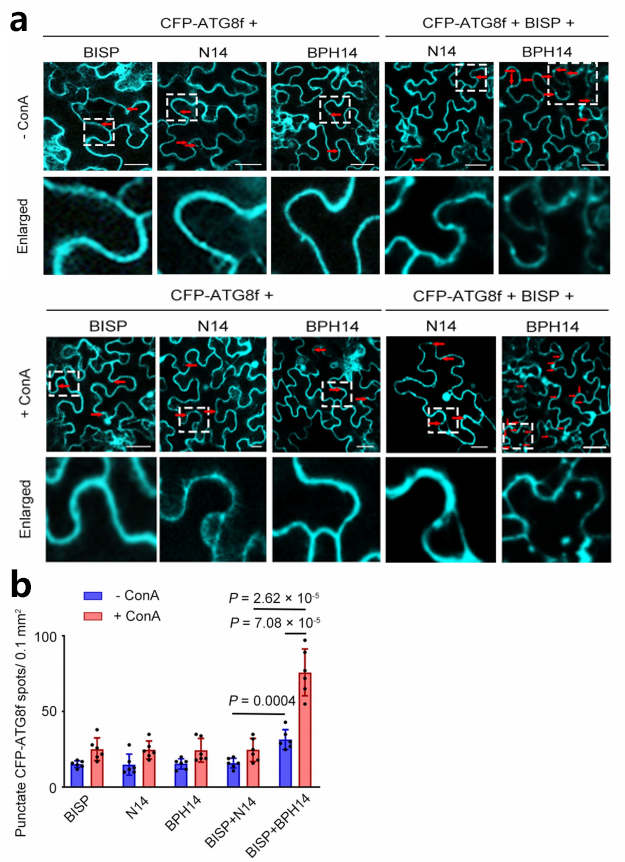
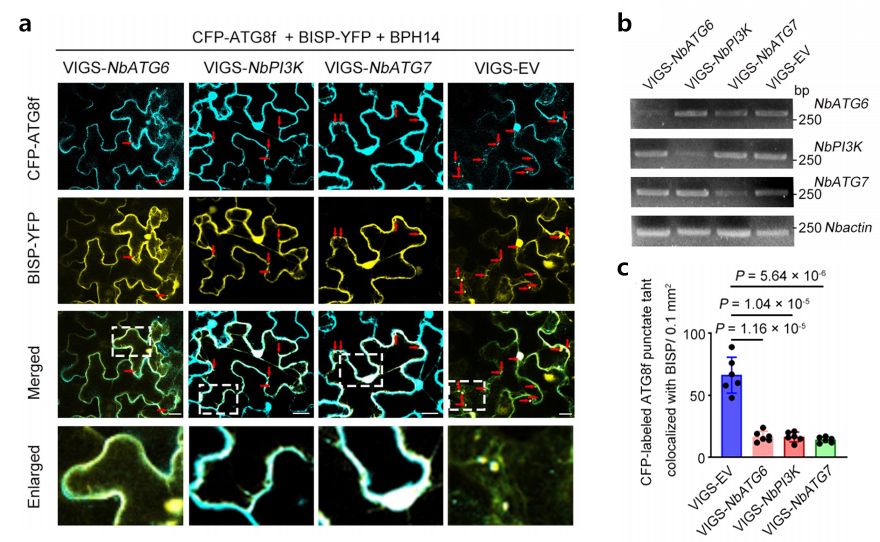
为了进一步评估自噬在BISP降解中的作用,作者使用青色荧光蛋白标记的ATG8F(CFP-ATG8F)作为监测自噬的标记物(Han et al., 2015;Klionsky et al., 2021)(图6a,b)。当CFP-ATG8F与BISP或BPH14在烟草叶片中共表达时,几乎很难检测到与自噬前体和自噬小体相对应的点状CFP结构。然而,当CFP-ATG8F与BISP和BPH14共同表达时,点状CFP结构的数量显著增加,这表明BISP和BPH14共表达诱导了自噬。这种相互作用是特异性的,因为CFP-ATG8F与BISP和N14共表达不会增加自噬小体的数量。刀豆素A(Concanamycin A, ConA)可以阻断自噬通量(Klionsky et al., 2021),在ConA处理BISP和BPH14共表达的细胞中,自噬小体的数量显著增加。当沉默核心自噬基因NbATG6、NbPI3K和NbATG7时,在共表达CFP-ATG8F和BPH14的烟草叶片中,自噬小体的数量明显减少(图7a,b,c)。
图6 (a)(上)在ConA缺失(上图)或存在(下图)时CFP-ATG8F标记的自噬点结构的共聚焦图像。研究结果显示,经农杆菌侵染烟草叶片后,CFP-ATG8f与BISP、N14或BPH14共表达,以及CFP-ATG8f和BISP与N14或BPH14共表达时细胞中的自噬小体。红色箭头表示CFP-ATG8f标记的自噬点。(下)显示放大后的自噬小体图像;(b)在存在或不存在ConA的情况下,CFP-NbATG8标记的自噬点的平均数量(Guo et al., 2023)。
图7 NbATG6、NbPI3K或NbATG7的沉默抑制了CFP-ATG8f标记的自噬点的产生(Guo et al., 2023)。(a)CFP-ATG8f标记的自噬点结构与BISP-YFP共定位的共聚焦图像。CFP-ATG8f、BISP-YFP和BPH14在沉默叶片(VIGS-NbATG6、VIGS-NbPI3K和VIGS-NbATG7)或非沉默对照叶片(VIGS-EV)中瞬时共表达。红色箭头表示CFP-ATG8f标记的自噬点与BISP-YFP共定位。(下)显示放大后的自噬小体图像;(b)通过半定量RT-PCR检测NbATG6、NbPI3K和NbATG7的转录水平。NbACTB转录水平作为对照;(c)沉默和对照叶片中CFP-NBATG8标记的自噬点的平均数量。
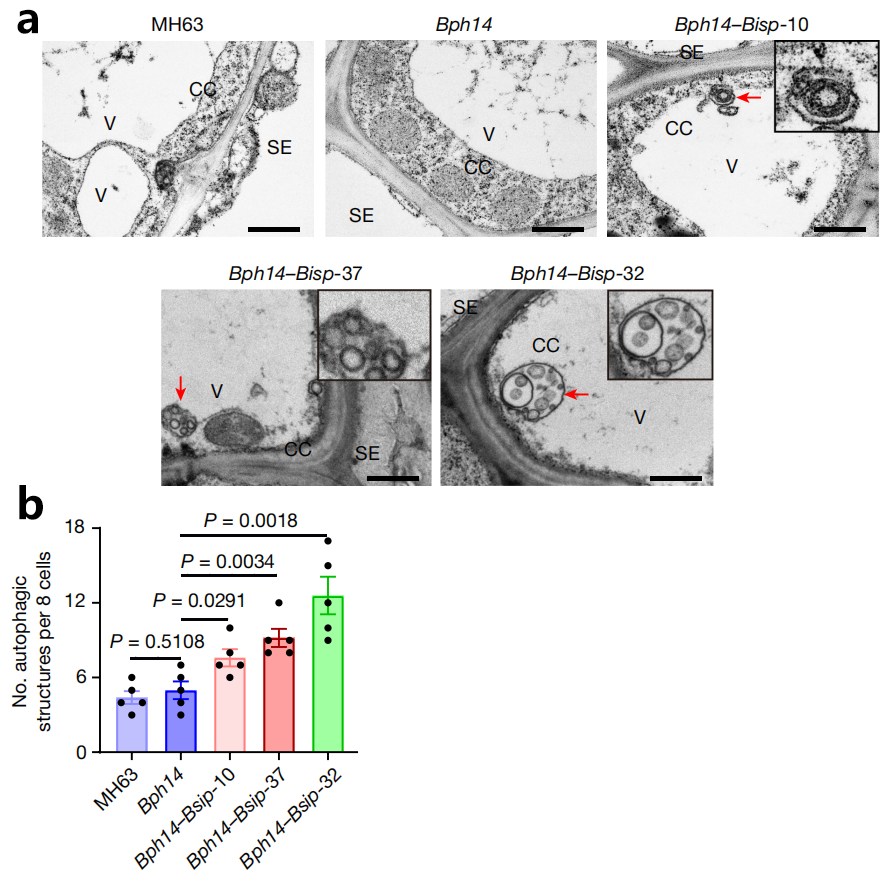
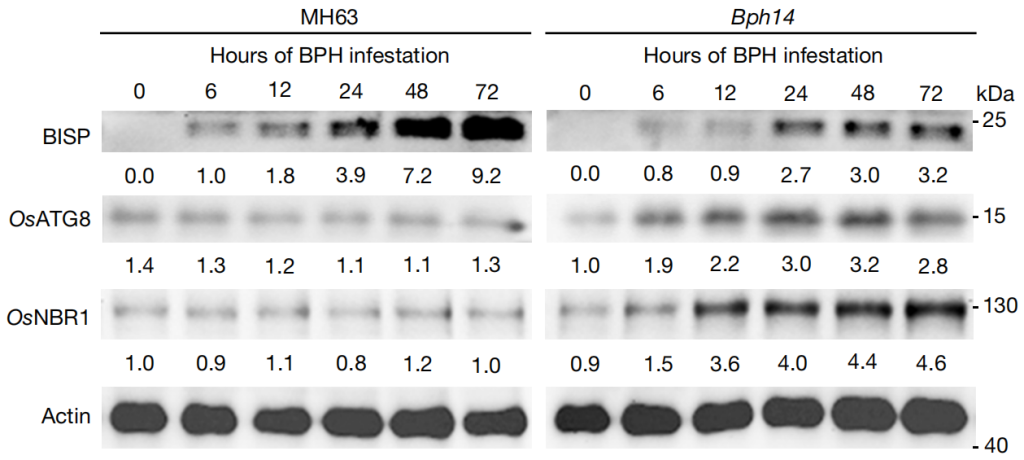
此外,通过透射电镜,作者观察到与Bph14和MH63植物相比,未感染的Bph14-Bisp植物韧皮部伴侣细胞中有更多的双膜自噬小体结构(图8a,b)。因此,OsATG8蛋白和OsATG8a、OsATG8b和OsATG8c转录本在未感染的Bph14-Bisp植物中显著高于Bph14和MH63植物中的蛋白。这些观察结果表明,BISP通过BPH14依赖的方式降解。
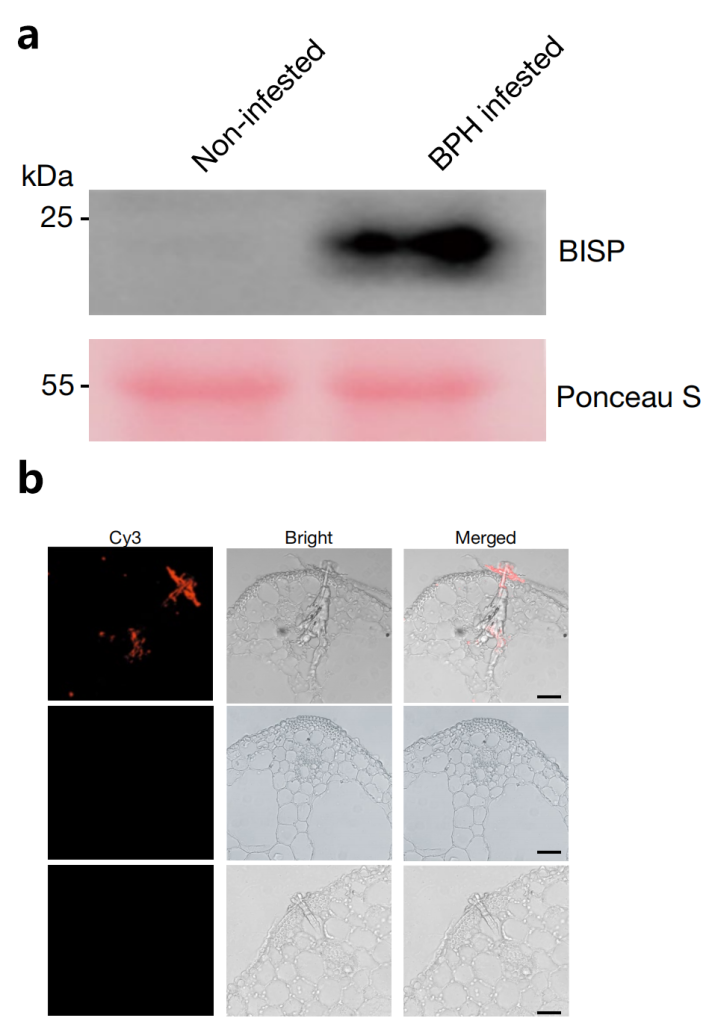
图9(a)在BPH侵染期间,BISP被输送到水稻叶鞘中。用抗BISP抗体分析叶鞘蛋白。Ponceau S染色作为上样对照;(b)免疫组化染色显示受BPH感染的水稻叶鞘中的BISP。用抗BISP抗体检测未感染(中)和感染BPH(下)的鞘,免疫前兔血清检测作为阴性对照(Guo et al., 2023)。
原文研究的内容比较多,小远这里就不再一一为大家解读了,仅挑出与蛋白自噬降解的相关实验给大家作个介绍,原文中还有更深入的机制研究,大家感兴趣的话可以自行阅读噢!
References:
Ashford T P, Porter K R. Cytoplasmic components in hepatic cell lysosomes[J]. The Journal of cell biology, 1962, 12(1): 198.
Bassham D C, Laporte M, Marty F, et al. Autophagy in development and stress responses of plants[J]. Autophagy, 2006, 2(1): 2-11.
Chung T. How phosphoinositides shape autophagy in plant cells[J]. Plant Science, 2019, 281: 146-158.
De Duve C, Pressman B C, Gianetto R, et al. Tissue fractionation studies. 6. Intracellular distribution patterns of enzymes in rat-liver tissue[J]. Biochemical Journal, 1955, 60(4): 604.
Dice J F. Chaperone-mediated autophagy[J]. Autophagy, 2007, 3(4): 295-299.
Guo J, Wang H, Guan W, et al. A tripartite rheostat controls self-regulated host plant resistance to insects[J]. Nature, 2023: 1-9.
Hamasaki M, Furuta N, Matsuda A, et al. Autophagosomes form at ER–mitochondria contact sites[J]. Nature, 2013, 495(7441): 389-393.
Han S, Wang Y, Zheng X, et al. Cytoplastic glyceraldehyde-3-phosphate dehydrogenases interact with ATG3 to negatively regulate autophagy and immunity in Nicotiana benthamiana[J]. The Plant Cell, 2015, 27(4): 1316-1331.
Klionsky D J, Abdel-Aziz A K, Abdelfatah S, et al. Guidelines for the use and interpretation of assays for monitoring autophagy[J]. Autophagy, 2021, 17(1): 1-382.
Klionsky D J, Emr S D. Autophagy as a regulated pathway of cellular degradation[J]. Science, 2000, 290(5497): 1717-1721.
Le Bars R, Marion J, Le Borgne R, et al. ATG5 defines a phagophore domain connected to the endoplasmic reticulum during autophagosome formation in plants[J]. Nature communications, 2014, 5(1): 4121.
Liu Y, Bassham D C. Autophagy: pathways for self-eating in plant cells[J]. Annual review of plant biology, 2012, 63: 215-237.
Luo H, Wong J, Wong B. Protein degradation systems in viral myocarditis leading to dilated cardiomyopathy[J]. Cardiovascular research, 2010, 85(2): 347-356.
Malinova I, Qasim H M, Brust H, et al. Parameters of starch granule genesis in chloroplasts of Arabidopsis thaliana[J]. Frontiers in Plant Science, 2018, 9: 761.
Marshall R S, Vierstra R D. Autophagy: the master of bulk and selective recycling[J]. Annual review of plant biology, 2018, 69: 173-208.
Marshall R S, Vierstra R D. Autophagy: the master of bulk and selective recycling[J]. Annual review of plant biology, 2018, 69: 173-208.
Masclaux-Daubresse C, Chen Q, Havé M. Regulation of nutrient recycling via autophagy[J]. Current opinion in plant biology, 2017, 39: 8-17.
Mitou G, Budak H, Gozuacik D. Techniques to study autophagy in plants[J]. International Journal of Plant Genomics, 2009, 2009.
Ohsumi Y. Molecular dissection of autophagy: two ubiquitin-like systems[J]. Nature reviews Molecular cell biology, 2001, 2(3): 211-216.
Poxleitner M, Rogers S W, Lacey Samuels A, et al. A role for caleosin in degradation of oil‐body storage lipid during seed germination[J]. The Plant Journal, 2006, 47(6): 917-933.
Tang J, Bassham D C. Autophagy in crop plants: what’s new beyond Arabidopsis?[J]. Royal Society Open Biology, 2018, 8(12): 180162.
Tsukada M, Ohsumi Y. Isolation and characterization of autophagy-defective mutants of Saccharomyces cerevisiae[J]. FEBS letters, 1993, 333(1-2): 169-174.
Van der Wilden W, Herman E M, Chrispeels M J. Protein bodies of mung bean cotyledons as autophagic organelles[J]. Proceedings of the National Academy of Sciences, 1980, 77(1): 428-432.
van Doorn W G, Papini A. Ultrastructure of autophagy in plant cells: a review[J]. Autophagy, 2013, 9(12): 1922-1936.
van Wersch S, Tian L, Hoy R, et al. Plant NLRs: the whistleblowers of plant immunity[J]. Plant Communications, 2020, 1(1).
Wang P, Mugume Y, Bassham D C. New advances in autophagy in plants: regulation, selectivity and function[C]//Seminars in cell & developmental biology. Academic Press, 2018, 80: 113-122.
Yoshimoto K, Ohsumi Y. Unveiling the molecular mechanisms of plant autophagy—from autophagosomes to vacuoles in plants[J]. Plant and Cell Physiology, 2018, 59(7): 1337-1344.
Zhuang X, Chung K P, Cui Y, et al. ATG9 regulates autophagosome progression from the endoplasmic reticulum in Arabidopsis[J]. Proceedings of the National Academy of Sciences, 2017, 114(3): E426-E435.

-3-852x1024.png)