酵母单杂、酵母双杂、酵母三杂,仅仅一个字的区别,你对它们了解吗?这些经常用到的实验,它们的原理你确定都搞清楚了吗?如果没有,那么今天你就来对地方了,因为伯小远下面打算非常详细的给你讲一遍它们的原理,让你以后不再为不懂原理而烦恼!
在了解酵母杂交实验之前我们先来了解一个转录因子——Gal4,了解它可以帮助大家更好的理解酵母杂交实验的原理!
Gal4转录因子
真核基因转录受大量调控因子调节。进化保守的Gal4是真核细胞普遍存在的转录激活因子。在半乳糖的诱导下,酵母Gal4结合在半乳糖代谢酶系GAL基因启动子的上游激活序列(Upstream activating sequence,UAS)上,调控Galactokinase(Gal 1),Galactose permease(Gal2),Galactose 1-phosphate uridyltransferase(Gal7),Galactose mutarotase and UDP-galactose 4-epimerase(Gal10)等基因的转录。
酵母单杂交与酵母双杂交
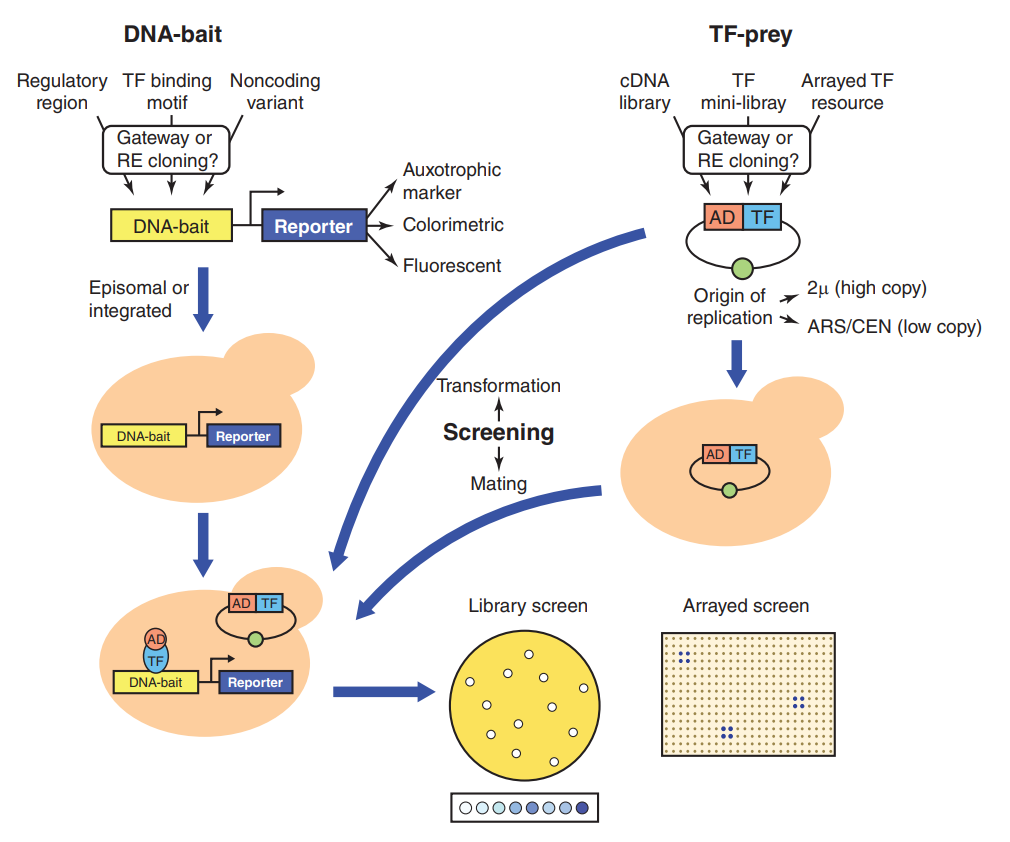
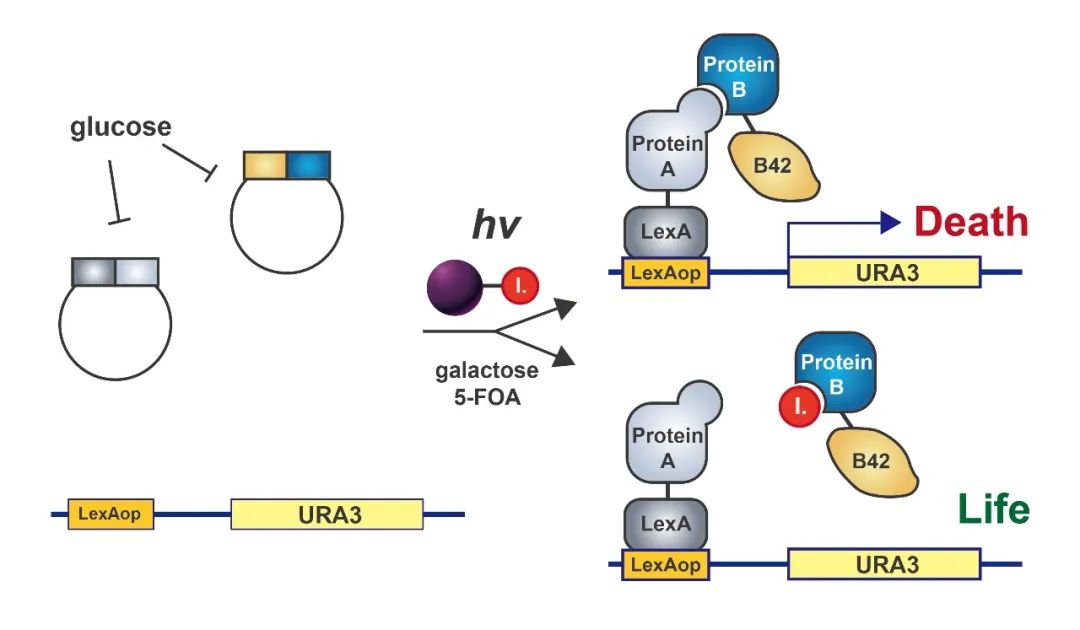
 图1 酵母单杂实验原理(Ouwerkerk, Pieter BF.and Annemarie H., 2001)(不同的实验体系可能会存在一些差异,但原理基本都差不多,这里主要帮助大家理解原理)。
图1 酵母单杂实验原理(Ouwerkerk, Pieter BF.and Annemarie H., 2001)(不同的实验体系可能会存在一些差异,但原理基本都差不多,这里主要帮助大家理解原理)。
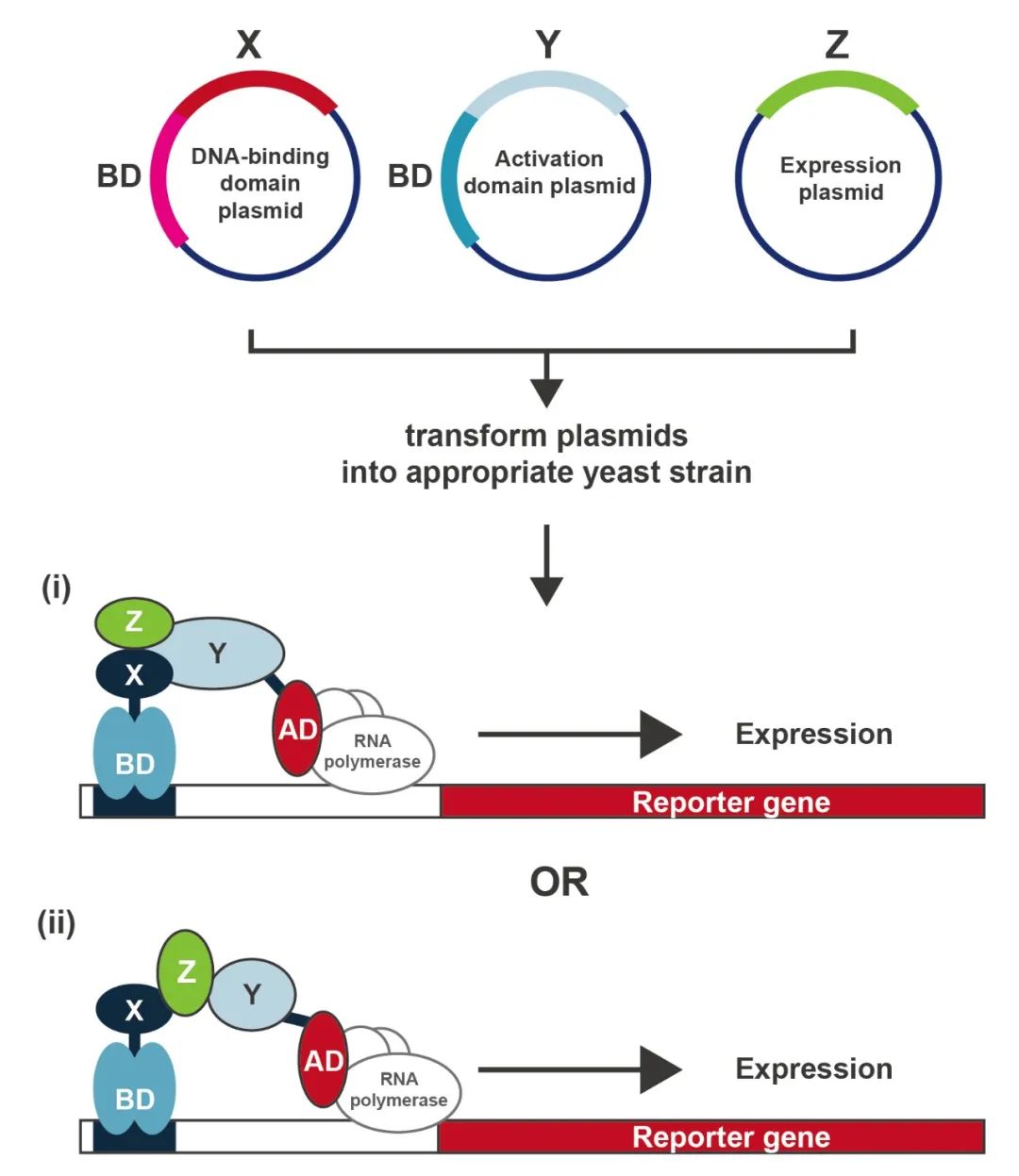
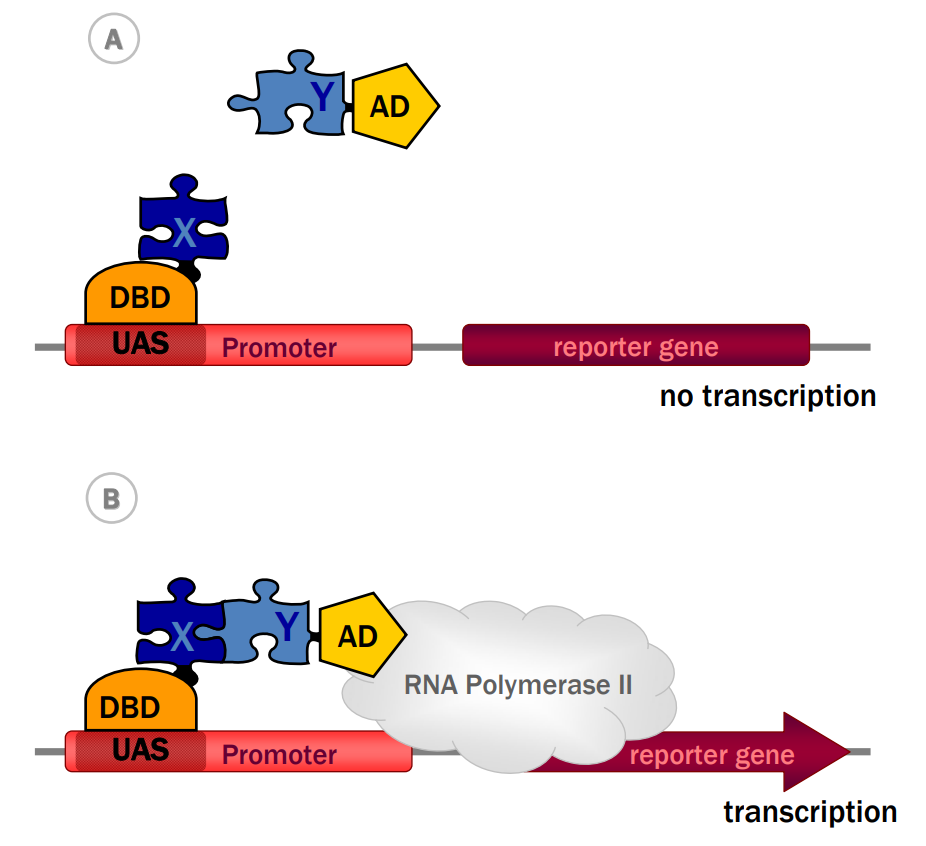 图3 酵母双杂实验原理(Brückner et al., 2009)。(A)感兴趣的蛋白质X,被融合到DNA结合域(DBD),该结构被称为诱饵。潜在的相互作用蛋白Y被融合到激活域(AD)上,该结构被称为猎物。(B)诱饵,即DBD-X融合蛋白,结合启动子的上游激活子序列(UAS)。诱饵与猎物(即AD-Y融合蛋白)的相互作用,招募AD,从而重建一个功能性转录因子,导致RNA聚合酶II的进一步招募和报告基因的后续转录。
图3 酵母双杂实验原理(Brückner et al., 2009)。(A)感兴趣的蛋白质X,被融合到DNA结合域(DBD),该结构被称为诱饵。潜在的相互作用蛋白Y被融合到激活域(AD)上,该结构被称为猎物。(B)诱饵,即DBD-X融合蛋白,结合启动子的上游激活子序列(UAS)。诱饵与猎物(即AD-Y融合蛋白)的相互作用,招募AD,从而重建一个功能性转录因子,导致RNA聚合酶II的进一步招募和报告基因的后续转录。
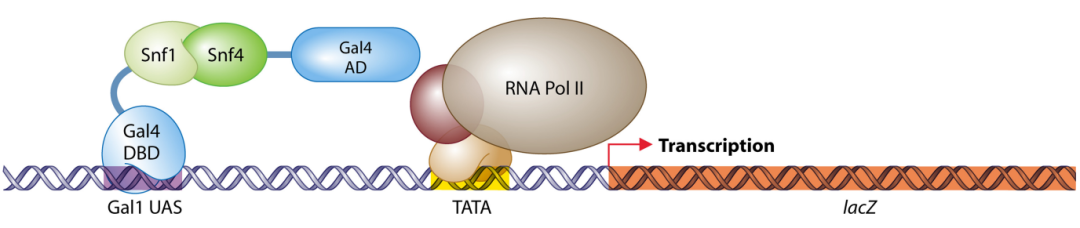
图4 蛋白SNF1与SNF4互作示意图(Stynen et al., 2012) 。
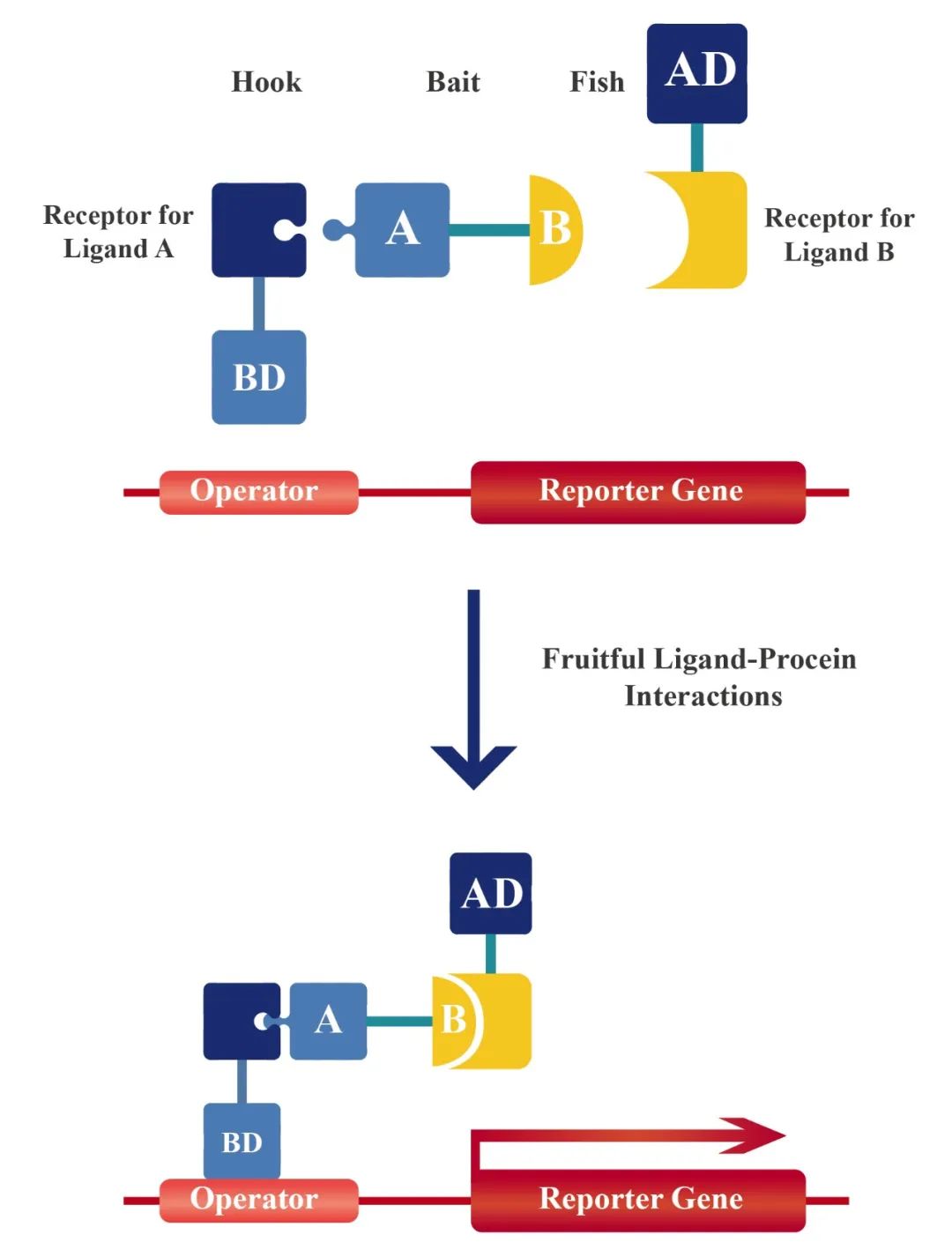
自从酵母双杂交技术开发以来,就被证明是检测蛋白质相互作用的有力工具。在另一方面,有机配体的二聚体可以导致与受体融合的两个独立蛋白质的二聚或寡聚。为了使酵母双杂交系统能够用于研究小分子配体与受体的相互作用,研究者设想使用两个不同的小有机配体合成异源二聚体作为第三个杂化分子,使DNA结合结构域与其中一个配体的受体融合,激活结构域与第二个配体的受体融合,从而激活报告基因。该系统被称之为酵母三杂交系统,可以用于直接识别编码感兴趣的配体的受体cDNA,或筛选与特定受体结合的新配体。
将酵母双杂系统进行简单的改造之后,除了上面讲到的用于研究受体与配体之间的互作之外,还可以用来研究3个蛋白之间的互作,具体信息如下:
小分子配体在探索蛋白质的细胞功能方面具有不可估量的价值。这些配体的结合导致其目标蛋白的激活或失活,类似于相应基因中功能获得或丧失突变所产生的效应。利用具有细胞渗透性的小分子配体来改变蛋白质的功能是很有吸引力的,因为其固有的条件可以通过添加或移除这种化合物来简单、快速地控制目标蛋白的活性。为了研究目标蛋白对应的小分子配体是什么,在酵母双杂的基础上开发以下方法:
酵母三杂
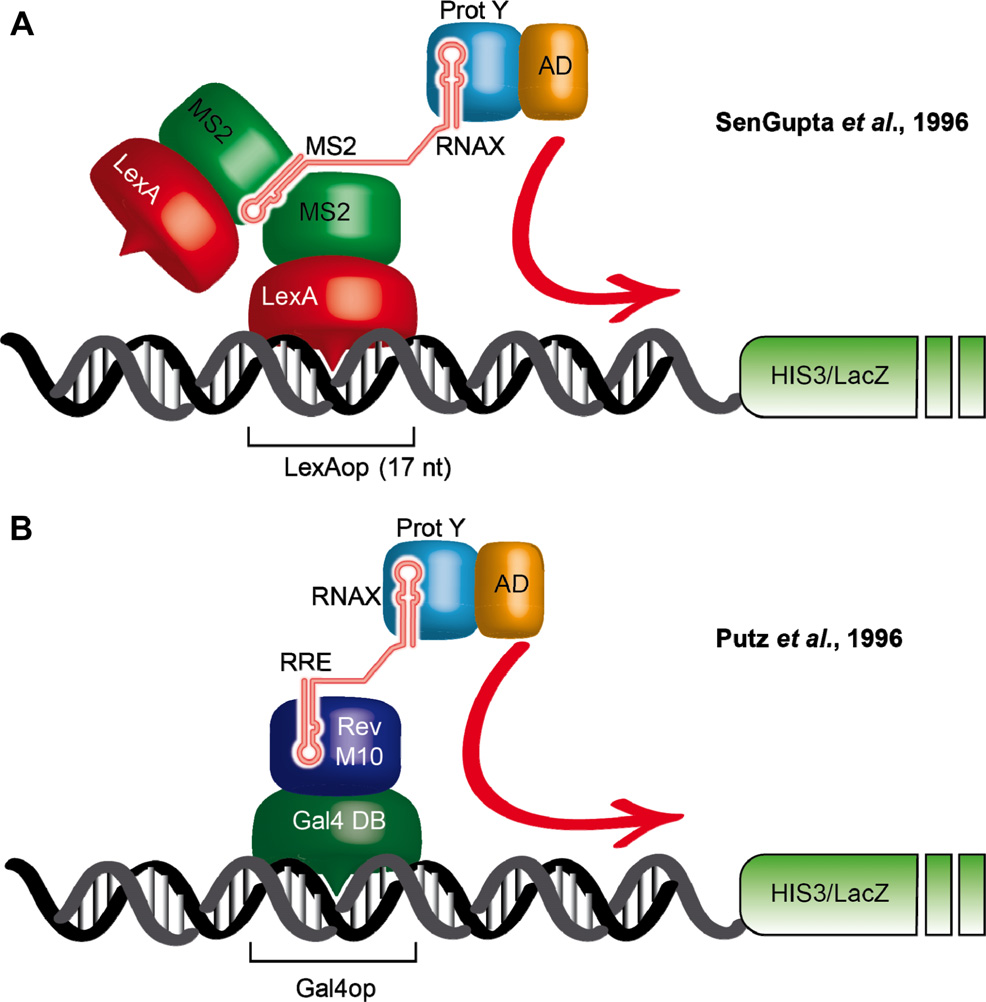
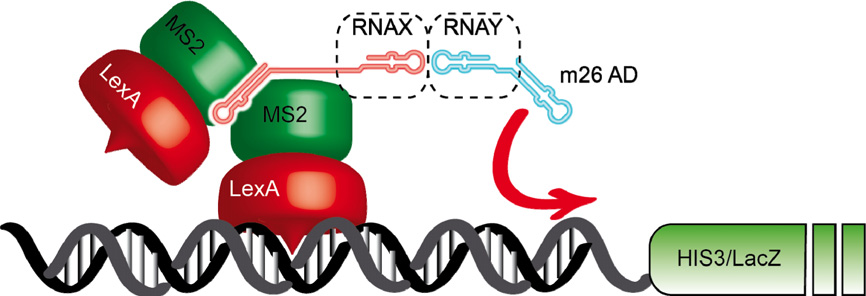
图9 酵母三杂交系统(Brückner et al., 2009)。(A)SenGupta等人设计的酵母三杂交系统(SenGupta et al., 1996)。第一个杂交蛋白包含LexA DNA结合域和噬菌体MS2外壳蛋白(CP)。MS2 CP必须形成二聚体才能与RNA靶点结合。第二个RNA杂交分子包含一个MS2结合位点,该位点可以与任何感兴趣的RNA X相连。第三个杂交蛋白是蛋白质Y与RNA诱饵和Gal4激活域(AD)相互作用的融合蛋白。RNA X与蛋白Y的相互作用导致在17-nt LexA操作子(LexAop)上组装成一个完整的反式激活复合物,该复合物位于两个报告基因HIS3和LacZ的上游。8和4个LexAop分别位于LacZ基因和HIS3基因的上游。(B)在Putz等人1996年的系统中,第一个杂交蛋白由与HIV Rev M10突变蛋白融合的Gal4 DNA结合域组成。该蛋白与其RNA靶标,Rev反应元件(RRE)结合。第三个杂交蛋白和报告基因在两个系统中是相同的。
酵母三杂交筛库体系不仅可以利用RNA作为诱饵去筛选与之互作的蛋白,也可以利用蛋白作为诱饵去筛选与其互作的RNA。另外,由于某些RNA具有激活报告基因转录的能力,这意味着它们不需要任何AD蛋白就能触发报告基因的表达。根据这一特性设计出了一种新型RNA杂交系统用于检测RNA-RNA相互作用,其原理如下:
Brückner A, Polge C, Lentze N, et al. Yeast two-hybrid, a powerful tool for systems biology[J]. International journal of molecular sciences, 2009, 10(6): 2763-2788.
Causier B, Davies B. Analysing protein-protein interactions with the yeast two-hybrid system[J]. Plant molecular biology, 2002, 50(6): 855-870.
Fields S, Song O. A novel genetic system to detect protein–protein interactions[J]. Nature, 1989, 340(6230): 245-246.
Huang J, Schreiber S L. A yeast genetic system for selecting small molecule inhibitors of protein–protein interactions in nanodroplets[J]. Proceedings of the National Academy of Sciences, 1997, 94(25): 13396-13401.
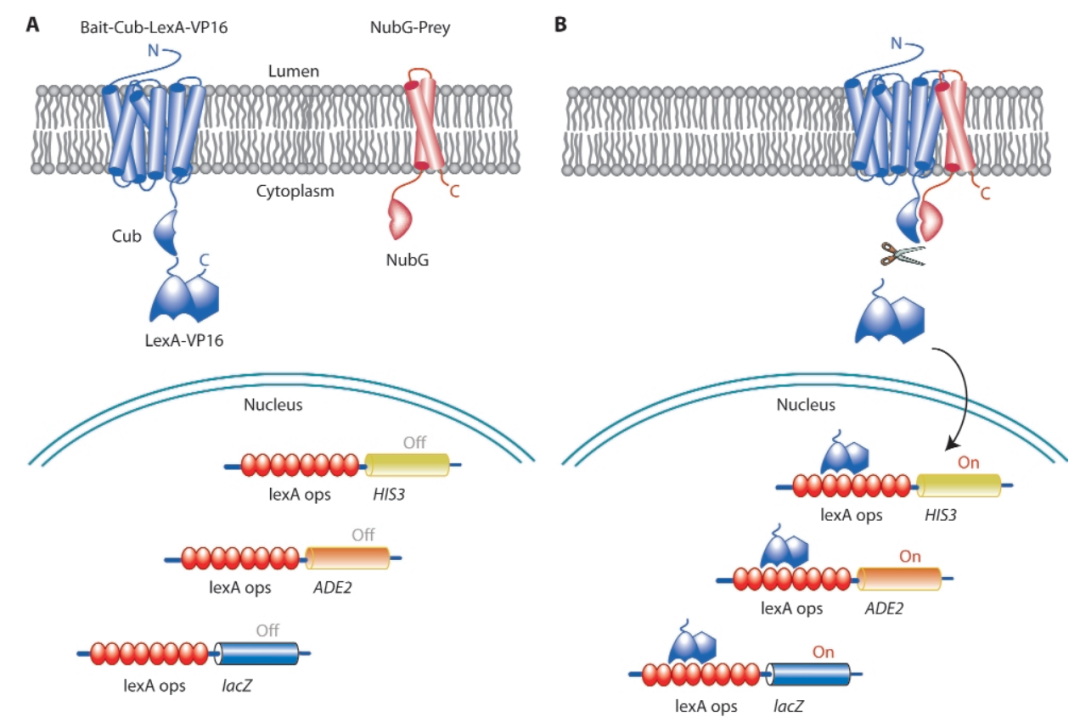
Iyer K, Burkle L, Auerbach D, et al. Utilizing the split-ubiquitin membrane yeast two-hybrid system to identify protein-protein interactions of integral membrane proteins[J]. Science’s STKE, 2005, 2005(275): pl3-pl3.
Johnsson N, Varshavsky A. Split ubiquitin as a sensor of protein interactions in vivo[J]. Proceedings of the National Academy of Sciences, 1994, 91(22): 10340-10344.
Keegan L, Gill G, Ptashne M. Separation of DNA binding from the transcription-activating function of a eukaryotic regulatory protein[J]. Science, 1986, 231(4739): 699-704.
Licitra E J, Liu J O. A three-hybrid system for detecting small ligand–protein receptor interactions[J]. Proceedings of the National Academy of Sciences, 1996, 93(23): 12817-12821.
Melcher K, Johnston S A. GAL4 interacts with TATA-binding protein and coactivators[J]. Molecular and Cellular Biology, 1995, 15(5): 2839-2848.
Ouwerkerk P B F, Meijer A H. Yeast one‐hybrid screening for DNA‐protein interactions[J]. Current protocols in molecular biology, 2001, 55(1): 12.12. 1-12.12. 12.
Putz U, Skehel P, Kuhl D. A tri-hybrid system for the analysis and detection of RNA-protein interactions[J]. Nucleic acids research, 1996, 24(23): 4838-4840.
SenGupta D J, Zhang B, Kraemer B, et al. A three-hybrid system to detect RNA-protein interactions in vivo[J]. Proceedings of the national Academy of Sciences, 1996, 93(16): 8496-8501.
Sewell J A, Fuxman Bass J I. Options and considerations when using a yeast one-hybrid system[M]//Two-Hybrid Systems. Humana Press, New York, NY, 2018: 119-130.
Stagljar I, Korostensky C, Johnsson N, et al. A genetic system based on split-ubiquitin for the analysis of interactions between membrane proteins in vivo[J]. Proceedings of the National Academy of Sciences, 1998, 95(9): 5187-5192.
Stynen B, Tournu H, Tavernier J, et al. Diversity in genetic in vivo methods for protein-protein interaction studies: from the yeast two-hybrid system to the mammalian split-luciferase system[J]. Microbiology and molecular biology reviews, 2012, 76(2): 331-382.
Wu Y, Reece R J, Ptashne M. Quantitation of putative activator‐target affinities predicts transcriptional activating potentials[J]. The EMBO journal, 1996, 15(15): 3951-3963.
官网链接:plant.biorun.com