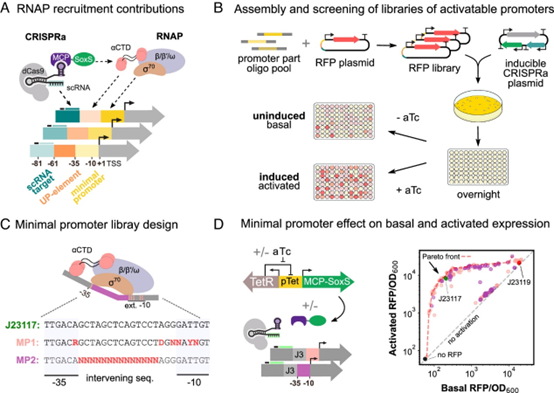
首先,作者对影响合成可激活启动子的RNAP(RNA聚合酶)招募的序列进行表征,以及对其基础表达水平和激活后表达水平的影响。RNAP招募受到RNAP sigma亚单位(σ)对最小启动子(−10和−35六聚体)的亲和力影响。此外,招募还受到−10和−35位点之间介导序列的GC含量的影响。启动子识别受到位于最小启动子上游的AT丰富的UP元件的增强,这些元件可以锚定RNAP的α亚单位。总体而言,这些区域的序列组成影响了RNAP在启动子上的招募、结合和启动转录。
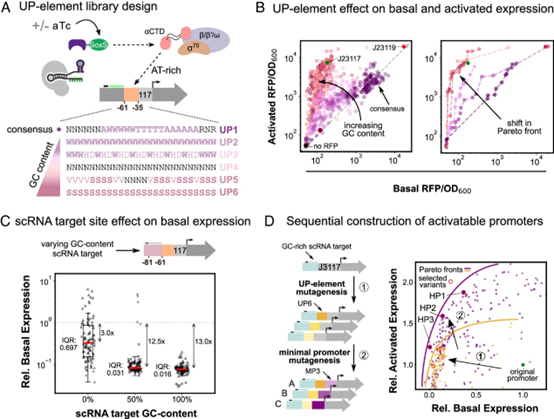
RNA聚合酶(RNAP)启动子识别受到位于最小启动子上游的富含AT碱基的UP元件的增强作用,该元件可以锚定RNAP的α亚单位(40, 48, 54)。对于有效的CRISPRa,RNAP应仅在存在目标序列特异性的scRNA时被招募到启动子上。因此,对于SoxS的转录激活,可以通过减少RNAP-UP元件之间的相互作用和降低基础表达水平来改善动态范围。作者设计了五个UP元件文库,通过突变富含AT碱基的大肠杆菌共识序列,使其含有不断增加的GC含量。正如预期的那样,共识UP元件和富含AT碱基的文库具有最高的基础表达水平。与更富含GC的文库相比,这些文库仅表现出三倍的激活效果。
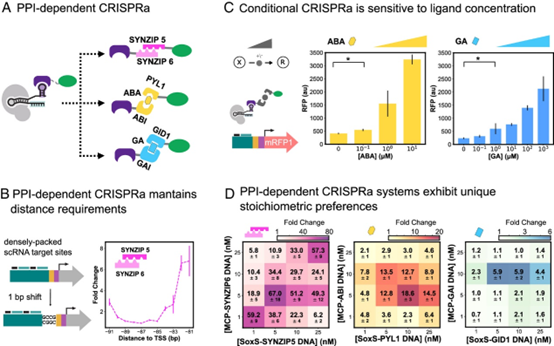
作者将三个先前表征的蛋白质-蛋白质异二聚化结构域整合到CRISPRa系统,将这些异源二聚化结构域融合到SoxS和MCP中,使SoxS能够有条件地募集到CRISPRa复合物中。为SYNZIP-CRISPRa生成了MCP-SZP6和SoxS-SZP5结构域,为ABA-CRISPRa生成了MCP-ABI和SoxS-PYL1结构域,为GA-CRISPRa生成了MCP-GAI和SoxS-GID1。在最初的J3启动子中,当同源SYNZIP融合到MCP (MCP-SYNZIP)和SoxS(SoxS-SYNZIP)的C末端时,观察到5.7倍的激活。这种取向组合提供了最佳的激活效果,而当MCP以相反的方向融合时,激活率为2.8倍(SYNZIP-MCP和SoxS-SYNZIP),当SoxS以相反的方向融合时为1.4倍(MCP-SYNZIP&SYNZIP-SoxS)。当SoxS位于N端时,ABA-CRISPRa的折叠激活也最大化。对于ABA-和GA-CRISPRa,由于MCP-SYNZIP结果和在CRISPRa系统中使用C端MCP融合的先例,仅测试了C端MCP融合。
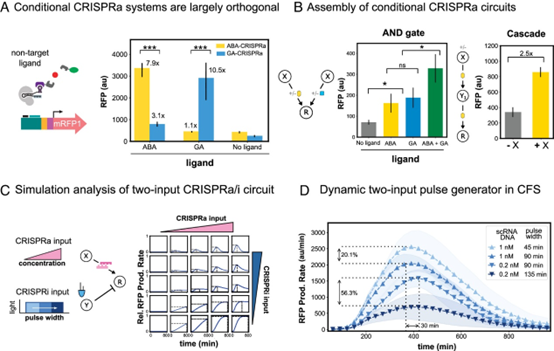
作者构建了两种类型的输入响应路径,旨在探索CRISPRa系统在多种环境刺激和多层级环境刺激处理中的应用:一个AND类逻辑门和一个CRISPRa级联路径。首先对CRISPRa系统的scRNA剂量-反应曲线进行了表征。ABA-CRISPRa对其目标配体高度特异,当存在GA时没有显著的激活(P = 0.21)。GA-CRISPRa在ABA存在时显示出3.1倍的交叉激活,与真核生物的报道一致。尽管如此,GA-CRISPRa对其对应配体仍保持三倍的高特异性,使得其在GA存在时激活增加了10.5倍。这些结果表明ABA-CRISPRa和GA-CRISPRa系统可用于正向的基因调控。
自然生物系统已经进化出含有各种可激活启动子的基因调控网络,能够对不断变化的环境条件做出动态反应。可激活启动子工程能够动态调控靶标基因的大型网络表达水平。CRISPRa/i 基因调控网络可以与现有的医疗诊断集成,以实现复杂的多种刺激信号处理。展望未来,这项工作可以促进细胞合成和工程生物材料的发展,为合成生物学和生物技术应用带来了新的可能性。
文章来源:植物生物技术Pbj