随着植物基因编辑技术的发展,科学家们开始尝试通过编辑基因的非编码区或编码区创制具有不同数量性状的新种质资源,甚至,有时候可以创造出基于相同位点的全新性状,这对于基因功能研究和分子育种应用都具有重大的意义,下面和伯小远一起来看看吧。
本次推文的主要内容是:
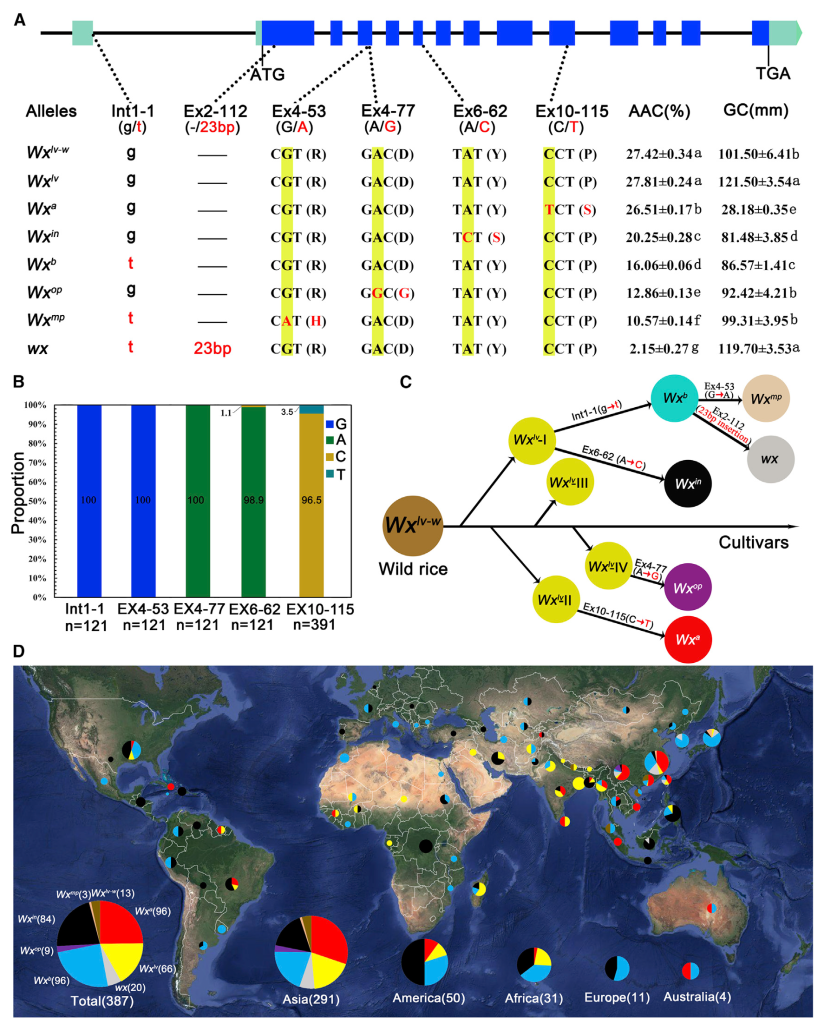
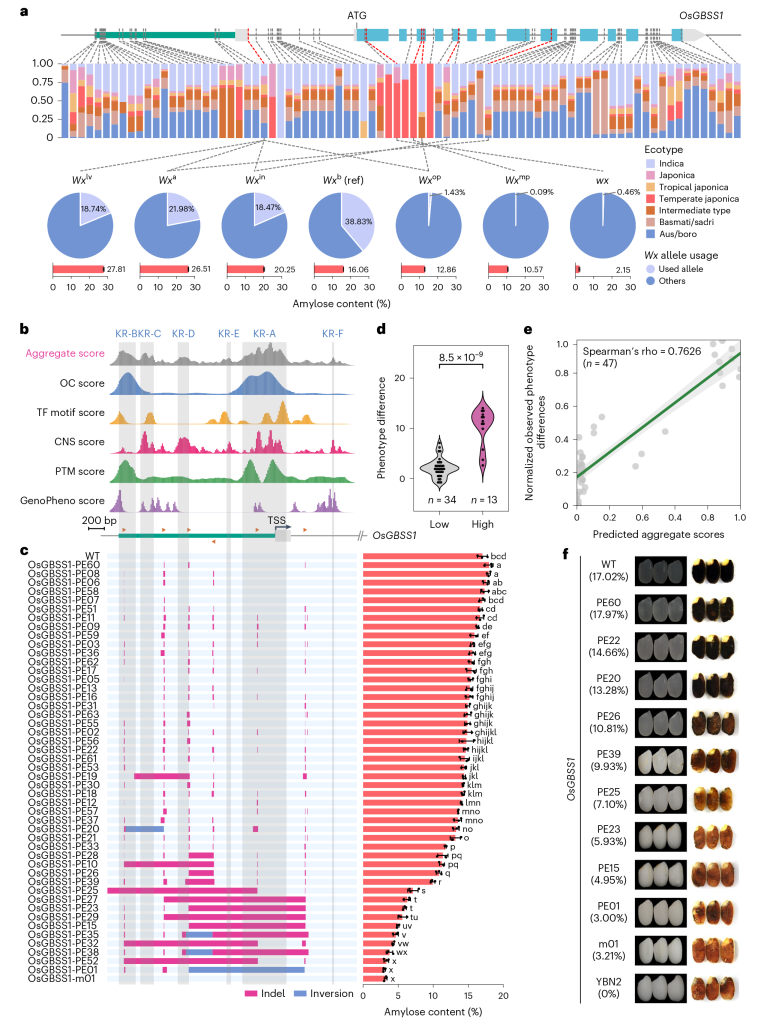
水稻胚乳中的直链淀粉由蜡质基因(Waxy,Wx)编码的颗粒结合型淀粉合成酶I(GBSSI)催化合成。Wx是决定稻米蒸煮食味品质最重要的基因 (Tian et al., 2009)。Wx的自然等位变异基因十分丰富(图1A)。自然界中稻米的AC在0-35%的范围浮动,AC通常与稻米蒸煮食味品质成反比,但AC却不是越低越好。在世界范围内,水稻的驯化过程遵循着从高AC到低AC的变化 (Zhang et al., 2019)。
图1 水稻中Wx基因的自然等位变异和进化关系 (Zhang et al., 2019)。(A)不同Wx等位基因的基因型和表型:最上方是Wx基因的基因组结构,下面列出了每个等位基因中6个功能多态性位点的核苷酸序列和编码氨基酸序列,右侧对应着携带相应Wx等位基因的近等基因系的成熟种子的表观直链淀粉含量(AAC)和胶稠度(GC)数据;(B)在野生稻样品中Wx基因的5个功能多态性位点核苷酸的分布,底部数字表示分析每个位点使用的样本数量;(C)水稻中不同Wx等位基因之间的进化关系;(D)不同Wx等位基因在世界范围内的分布:不同的颜色代表不同的Wx等位基因,每个饼状图的大小表示样本的数量,底部的饼状图总结了多个Wx等位基因在世界各地的分布情况,括号内的数字表示被测试品种的数量。
备注:表观直链淀粉含量(AAC)是指用碘比色法测得的直链淀粉含量,直链淀粉含量(AC)是指稻米淀粉中实际的直链淀粉含量。
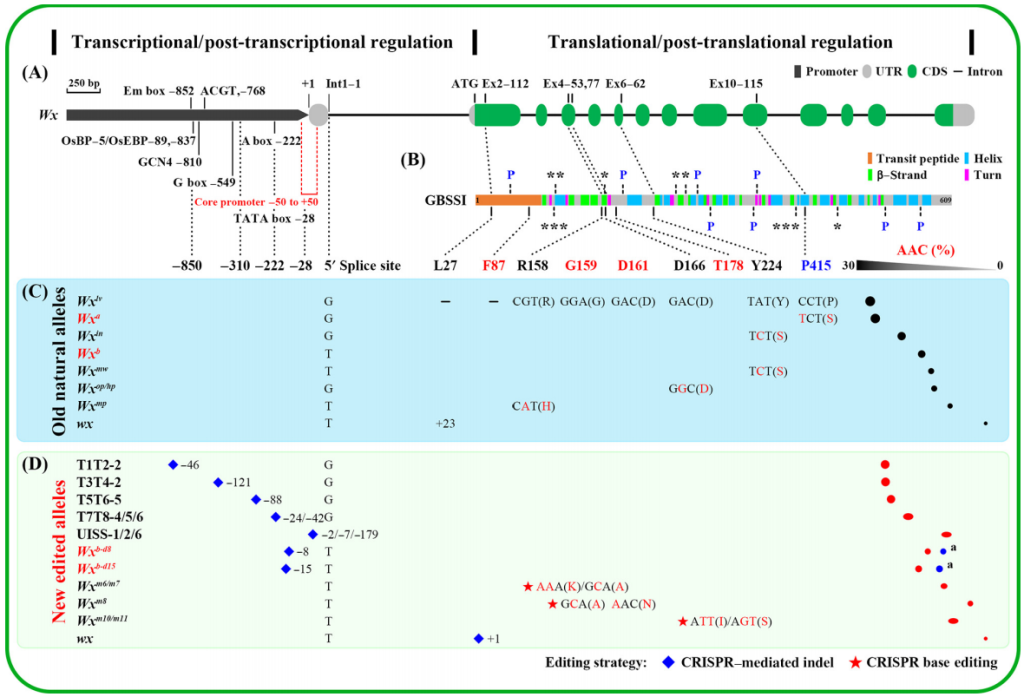
随着对Wx基因的自然变异及调控机制的深入认识使科学家们能够选择更合适的目标位点进行编辑。最开始科学家们是对Wx基因的CDS序列进行编辑 (Zhang et al., 2019),之后,有更多关于对Wx基因的启动子 (Huang et al., 2020)进行编辑、5’UTR (Zeng et al., 2020)进行编辑、CDS区进行精准编辑(CBE系统)(Xu et al., 2021)的报道,感兴趣的同学可以自行查看相应的文献喔!在这些研究中,我们可以看到人工创制的新种质资源可以对AAC含量进行微调(图2D),满足人们对稻米口感多元化的需求。
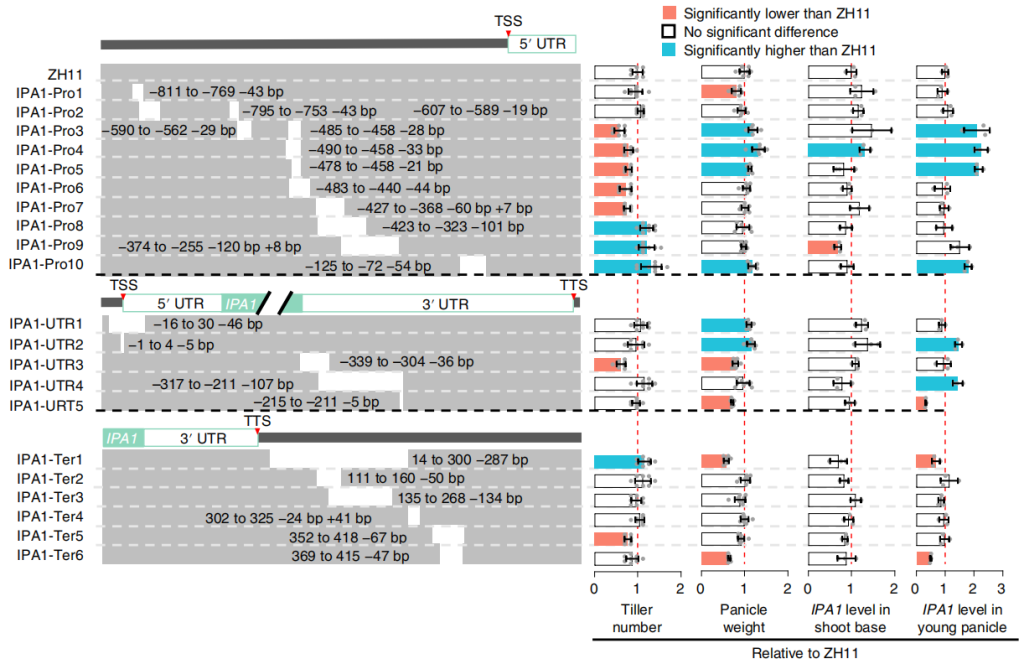
IPA1基因是一个典型的多效基因,在自然条件下,分蘖数和穗粒数是制约关系,但通过利用平铺删除策略编辑IPA1的顺式调控区(图3),得到了一个可以同时提高分蘖数和穗粒数的编辑材料IPA1-Pro10,成功实现了水稻穗部和分蘖的制约关系的解除 (Song et al., 2022)。
这个例子告诉我们,通过编辑基因的非编码区,可能像Wx基因那样得到不同数量性状的新种质资源,也可能像IPA1基因得到基于相同位点的全新性状,这些都为我们创制全新的遗传资源提供了新思路。
2023年6月,华中农业大学李国田团队和美国加州大学戴维斯分校Pamela C. Ronald团队合作在Nature上发表了题为“Genome editing of a rice CDP-DAG synthase confers multipathogen resistance”的研究论文,作者通过将人工诱变技术与全基因组测序技术相结合,克隆了一个水稻广谱抗病基因RBL1 (Sha et al., 2023)。RBL1编码胞苷二磷酸-二酰甘油(CDP-DAG)合成酶,该酶是一个重要的磷脂代谢调节枢纽,文章中也对其机制作了详细研究,感兴趣的同学可以自行查看文献喔,我们主要来看看作者通过基因编辑创制新种质的那部分工作。
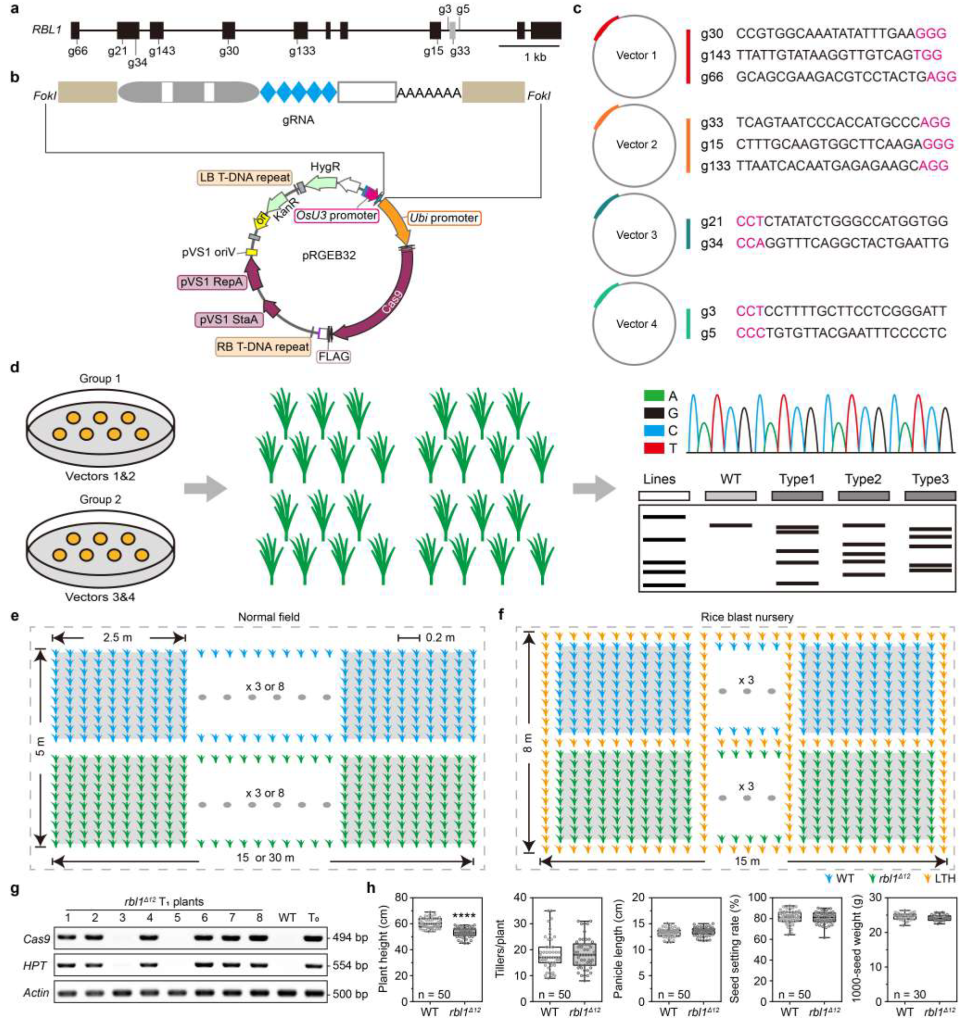
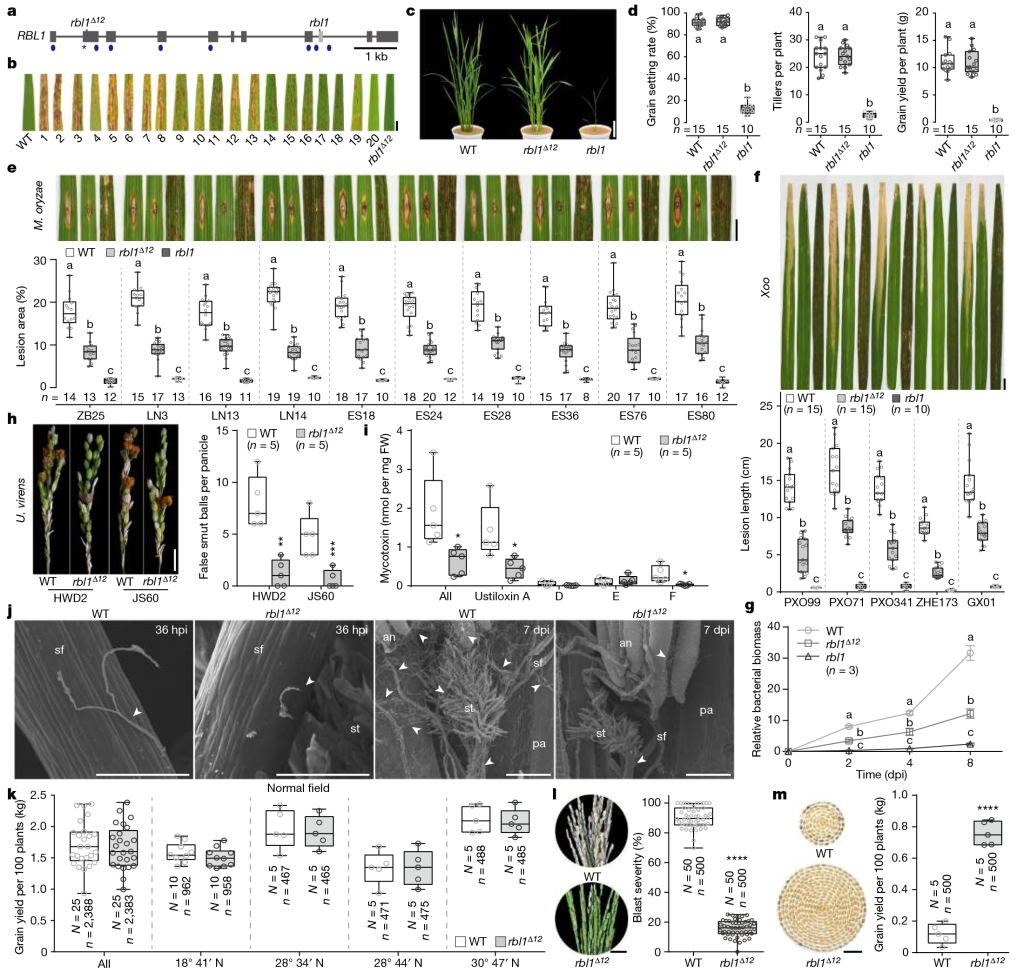
rbl1类病斑突变体虽然具有广谱抗病能力但几乎“绝收”(图4),所以这个基因在育种上非常“不好用”。作者通过对RBL1的基因编码区进行多重基因组编辑(图5),靶向其多个位点,在后代中,发现RBL1Δ12株系只在孕穗期表现出微弱的类病斑表型,其显示出对不同地区分离的10个稻瘟菌、5个白叶枯菌、2个稻曲菌生理小种的抗性,同时,该株系结实正常(图6)。
图4 rbl1突变体穗、种子的表型 (Sha et al., 2023)。
图5 对RBL1的基因组编辑及大田实验 (Sha et al., 2023)。(a)RBL1的基因结构及gRNA的对应位置;(b)使用的编辑载体的图谱;(c)设计的sgRNA的序列;(d)混转示意图;(e)大田实验规划;(f)稻瘟病病圃实验规划;(g)对RBL1Δ12T1代无转基因成分株系的鉴定;(h)比较RBL1Δ12和野生型Kitaake的农艺性状,包括株高、分蘖数、穗数、结实率、千粒重。结果显示,RBL1Δ12株系除了在株高上有明显下降,其余性状与野生型无显著差异。
图6 RBL1Δ12株系不仅拥有广谱抗病能力,其大田产量也并未受到影响 (Sha et al., 2023)。(a)RBL1的基因结构、gRNA的对应位置、RBL1Δ12及rbl1突变体的突变位置;(b)RBL1的编辑株系;(c)WT、RBL1Δ12和rbl1在温室生长的表型比较;(d)WT、RBL1Δ12和rbl1的农艺性状比较;(e)不同稻瘟菌生理小种感染不同株系14天后病斑面积的测定;(f)不同白叶枯菌生理小种感染不同株系14天后病斑长度的测定;(g)白叶枯菌PXO99感染不同株系后的生长情况;(h)感染不同稻曲菌生理小种的穗;(i)分析(h)材料中稻曲菌素的含量;(j)感染稻曲菌的颖花;(k)正常大田环境下,WT与RBL1Δ12的产量比较;(l、m)在稻瘟菌病圃条件下,测试不同株系的稻瘟病抗性及产量。
简而言之,抗病基因通常是个“偏科生”,即抗病的同时带来产量下降,但RBL1Δ12却是个“全能王”,平衡了抗病和产量的关系。
作者也在文章中指出,RBL1基因在短柄草、小麦、大麦、高粱、玉米等物种中高度保守(图7)。这也启示我们可以试试在这些物种中对RBL1进行基因编辑以期创制出新种质,小远认为,其编码区和非编码区都可以试试的。
2022年12月,中国农业科学院蔬菜花卉研究所杨学勇团队在Nature Plants上发表了题为“Architecture design of cucurbit crops for enhanced productivity by a natural allele”的研究论文,作者通过筛选,在几千份瓜类种质中找到了由显性单基因控制的南瓜矮化种质SXHD82,经过图位克隆和遗传验证发现,该品种在南瓜矮化基因CmoYABBY1的5’UTR区缺失了76bp,在这76bp中有一个在葫芦科作物中保守的长度为51bp的B区,对B区的突变不会增加基因转录量但会增加YABBY1蛋白的翻译量,同时YABBY1的蛋白水平与茎长负相关,不同的缺失形式可使南瓜生长出不同的主茎长度(图8),而且,在黄瓜和西瓜中对YABBY1基因进行微调也可实现对主茎长度的控制(图9)。
图8 使用CRISPR/Cas9技术靶向CmoYABBY1的5’UTR B区可产生一系列茎长减少的转基因南瓜 (Wang et al., 2022)。(a)CmoYABBY1的5’UTR中76bp的缺失、A区的缺失、B区的缺失都不会影响基因的转录,但76bp的缺失使蛋白翻译效率升高、A区的缺失不影响蛋白翻译效率、B区的缺失也会使蛋白翻译效率升高;(b)对B区设计编辑靶点,并展示了四个株系的编辑类型;(c、d)野生型和四个编辑株系的表型对比;(e)茎长数据比较;(f)叶片形成簇的频率数据比较;(g)CmoYABBY1基因的相对表达量比较;(h)CmoYABBY1的翻译效率比较。
WOX基因是一类非常重要的基因家族,其通过调控分生组织干细胞的产生和成熟来控制植物的生长发育。在不同物种中,WOX8、WOX9基因似乎表现出基因功能分化:拟南芥中的wox8 wox9、wox9均表现出胚发育缺陷;番茄在WOX8/9基因的自然变异和多个突变体产生花序的过度分枝;在番茄近亲辣椒、矮牵牛中,wox8/9突变体产生的是茎分生组织增生且植株几乎不产生花;水稻中wox8/9突变体的侧枝分生组织增生、节间缩短且影响了花序分枝。
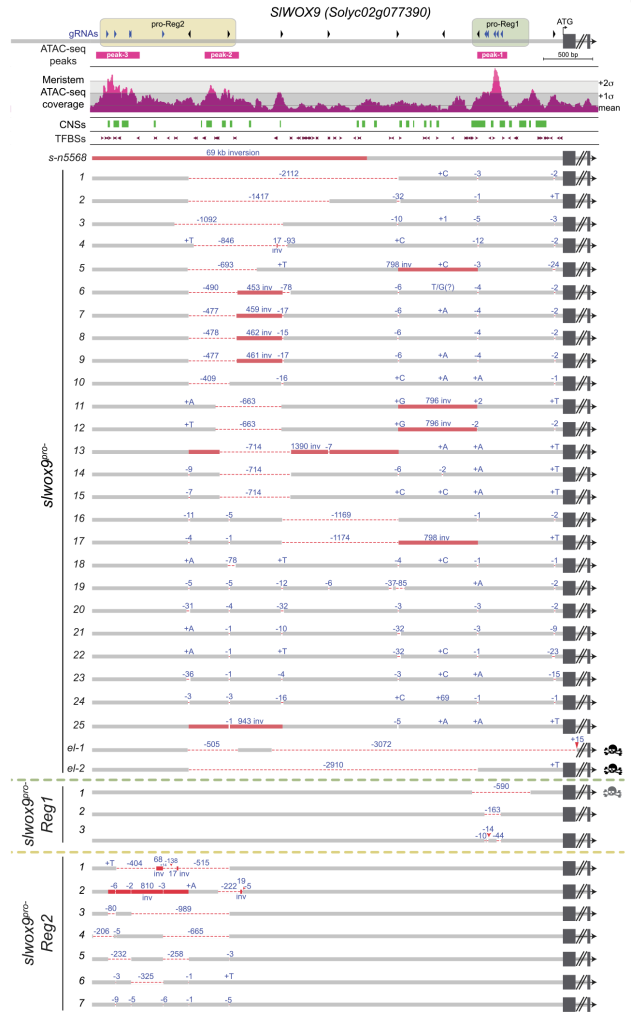
2021年,冷泉港实验室的Zachary B. Lippman和Idan Efroni团队在Cell杂志上发表了题为“Conserved pleiotropy of an ancient plant homeobox gene uncovered by cis-regulatory dissection”的研究论文,作者通过开放染色质检测技术(ATAC-seq)+CRISPR/Cas9介导的启动子编辑系统(图10)揭示了WOX9基因具有多效性,并且其在茄科甚至十字花科中的功能是保守的,而并非之前所认为的WOX9同源基因在进化过程中发生了功能分化。
关于WOX9的有趣故事可见文章“17年前发表过的基因如今照样发Cell”。这个例子告诉我们,如果一个基因具有多效性,编码其启动子的不同区域,可能得到完全不同的性状。
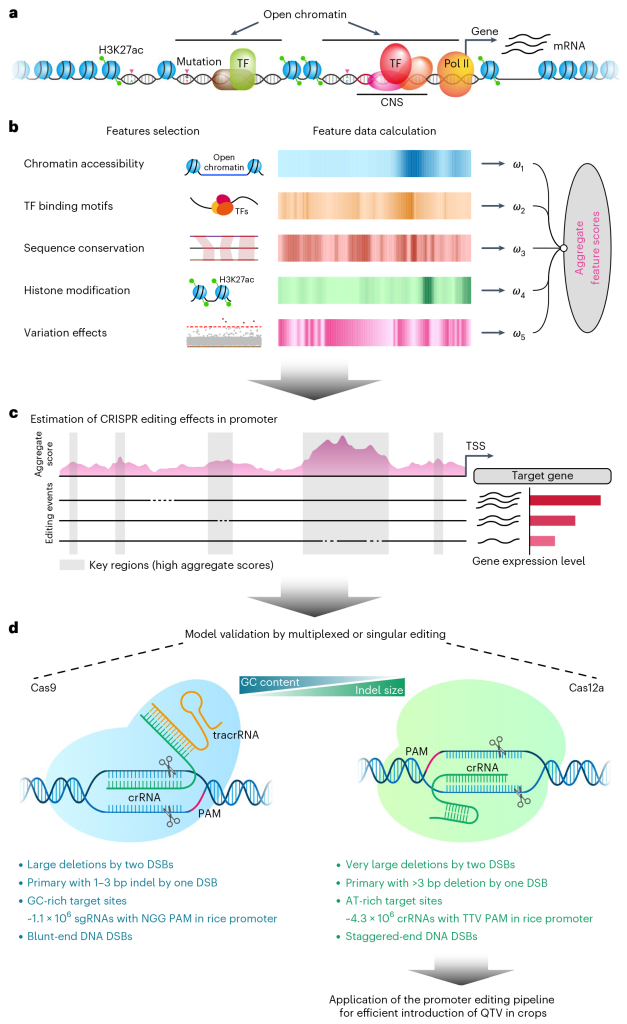
图11 使用不同的CRISPR/Cas工具和编辑策略来编辑植物基因的启动子 (Tang et al., 2023)。(a)单gRNA介导的启动子编辑,可以产生小的Indels、多核苷酸缺失或大的序列缺失,这是编辑启动子最简单的方法;(b)多gRNA介导的启动子编辑,可以产生多个位点的Indels、大的序列缺失或混合的编辑事件,产生的基因型是较为复杂的;(c)同源模板的定向修复,外源供体DNA可用于修复启动子区域的双链断裂,使启动子产生特定的变化;(d)可调控基因表达的Cas调控因子,将无催化活性的dCas9和转录效应因子融合,通过结合启动子区特异的DNA序列来控制基因的表达;(e)表观遗传编辑,将dCas9和表观效应因子例如DNA甲基转移酶或去甲基化酶融合,选择性地改变启动子区的DNA甲基化水平,进而调节基因表达;(f)碱基编辑,由一个脱氨酶和nCas9融合而成,可以在启动子区获得单个或多个核苷酸的替换;(g)先导编辑(PE),该策略将nCas9、反转录酶和带有特定RT模板的pegRNA融合,可实现启动子区核苷酸的替换、插入、缺失或以上情况的组合。
2023年3月,电子科技大学张勇、扬州大学张韬和马里兰大学戚益平团队合作在Nature Plants上发表了题为“An efficient CRISPR-Cas12a promoter editing system for crop improvement”的研究论文,作者使用基于多组学数据的模型预测来进行启动子靶向编辑(简称CAPE系统,图12),利用CAPE系统成功靶向OsGBSSI、OsGS3、OsD18基因的启动子,并分别获得了含不同淀粉含量的连续数量性状变异(QTV)材料集合(图13)、含不同籽粒大小的QTV材料集合、半矮秆QTV材料集合,其中,靶向OsD18启动子的材料中,得到了一个绿色革命数量性状,其产量和抗倒伏性状与sd1突变体无显著差异 (Zhou et al., 2023)。
图12 CAPE系统的设计 (Zhou et al., 2023)。(a)启动子区域内的染色质结构精确地调控了基因的表达;(b)利用基因组的特征来建立预估模型,选取基因组的五个特征——染色质可及性、转录因子结合位点、序列保守性、H3K27ac组蛋白修饰、变异效应作为输入数据,预估模型为启动子区域内的每个特征分配分数,然后将所有的特征分数与各自的权重聚和在一起,以确定调控区域对基因表达的假定影响;(c)基于CPISPR的启动子编辑对靶基因表达的影响,启动子区域的得分用粉色区域表示,预测的重要区域用灰色区域表示;(d)比较CRISPR9和CRISPR12a系统的特征,后者更适合作为启动子编辑工具。
备注:OsGBSS1基因即为前文提到过的Wx基因。
本文所说的“微调”其实是相比于之前大家常做的基因编辑只产生“功能缺失”的效果而言的,读完全文,让我们来想想,如何做“微调”呢?“微调”能产生哪些效果呢?
通过编辑基因的非编码区,可能像Wx基因、YABBY1基因那样得到不同数量性状的新种质资源,也可能像IPA1那样得到基于相同位点的全新性状,还可能像WOX9基因那样得到基于相同位点的完全不同的性状;通过编辑基因的编码区,也可能像RBL1基因那样得到基于相同位点的全新性状。最新发表的一些文章也给我们提供了新的解决方案。这些例子都为我们精确改良或创造目标农艺性状,甚至从头创制基于定向进化的全新植物提供了新思路。当然,更多的例子等你来分享给大家喔!
Hendelman A, Zebell S, Rodriguez-Leal D, et al. Conserved pleiotropy of an ancient plant homeobox gene uncovered by cis-regulatory dissection. Cell. 2021;184(7):1724-1739.e16. doi:10.1016/j.cell.2021.02.001
Huang L, Sreenivasulu N, Liu Q. Waxy Editing: Old Meets New. Trends Plant Sci. 2020;25(10):963-966. doi:10.1016/j.tplants.2020.07.009
Huang L, Li Q, Zhang C, et al. Creating novel Wx alleles with fine-tuned amylose levels and improved grain quality in rice by promoter editing using CRISPR/Cas9 system. Plant Biotechnol J. 2020;18(11):2164-2166. doi:10.1111/pbi.13391
Jiao Y, Wang Y, Xue D, et al. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice. Nat Genet. 2010;42(6):541-544. doi:10.1038/ng.591
Sha G, Sun P, Kong X, et al. Genome editing of a rice CDP-DAG synthase confers multipathogen resistance [published online ahead of print, 2023 Jun 14]. Nature. 2023;10.1038/s41586-023-06205-2. doi:10.1038/s41586-023-06205-2
Song X, Meng X, Guo H, et al. Targeting a gene regulatory element enhances rice grain yield by decoupling panicle number and size. Nat Biotechnol. 2022;40(9):1403-1411. doi:10.1038/s41587-022-01281-7
Tang X, Zhang Y. Beyond knockouts: fine-tuning regulation of gene expression in plants with CRISPR-Cas-based promoter editing [published online ahead of print, 2023 Jun 6]. New Phytol. 2023;10.1111/nph.19020. doi:10.1111/nph.19020
Tian Z, Qian Q, Liu Q, et al. Allelic diversities in rice starch biosynthesis lead to a diverse array of rice eating and cooking qualities. Proc Natl Acad Sci U S A. 2009;106(51):21760-21765. doi:10.1073/pnas.0912396106
Wang S, Wang K, Li Z, et al. Architecture design of cucurbit crops for enhanced productivity by a natural allele. Nat Plants. 2022;8(12):1394-1407. doi:10.1038/s41477-022-01297-6
Xu Y, Lin Q, Li X, et al. Fine-tuning the amylose content of rice by precise base editing of the Wx gene. Plant Biotechnol J. 2021;19(1):11-13. doi:10.1111/pbi.13433
Zeng D, Liu T, Ma X, et al. Quantitative regulation of Waxy expression by CRISPR/Cas9-based promoter and 5’UTR-intron editing improves grain quality in rice. Plant Biotechnol J. 2020;18(12):2385-2387. doi:10.1111/pbi.13427
Zhang J, Zhang H, Botella JR, Zhu JK. Generation of new glutinous rice by CRISPR/Cas9-targeted mutagenesis of the Waxy gene in elite rice varieties. J Integr Plant Biol. 2018;60(5):369-375. doi:10.1111/jipb.12620
Zhang C, Zhu J, Chen S, et al.Wxlv, the Ancestral Allele of Rice Waxy Gene. Mol Plant. 2019;12(8):1157-1166. doi:10.1016/j.molp.2019.05.011
Zhang L, Yu H, Ma B, et al. A natural tandem array alleviates epigenetic repression of IPA1 and leads to superior yielding rice. Nat Commun. 2017;8:14789. Published 2017 Mar 20. doi:10.1038/ncomms14789
Zhou J, Liu G, Zhao Y, et al. An efficient CRISPR-Cas12a promoter editing system for crop improvement. Nat Plants. 2023;9(4):588-604. doi:10.1038/s41477-023-01384-2