近日来自马萨诸塞大学的Scot A. Wolfe教授课题组在Nature Methods上发表了题为“Genome-wide profiling of prime editor off-target sites in vitro and in vivo using PE-tag”的研究论文,报道了一种用于识别潜在的引导编辑器脱靶位点的全基因组方法,称为PE-tag。该方法使用Prime编辑器和靶向目标位点的 pegRNA,以促进逆转录酶介导的扩增标签添加。PE 标签在体外对纯化的基因组 DNA 执行最有效;然而,它也可以在细胞培养物中或在递送合适的试剂时在体内进行。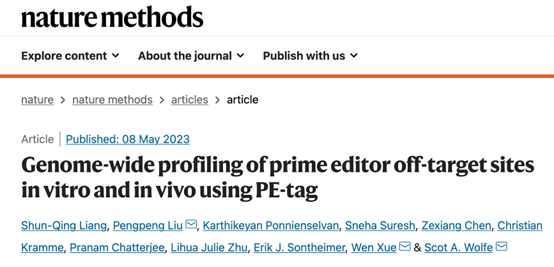
为了确定给定目标位点在整个基因组中的编辑结果,作者设计了Prime editing guide RNA (pegRNA),以在引导编辑器编辑的基因组位点附加扩增标签。然后用携带寡核苷酸的定制 Tn5 转座酶标记经引导编辑器处理的基因组 DNA,该寡核苷酸具有独特的分子标识符 (UMI) 和 i5 Illumina 接头序列。该酶促过程通过半随机插入转座酶携带的 DNA 接头序列将基因组 DNA 分解成小片段。使用标签特异性引物和 i5 引物的 PCR 允许选择性扩增和基于 Illumina 测序的基因组区域鉴定,这些区域已被引导编辑器修改。作者将这个系统命名为 PE-tag(图 1a)。
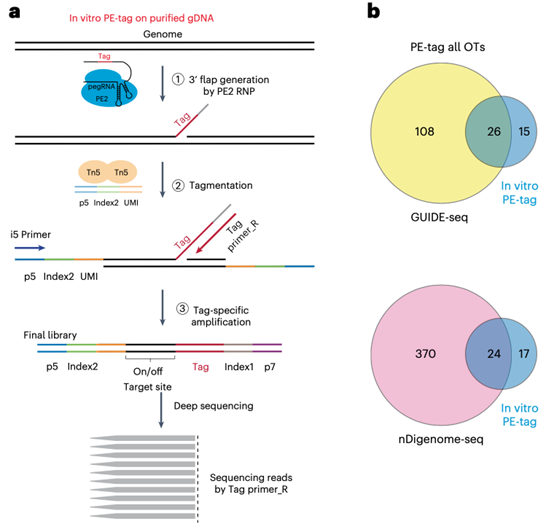
图1. 体外 PE 标签的示意图概述
作者比较了通过 PE 标签与 GUIDE-seq3 识别 prime editor 脱靶位点的假阳性和假阴性率。GUIDE-seq 是一种基于 Cas9 核酸酶的稳健方法,用于通过 DNA 修复介导的共同递送的双链寡核苷酸插入来识别基因组内的核酸酶切割位点,该寡核苷酸标记这些位点以进行选择性扩增。作者发现,在评估每种方法指定的潜在主要编辑器脱靶位点时,PE 标签的假阳性率低于 GUIDE-seq(图 1b)。PE 标签在体外对纯化的基因组 DNA 或细胞培养物中通过主要编辑试剂的瞬时表达进行捕获脱靶位点时最为有效。重要的是,他们发现 prime editor的活性脱靶位点更少——因此比被编程为编辑同一目标位点的 Cas9 核酸酶更精确。
总的来说,PE-tag 可以在各种目标位点(包括具有治疗应用的位点)为引导编辑器识别脱靶位点。作者相信,这种多功能的脱靶分析方法为评估各种引导编辑平台的基因组特异性提供了一种重要的方法。文章来源:植物生物技术Pbj




