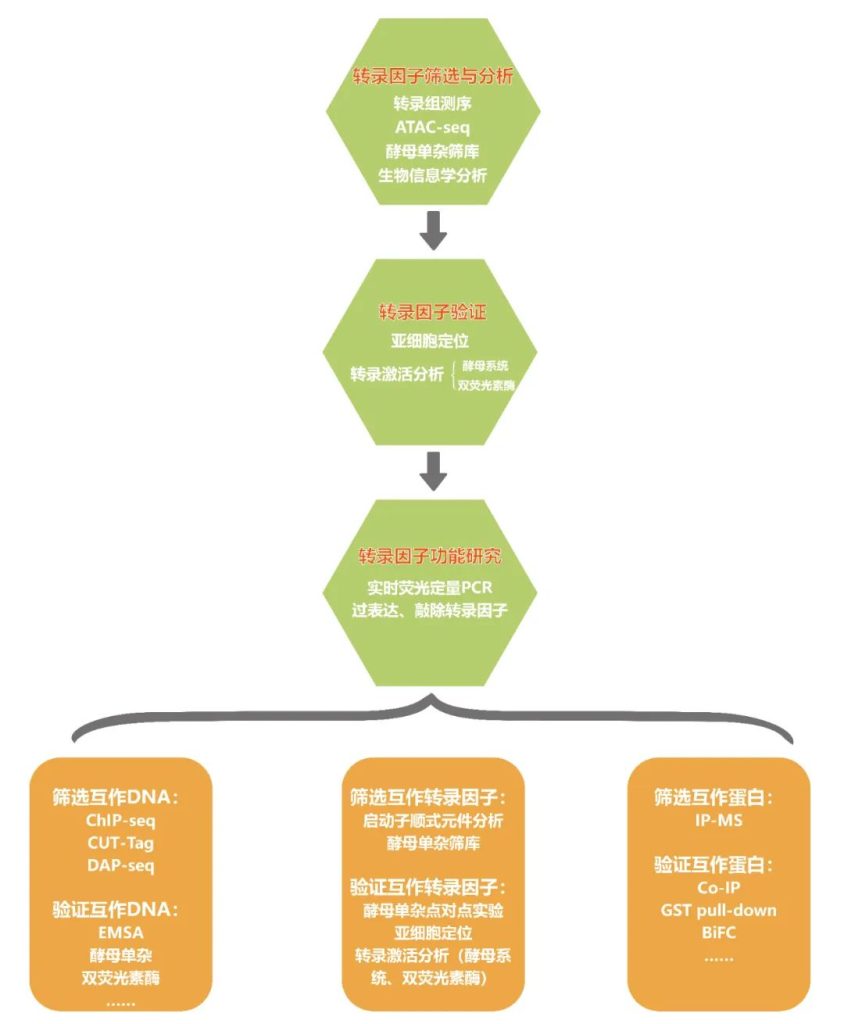
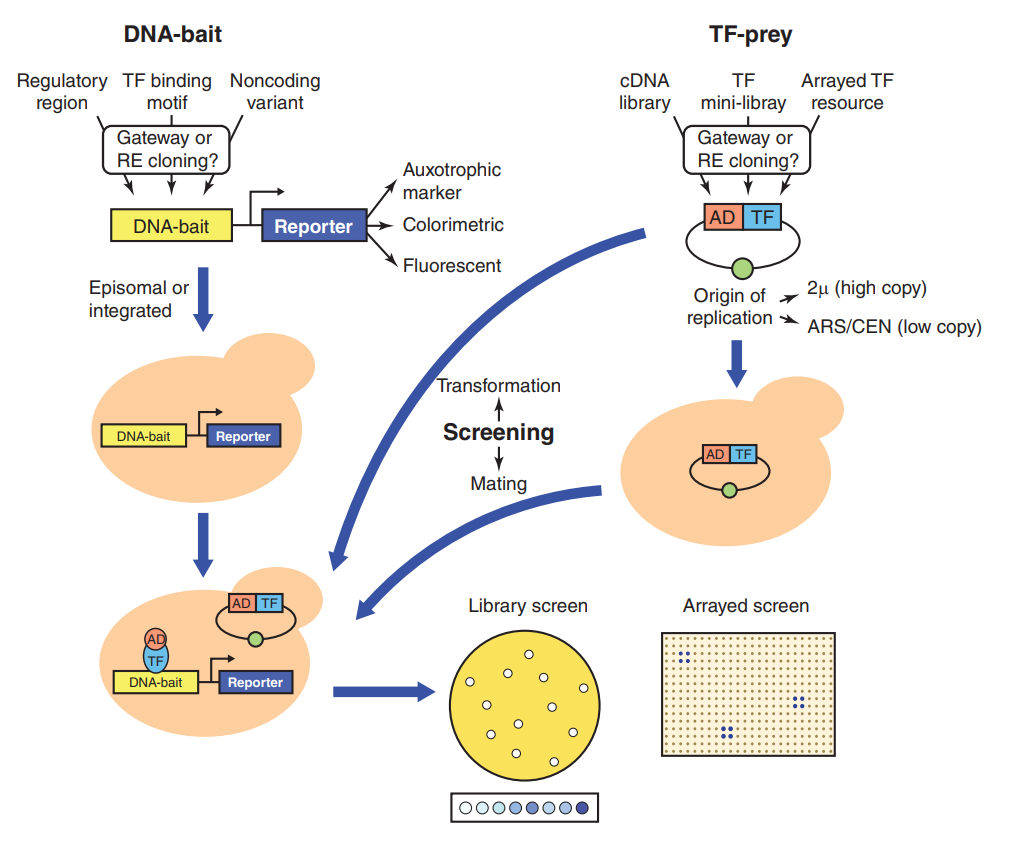
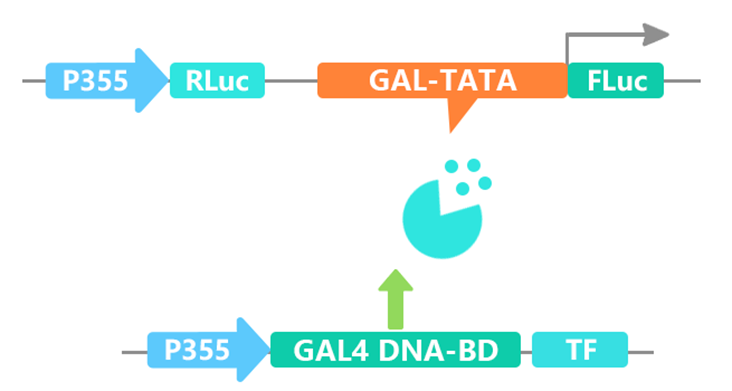
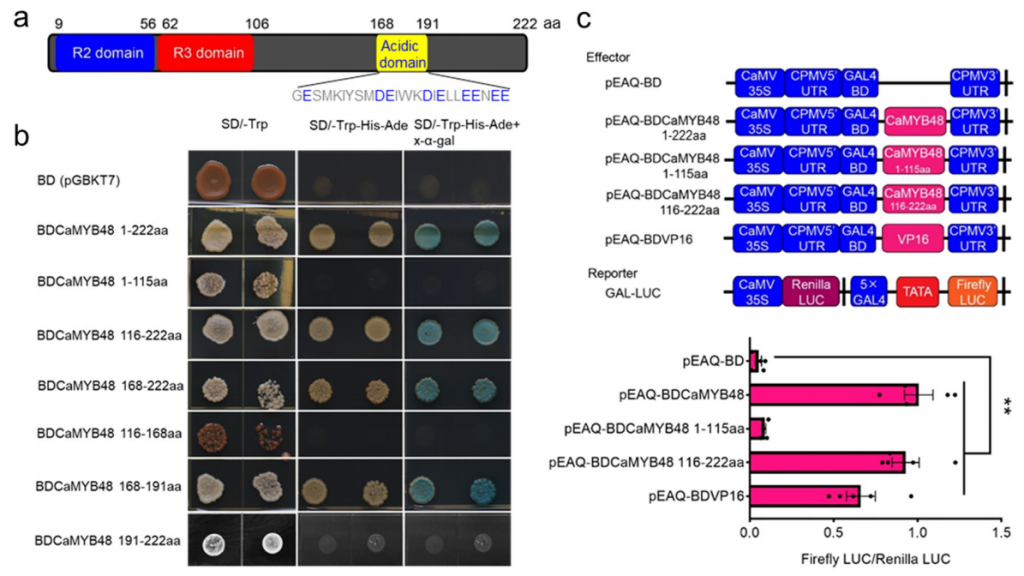
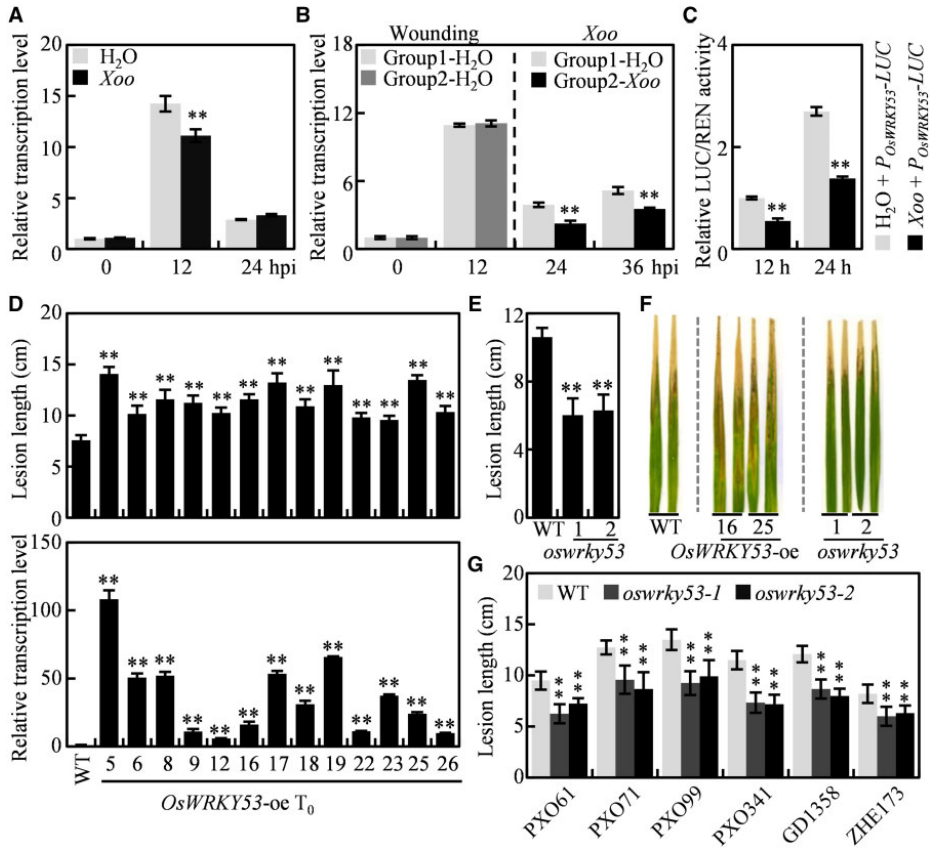
在先前的推文中,小远发现大家对转录因子相关的文章比较感兴趣,因此猜测应该很多人都在做这方面的研究,为了更好的帮助大家开展转录因子的研究,本次推文主要是和大家一起来复盘一下转录因子的常规研究思路及方法,内容可以归纳为如下:
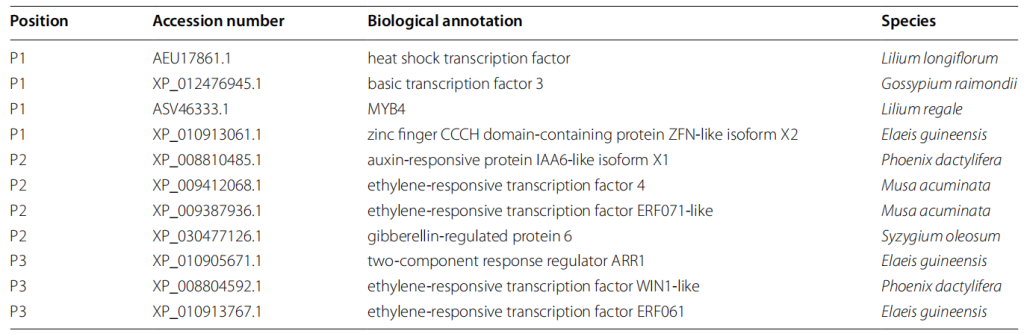
表1 转录因子筛选结果(Cao et al., 2022)。
(1)蛋白功能结构域预测:MEME(http://meme-suite.org/tools/meme);
(2)信号肽序列分析网站:SignaIP(http://www.cbs.dtu.dk/services/SignalP/);
(3)蛋白的糖基化位点、磷酸化位点和蛋白激酶结合位点预测网站:NetPhos 3.1 Server(http://www.cbs.dtu.dk/services/NetNGlyc/);
(4)蛋白亲/疏水性分析网站:
ExPasy-ProtScale(https://web.expasy.org/protscale/);
(5)蛋白质二级结构预测网站:
SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)。
表2 蛋白质亚细胞定位的部分预测软件和网站(Donnes and Hoglund, 2004)。
Cao Y, Bi M, Yang P, et al. Construction of yeast one-hybrid library and screening of transcription factors regulating LhMYBSPLATTER expression in Asiatic hybrid lilies (Lilium spp.). BMC Plant Biol. 2021, 21(1): 563.
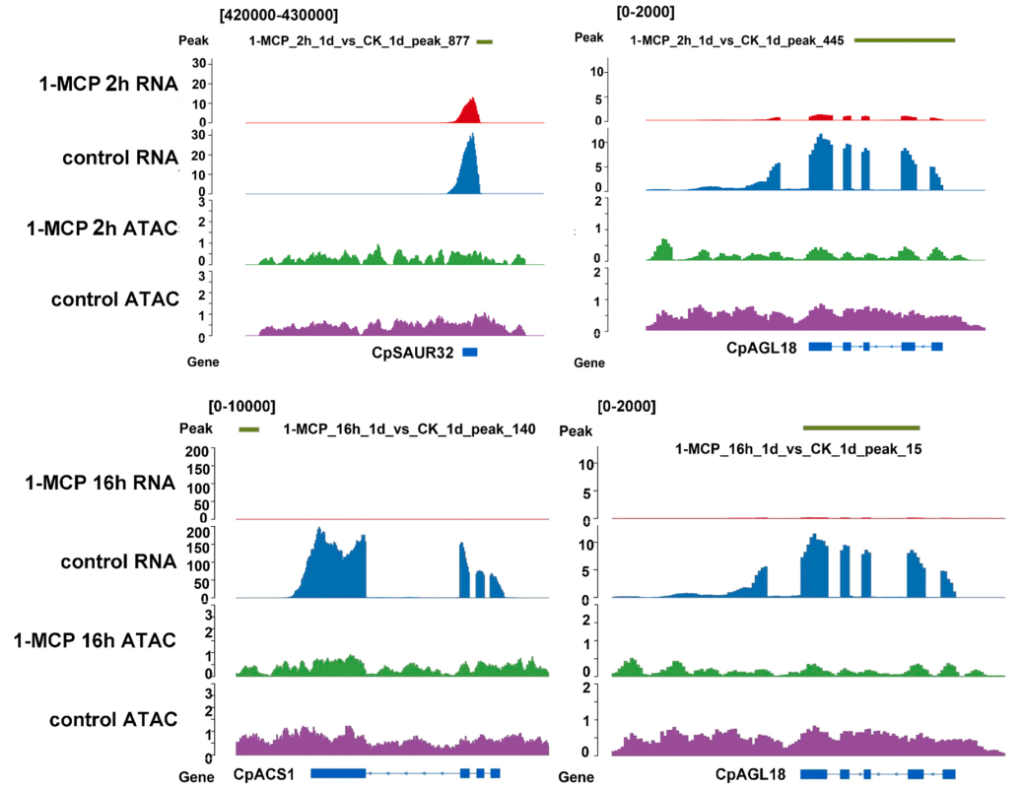
Cai J, Wu Z, Song Z, et al. ATAC-seq and RNA-seq reveal the role of AGL18 in regulating fruit ripening via ethylene-auxin crosstalk in papaya. Postharvest Biology and Technology. 2022, 191.
Dönnes P, Höglund A. Predicting protein subcellular localization: past, present, and future. Genomics, proteomics & bioinformatics, 2004, 2(4): 209-215.
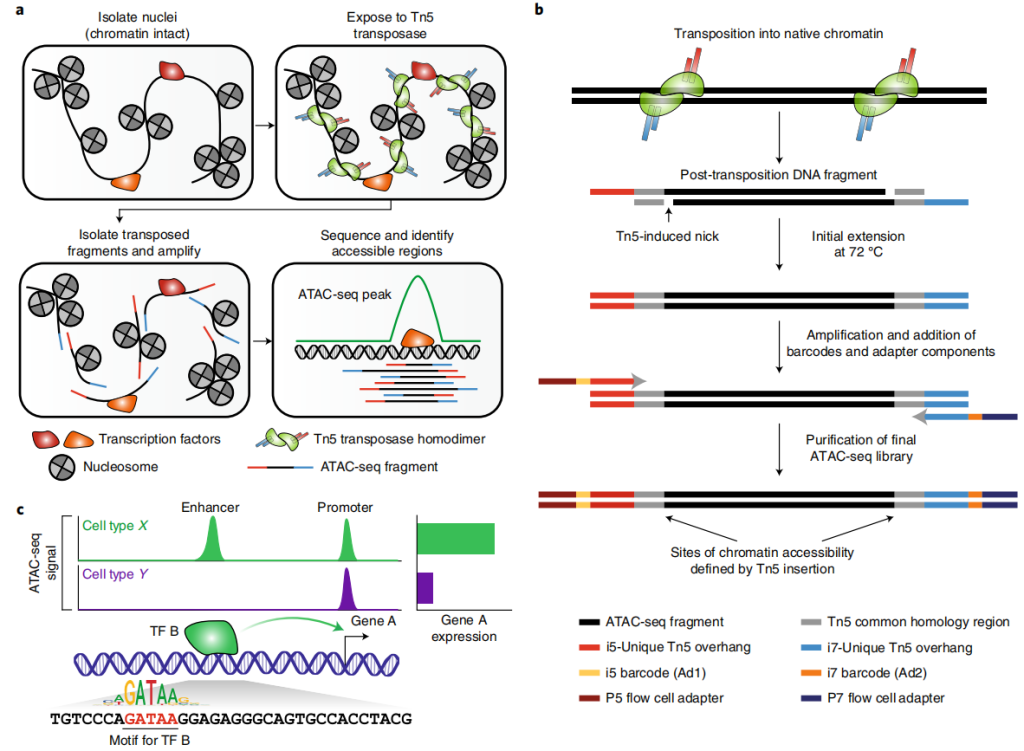
Grandi FC, Modi H, Kampman L, et al. Chromatin accessibility profiling by ATAC-seq. Nat Protoc. 2022, 17(6): 1518-1552.
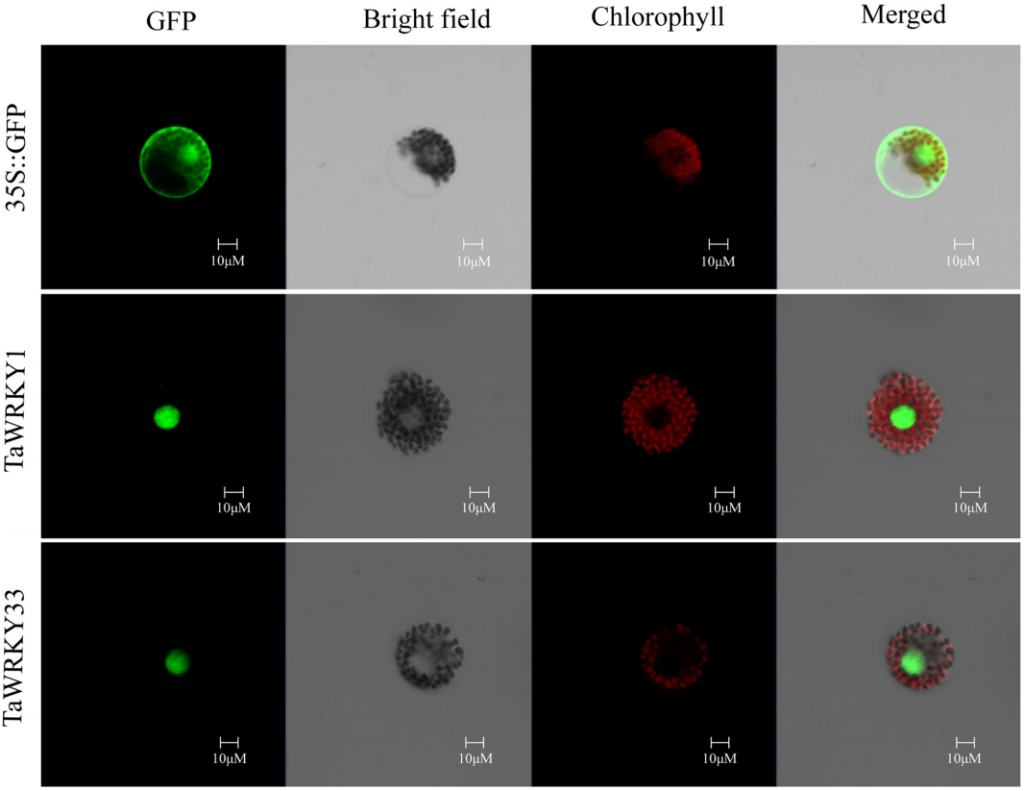
He GH, Xu JY, Wang YX, et al. Drought-responsive WRKY transcription factor genes TaWRKY1 and TaWRKY33 from wheat confer drought and/or heat resistance in Arabidopsis. BMC Plant Biol. 2016, 16(1): 116.
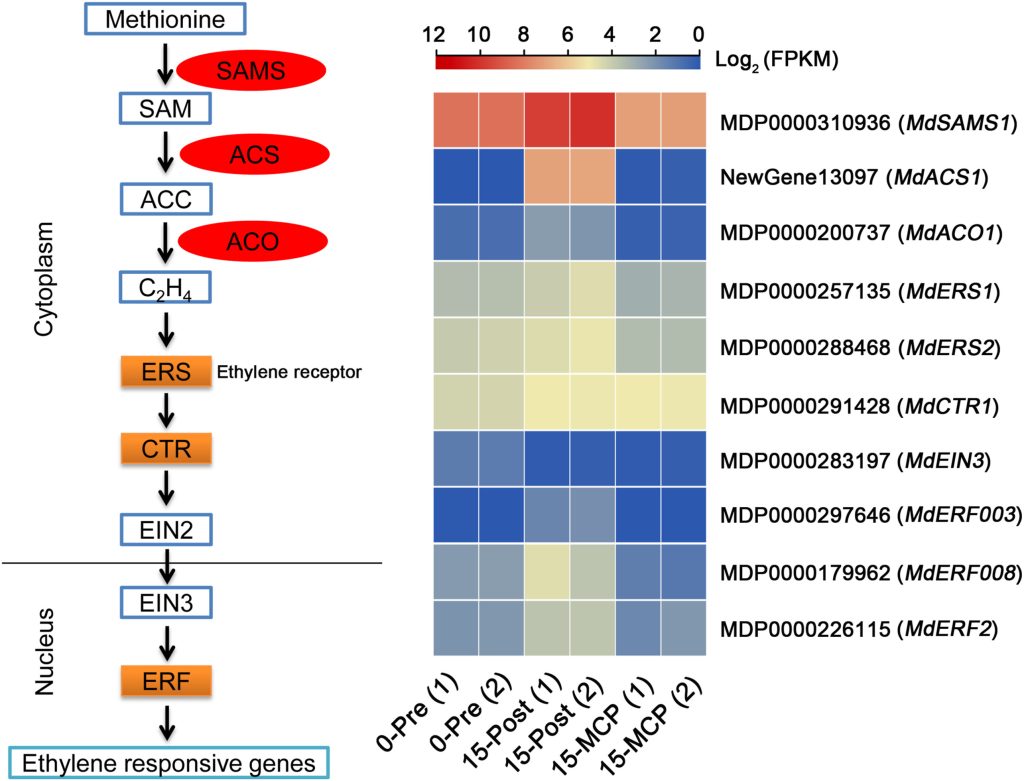
Li T, Zhang X, Wei Y, et al. Comparative Transcriptome Analysis of the Climacteric of Apple fruit Uncovers the Involvement of transcription factors affecting ethylene biosynthesis. Horticultural Plant Journal. 2022.
Sewell JA, Fuxman Bass JI. Options and considerations when using a yeast one-hybrid system[M]//Two-Hybrid Systems. Humana Press, New York, NY, 2018: 119-130.
Sun B, Zhou X, Chen C, et al. Coexpression network analysis reveals an MYB transcriptional activator involved in capsaicinoid biosynthesis in hot peppers. Hortic Res. 2020, 7(1): 162.
Trofimov K, Ivanov R, Eutebach M, et al. Mobility and localization of the iron deficiency-induced transcription factor bHLH039 change in the presence of FIT. Plant Direct. 2019, 3(12): e00190.
Xie W, Ke Y, Cao J, et al. Knock out of transcription factor WRKY53 thickens sclerenchyma cell walls, confers bacterial blight resistance. Plant Physiol. 2021, 187(3): 1746-1761.
Zhou R, Dong Y, Liu X, et al. JrWRKY21 interacts with JrPTI5L to activate the expression of JrPR5L for resistance to Colletotrichum gloeosporioides in walnut. Plant J. 2022, 111(4): 1152-1166.