近日,浙江省农业科学院徐盛春研究员团队开发了生物素标记的Tn5转座酶介导的CUT&Tag (B-CUT&Tag)方法,并成功应用于植物转录因子与DNA互作研究。该技术首次报道了利用CUT&Tag在植物中鉴定转录因子结合位点,提供的B-CUT&Tag流程可以不受起始材料用量和转录因子含量水平的限制,可广泛应用于植物和动物转录因子研究。相关研究成果在线发表在植物学知名期刊《Plant Biotechnology Journal》上。

绘制转录因子与染色质互作图谱是ENCODE计划的重要部分,对理解基因表达调控具有重要意义。CUT&Tag技术于2019年由Henikoff实验室首次开发,用于研究全基因组范围内特定的染色质修饰状态(如组蛋白修饰)及特定蛋白(如转录因子)DNA靶位点的研究,其功能等同于染色质免疫共沉淀 (ChIP)。与ChIP相比,CUT&Tag不需要依赖昂贵的非接触式超声破碎仪打断染色质,需要的起始材料少,在实验操作和NGS建库流程上更简洁,产生的数据信噪比高,重复性强。后续基于CUT&Tag进一步开发的单细胞CUT&Tag(sc-CUT&Tag)能够在动物组织中进行基于表观修饰状态的细胞分型,大大加深了人们对特定发育过程中单细胞表观遗传状态的认识。
然而,相较于大量存在的组蛋白修饰,转录因子由于含量相对少,或者其靶基因相对少等原因,利用常规CUT&Tag流程就特定转录因子-DNA互作研究仍然是一个挑战。目前,仅有在一些动物细胞中有少数的利用CUT&Tag进行转录因子结合位点的研究的成功例子,在植物中仍未见报道。因此,建立一个有效的可以用于植物转录因子-DNA互作的改进版CUT&Tag方法用于植物特别是作物的ENCODE研究,具有极重要的价值。
徐盛春团队利用生物素修饰的接头进行转座酶(pA/G-Tn5)的包埋,形成带生物素标记的转座子介导靶基因位点的切割及加接头(tagmentation)反应,称之为Biotinylated Tn5 Transposase-mediated CUT&Tag(B-CUT&Tag)。B-CUT&Tag在tagmentation反应后续使用生物素-链霉亲和素纯化体系去除未被切割的完整染色质(intact chromatin),特异性地纯化被生物素标记的tagmentation产物,有利于后续NGS PCR建库。基于此原理,B-CUT&Tag可以依据被研究的转录因子的含量及其靶基因的丰富程度灵活调整使用的细胞/细胞核数量。
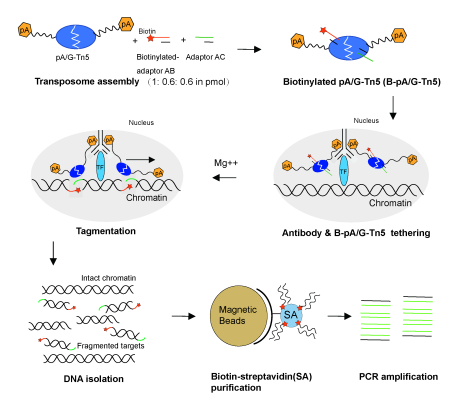
图1 B-CUT&Tag工作原理图
进一步地,文章展示了利用B-CUT&Tag进行拟南芥SPL9(AtSPL9)靶基因研究的具体结果。AtSPL9是植物中被研究得比较充分的转录因子,大量高水平的文章从表型、理化分析、遗传分析等角度充分确立了AtSPL9的靶基因、调控通路及机制,因此,AtSPL9是验证B-CUT&Tag效率的很好案例。AtSPL9 B-CUT&Tag 结果显示,AtSPL9的靶基因涵盖了之前已经明确的包括幼年向成年转化/开花、腺毛发育、类黄酮生物合成、蜡质合成和分枝等相关的靶基因。有趣的是,在基因组水平上整体反应了AtSPL9在调节生长-防御中的权衡(growth-defense trade-offs)。此外,作为ChIP-qPCR类似的功能,建立了CUT&Tag后qPCR的方法(B-CUT&Tag-qPCR),并成功验证了拟南芥SPL9和水稻PHR2与各自靶基因的结合,可作为B-CUT&Tag-seq的有益补充。该研究为分析转录因子-染色质相互作用提供了一种方便高效的CUT&Tag策略,广泛适用于作物改良的顺式调控元件的注释,也对动物中的转录因子研究具有广泛的参考价值。
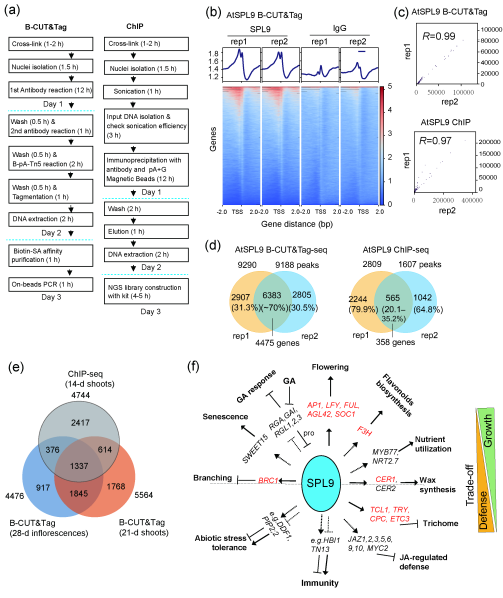
图2 B-CUT&Tag成功应用于拟南芥AtSPL9靶基因的鉴定。
浙江省农业科学院陶晓园副研究员为论文第一作者,浙江省农业科学院、湘湖实验室徐盛春研究员为论文通讯作者,中国科学院植物科学卓越创新中心王佳伟研究员,浙江大学关雪莹教授,浙江省农业科学院洪高洁研究员也参与了此项工作。本研究得到了浙江省重点研发项目、中央引导地方科技发展专项、国家自然科学基金等项目的资助。




