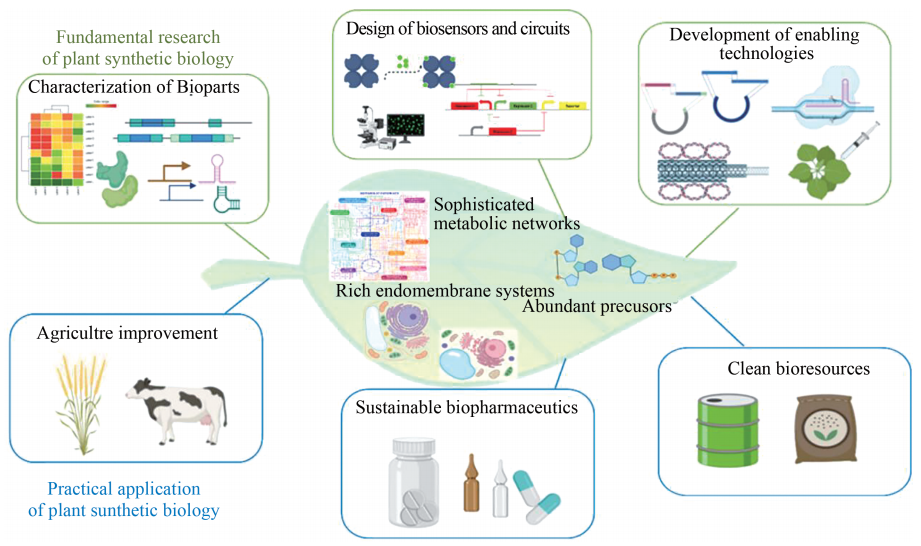
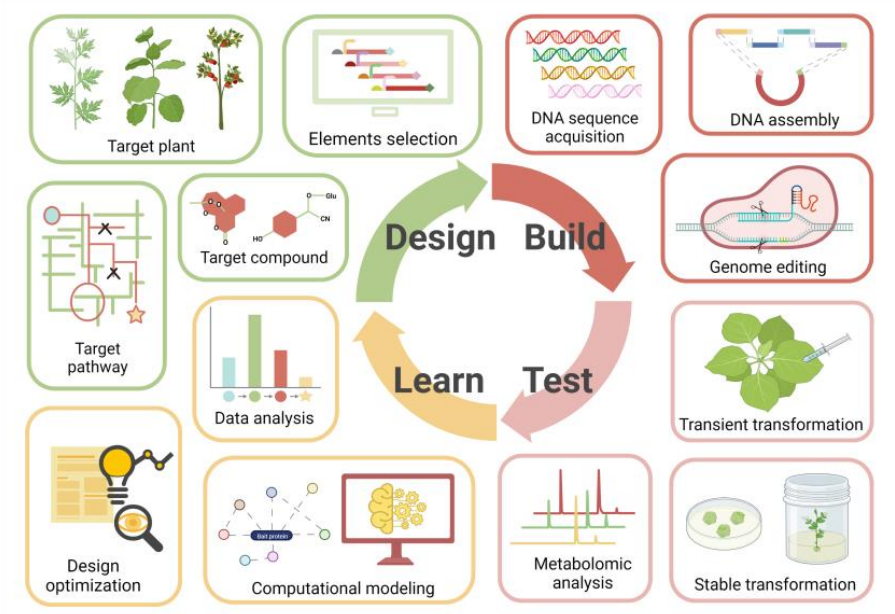
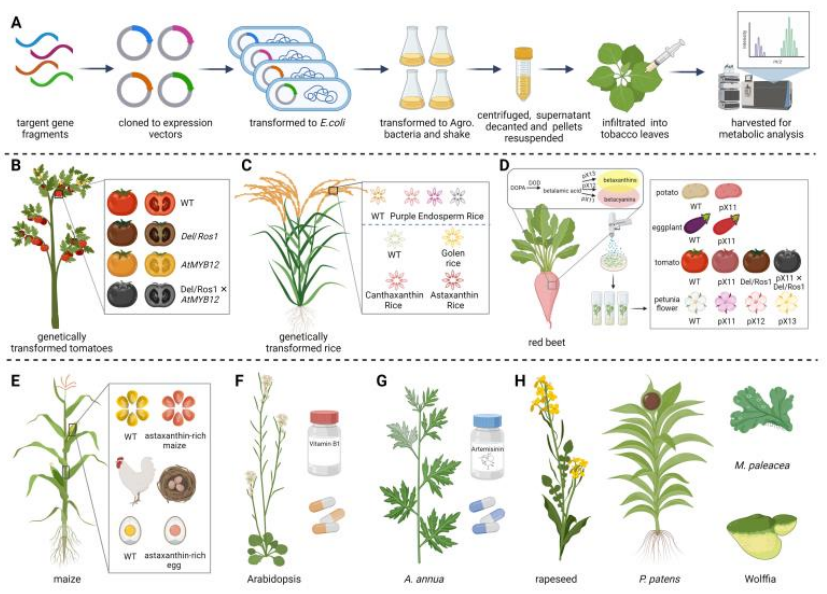
该领域在过去十多年的发展中,从设计生物装置、建立代谢通路到合成基因组,大多是在微生物底盘中进行的。但随着研究的深入,简单的单细胞体系已无法满足发展需求,需要向多细胞的复杂体系过渡。植物拥有丰富的内膜系统和细胞器、高度特化的生物合成基因簇、精细的代谢调控网络,为开展相关研究提供了理想的模式体系。以植物为底盘的合成生物学研究(图1),如设计检测环境变化的植物传感器、开发精准修饰的基因编辑技术、建立高效异源合成代谢途径等,不仅有助于人类加深对复杂生命运行规律的理解,还有望为解决农业生产、生物制药、能源环境等方面的困境与难题提供新策略,实现可持续发展。
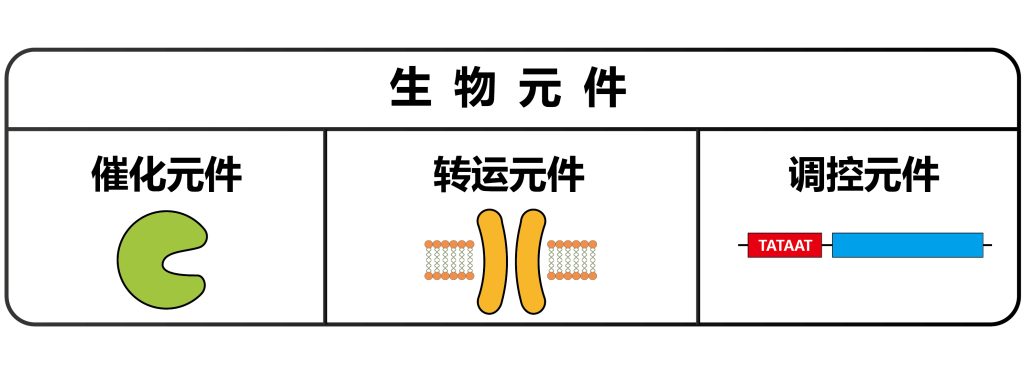
随着研究的深入,越来越多的生物元件被鉴定测试,这也为合成生物学的发展奠定了坚实的基础。例如,Feike等人通过系统分析发现pBdEF1α、p35S、pAtUBI10、pOsUBI3、pZmUBI、pBdUBI10和pOsPGD1这7个启动子和t35S、tNOS、tH4、tAdh和tOCS这5个终止子在单、双子叶植物的叶片和根中都有较高的强度,可用来驱动目的基因表达或用于构建多基因复合性状载体(Feike et al., 2019)。Pérez-González等人对烟草、鸡溶菌酶、拟南芥的基质结合区(MAR, matrix attachment regions)和矮牵牛的转化促进序列(Transformation boost sequence, TBS)进行测试,发现上述4种绝缘子序列均能够增强外源基因表达,并且拟南芥MAR序列还能够减小基因表达的变动幅度(Pérez-González et al., 2019)。此外,通过创建植物特异性启动子,获得转录活性更高的精短型的小核RNA启动子,为植物多基因编辑提供了强有力的工具(Hao et al., 2020)。
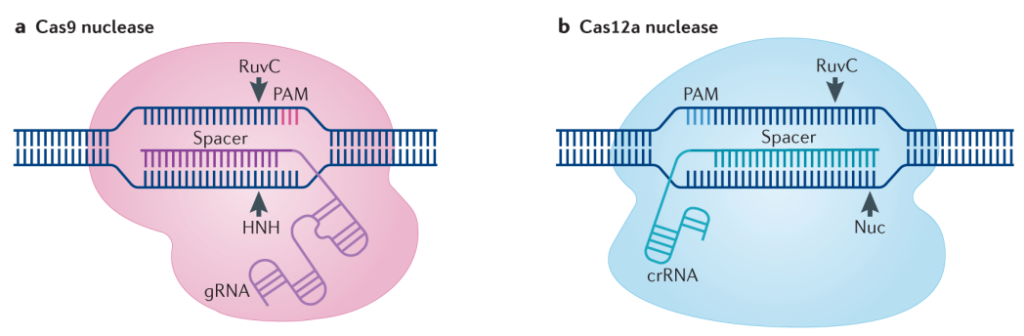
近几年,随着单碱基编辑技术的开发,基因编辑领域进入了一个更为精准的时代,目前常见的胞嘧啶碱基编辑器(Cytidine base editors,CBE)、腺嘌呤碱基编辑器(Adenine base editors,ABE)已在水稻、小麦、番茄、玉米、拟南芥等多个物种中实现了应用(邵洁等, 2020)。另外,为了解决单碱基编辑的脱靶问题,Lin等人建立起了适用于植物任意碱基编辑的技术——植物引导编辑技术(Plant prime editing,PPE),并在水稻和小麦的原生质体中实现12种类型、16个位点的精确编辑(Lin et al., 2020)。新型的饱和靶向内源基因突变碱基编辑器(Saturated targeted endogenous mutagenesis editors,STEME)的成功建立,实现了水稻乙酰辅酶A羧化酶基因的定向进化,从而获得除草剂抗性突变,这也为快速获得有益农艺性状提供了可能(Li et al., 2020)。另外,为了避免外源片段整合到基因组中的风险,科学家们还尝试将Cas蛋白和gRNA在体外组装成核糖核蛋白复合体(Ribonucleoprotein,RNP)进行DNA-free的编辑,并已在14种植物中测试成功(Metje-Sprink et al., 2019)。
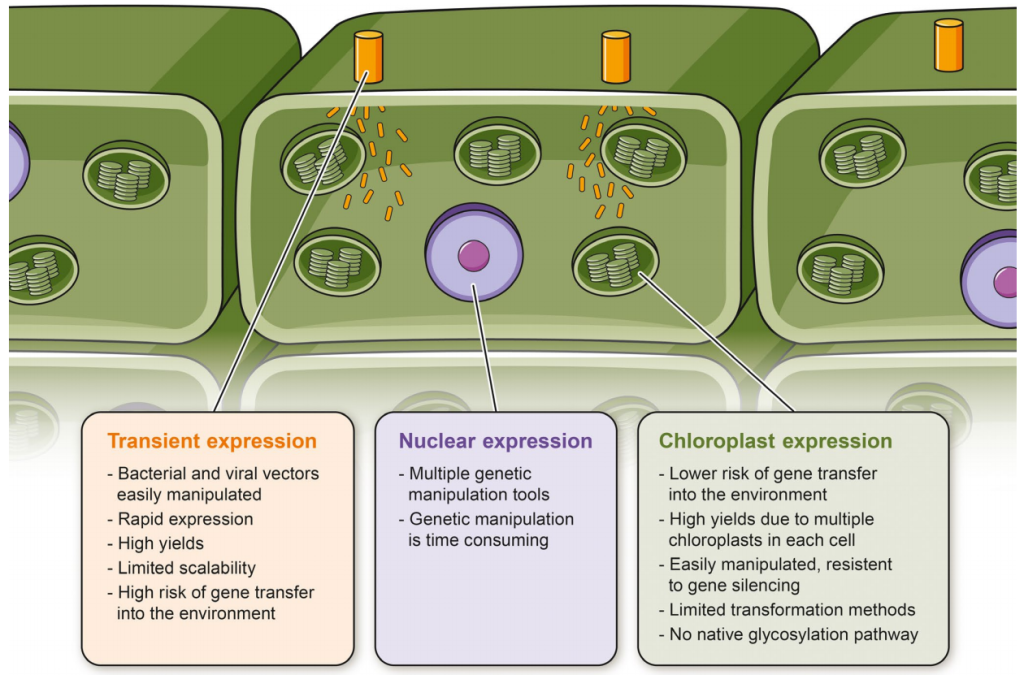
图11 瞬时(黄色)、核(紫色)和叶绿体(绿色)表达系统的简化示意图(Burnett et al., 2020)。
(1)核基因组遗传转化
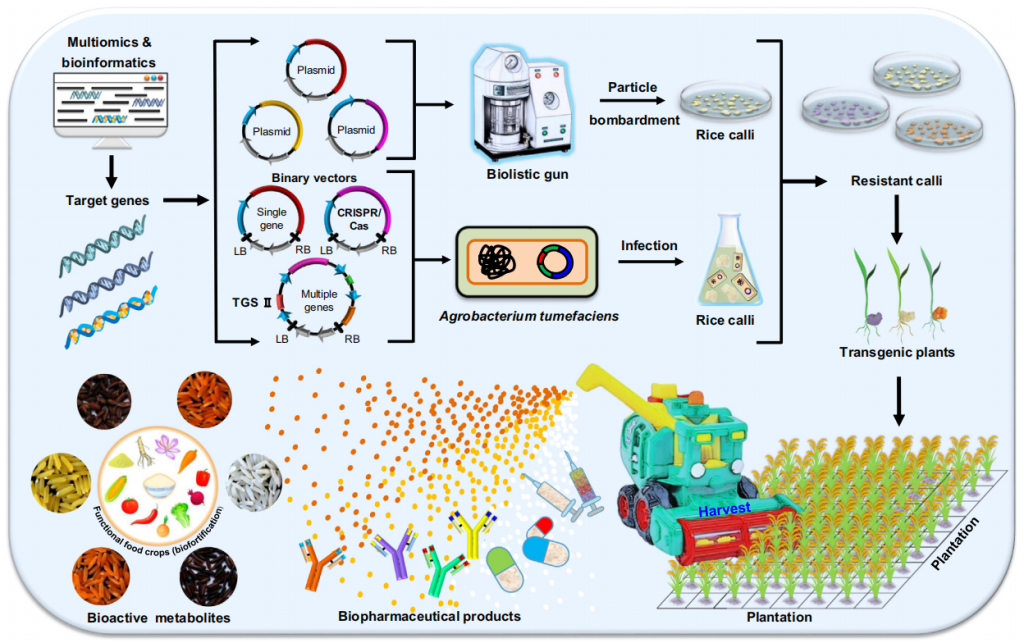
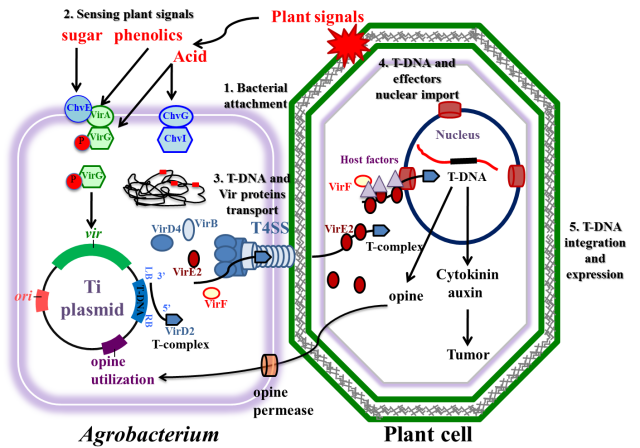
目前常用的核基因组稳定遗传转化手段主要有农杆菌介导法、基因枪法、花粉管通道遗传转化法、显微注射法和最新的纳米颗粒转化法。其中农杆菌介导的遗传转化方法(图12)已成为双子叶植物和单子叶植物(如水稻)中最常用的基因传递技术,但其转化效率受受体植物、基因型、外植体、载体、细菌菌株以及培养基组成等的影响。最新开发的纳米颗粒转化法是将装载外源基因的纳米粒子,通过基因枪技术或借助磁场等转入植物中,从而获得转基因植株。该技术具有穿透性强、装载量大、保护外源基因不被降解以及较短的遗传转化周期等特点。
图12 根癌农杆菌介导的植物转化过程(Hwang et al., 2017)。(1)根癌农杆菌附着于植物细胞;(2)根癌农杆菌感应植物信号后开始调控毒力基因;(3)T-DNA和毒力蛋白的产生和运输,T-复合物从细菌细胞进入植物细胞;(4)T-DNA和效应蛋白进入细胞核;(5)T-DNA在植物基因组中的整合和表达。
(2)瞬时转化
瞬时转化因其操作简单、转化效率高和检测周期短等特点被广泛应用于利用植物底盘生产天然产物。瞬时转化不仅大大缩短了表达系统的开发时间,还可用于测试转基因载体以及快速获得重组蛋白进行功能分析。目前,常用的瞬时转化体系有烟草叶片、愈伤组织、悬浮细胞、原生质体、毛状根等,关于这一部分内容的详细讲解,大家感兴趣的话可以查阅小远的往期文章:没有转化体系的物种,如何研究其基因功能?(二)。
(3)叶绿体遗传转化
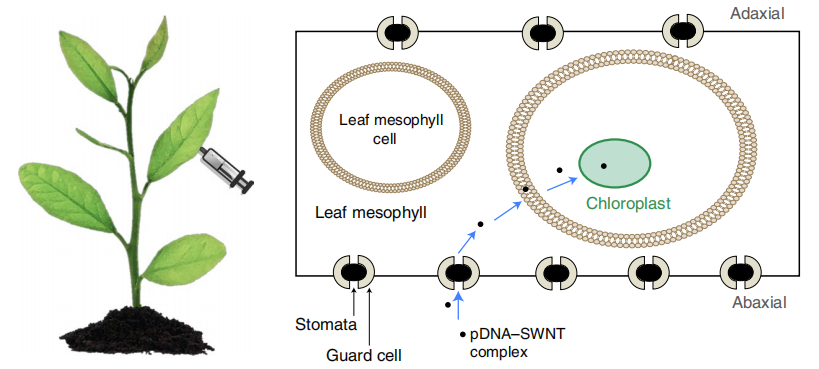
比起细胞核转基因,叶绿体的转基因具有多重优势,主要包括:1、由于缺乏表观遗传或转录后基因沉默机制,蛋白表达水平高且稳定;2、叶绿体DNA通过母系遗传,几乎不存在转基因不良扩散的风险;3、叶绿体中多为原核性质的转录翻译机制,可以运用多顺反子调控的表达策略等(Kuroda et al., 2001; Ruf et al., 2007; Long et al., 2018)。目前叶绿体的遗传转化一般是通过基因枪法或PEG介导的转化将转基因引入叶绿体基因组中。此外,人们还可以利用脂质交换膜渗透(LEEP)技术,借助纳米颗粒将基因在无外力作用的情况下将DNA运输到植物细胞的叶绿体中。进入叶绿体后,叶绿体的弱酸性环境就会促使DNA从纳米颗粒中释放出来,进而合成相应的蛋白。因为植物细胞一般有几十个叶绿体,所以以叶绿体作为生物反应器可以高效地表达如抗体和口服型疫苗等外源蛋白,还可以创制抗病抗虫抗干旱等非生物胁迫的转基因植物材料。
References:
Allen QM, Febres VJ, Rathinasabapathi B, et al. Engineering a Plant-Derived Astaxanthin Synthetic Pathway Into Nicotiana benthamiana. Front Plant Sci. 2022, 12: 831785.
Burnett MJB, Burnett AC. Therapeutic recombinant protein production in plants: Challenges and opportunities. Plants, People, Planet, 2020, 2(2): 121-132.
Dong W, Stockwell VO, Goyer A. Enhancement of Thiamin Content in Arabidopsis thaliana by Metabolic Engineering. Plant Cell Physiol. 2015, 56(12): 2285-96.
Feike D, Korolev AV, Soumpourou E, et al. Characterizing standard genetic parts and establishing common principles for engineering legume and cereal roots. Plant Biotechnol J. 2019, 17(12): 2234-2245.
Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome. Science. 2010, 329(5987): 52-6.
Hartley JL, Temple GF, and Brasch MA. DNA cloning using in vitro site-specific recombination. Genome Res. 2000, 10: 1788-1795.
Hwang HH, Yu M, Lai EM. Agrobacterium-mediated plant transformation: biology and applications. Arabidopsis Book. 2017, 15, e0186.
Hao Y, Zong W, Zeng D, et al. Shortened snRNA promoters for efficient CRISPR/Cas-based multiplex genome editing in monocot plants. Sci China Life Sci. 2020, 63(6): 933-935.
Kuroda H, Maliga P. Complementarity of the 16S rRNA penultimate stem with sequences downstream of the AUG destabilizes the plastid mRNAs. Nucleic Acids Res. 2001, 29(4): 970-5.
Kwak SY, Lew TTS, Sweeney CJ, et al. Chloroplast-selective gene delivery and expression in planta using chitosan-complexed single-walled carbon nanotube carriers. Nat Nanotechnol. 2019, 14(5): 447-455.
Long BM, Hee WY, Sharwood RE, et al. Carboxysome encapsulation of the CO2-fixing enzyme Rubisco in tobacco chloroplasts. Nat Commun. 2018, 9(1):3570.
Lin Q, Zong Y, Xue C, et al. Prime genome editing in rice and wheat. Nat Biotechnol. 2020, 38(5): 582-585.
Li C, Zhang R, Meng X, et al. Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors. Nat Biotechnol. 2020, 38(7): 875-882.
Liu X, Zhang P, Zhao Q, et al. Making small molecules in plants: A chassis for synthetic biology-based production of plant natural products. J Integr Plant Biol. 2022.
Mohanta TK, Bashir T, Hashem A, et al. Genome Editing Tools in Plants. Genes (Basel). 2017, 8(12): 399.
Metje-Sprink J, Menz J, Modrzejewski D, et al. DNA-Free Genome Editing: Past, Present and Future. Front Plant Sci. 2019, 9: 1957.
Molina-Hidalgo FJ, Vazquez-Vilar M, D’Andrea L, et al. Engineering Metabolism in Nicotiana Species: A Promising Future. Trends Biotechnol. 2021, 39(9): 901-913.
Naves ER, de Ávila Silva L, Sulpice R, et al. Capsaicinoids: Pungency beyond Capsicum. Trends Plant Sci. 2019, 24(2): 109-120.
Pérez-González A, Caro E. Benefits of using genomic insulators flanking transgenes to increase expression and avoid positional effects. Sci Rep. 2019, 9(1): 8474.
Pickar-Oliver A, Gersbach CA. The next generation of CRISPR-Cas technologies and applications. Nat Rev Mol Cell Biol. 2019, 20(8): 490-507.
Ruf S, Karcher D, Bock R. Determining the transgene containment level provided by chloroplast transformation. Proc Natl Acad Sci U S A. 2007, 104(17): 6998-7002.
Sleight SC, Bartley BA, Lieviant JA, et al. In-Fusion BioBrick assembly and re-engineering. Nucleic Acids Res. 2010, 38(8): 2624-36.
Shen BR, Wang LM, Lin XL, et al. Engineering a New Chloroplastic Photorespiratory Bypass to Increase Photosynthetic Efficiency and Productivity in Rice. Mol Plant. 2019, 12(2): 199-214.
Tusé D, Nandi S, McDonald KA, et al. The Emergency Response Capacity of Plant-Based Biopharmaceutical Manufacturing-What It Is and What It Could Be. Front Plant Sci. 2020, 11: 594019.
Zhu Q, Tan J, Liu YG. Molecular farming using transgenic rice endosperm. Trends Biotechnol. 2022, 40(10): 1248-1260.
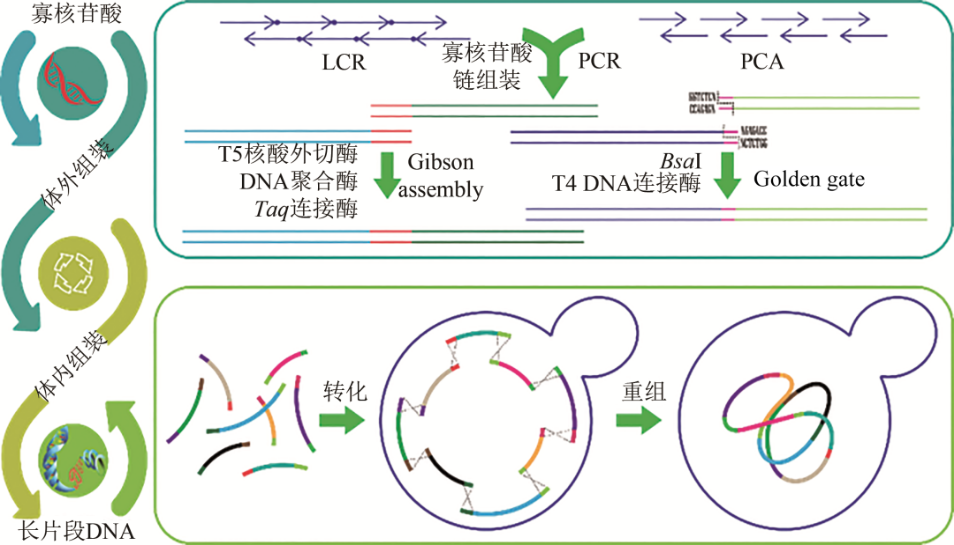
彭凯, 逯晓云, 程健, 等. DNA合成、组装与纠错技术研究进展. 合成生物学, 2020, 1(6): 697-708.
邵洁, 刘海利, 王勇. 植物合成生物学的现在与未来. 合成生物学, 2020, 1(4): 395-412.
张博, 马永硕, 尚轶, 等. 植物合成生物学研究进展. 合成生物学, 2020, 1(2): 121-140.