本文内容速览:
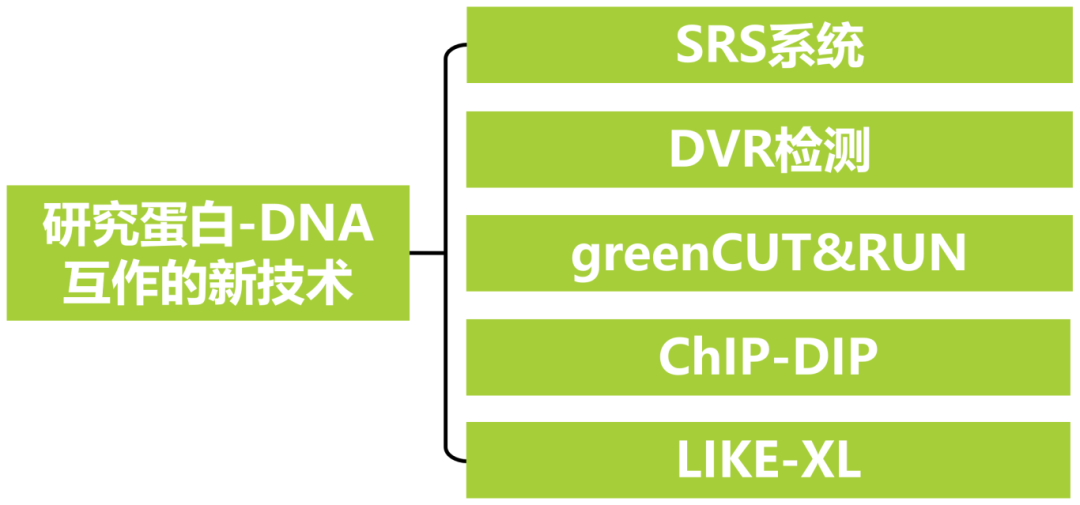
在许多细胞生命活动中,如复制、转录、表观遗传修饰、DNA修复等,都涉及到蛋白-DNA的相互作用。因此,研究蛋白-DNA的相互作用,对于我们了解DNA转录调控、蛋白表达机制以及揭示各种生命活动现象具有重要的指导作用。为了研究两者的互作关系,研究者们发明了很多方法,包括用于寻找目的基因启动子的DNA结合蛋白(反式作用因子)的技术:酵母单杂筛库和DNA Pull-down MS技术;用于寻找反式作用因子在目的基因启动子上结合的顺式作用元件的技术:染色质免疫共沉淀测序(ChIP-seq)、靶向剪切及标签技术(CUT&Tag)和DNA亲和纯化测序(DAP-seq);以及用于鉴定两者之间是否互作的技术:酵母单杂实验(Y1H)、双荧光素酶报告基因实验(Dual-LUC)、凝胶迁移实验(EMSA)、染色质免疫共沉淀-实时荧光定量PCR实验(ChIP-qPCR)和DNA拉下实验(DNA Pull-down)等。
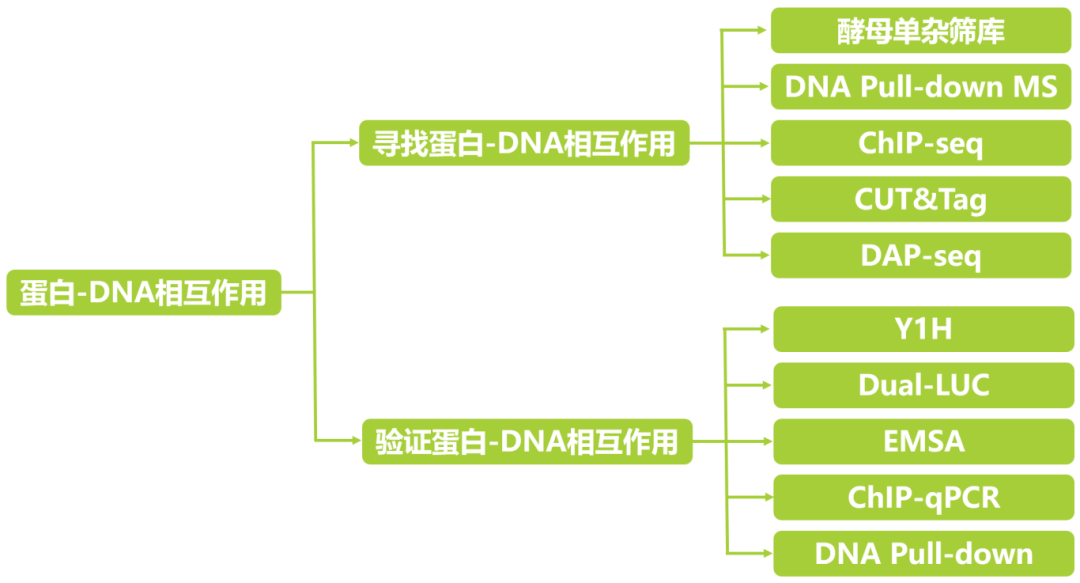
图1 研究蛋白-DNA相互作用的常用筛选技术和点对点验证技术总结。图片来源:《植物基因功能研究范式》。
除了上述常用的技术,随着研究深入,科学家们还开发出了更高效、灵敏的研究蛋白-DNA互作的新技术,接下来,大家就和小远一起来学习一下吧!
叶片黄化是植物中一种普遍存在的生理现象,其主要成因是叶绿素(Chl)的降解。研究表明,Stay-Green1(SGR1)基因在叶片衰老过程中对Chl的分解起着关键作用。当SGR1发生突变时,植物呈现绿色表型;而该基因的过表达则会导致叶片黄化。基于SGR1介导颜色变化的机理,2024年10月,中国农业科学院生物技术研究所梁成真课题组在Plant Biotechnology Journal杂志上发表了一篇题为“SRS: an intelligent and robust approach for confirmation of plant transcription factor-DNA interactions”的研究论文,该研究提出了一种以SGR1作为报告基因,用于探究蛋白和DNA之间的相互作用的新技术SGR1-based reporter system(SRS)。
作者首先从SGR1基因的三个亚组中分别挑选了来自拟南芥的AtSGR1、水稻的OsSGR1和银杏的GbSGR1、GbSGR1L,在烟草叶片中进行瞬时表达。结果显示,注射p35S::AtSGR1的烟草叶片表现出最为显著的叶绿素降解速率和黄化表型,这一结果表明AtSGR1更适合作为验证蛋白和DNA相互作用的报告基因(图2a)。为了消除烟草中NbSGR1同源基因非特异性激活可能导致的假阳性,作者利用CRISPR/Cas9技术敲除了烟草中的6个NbSGR1基因,成功创制了用于SRS系统的SGR1敲除植株CR19,该突变体在黑暗条件下表现出叶片黄化延迟和Chl降解缓慢的表型(图2e-h)。同时,作者还开发了用于追踪叶片由绿色到黄色变化的智能模型SMGY,通过小程序即可实现对叶片中叶绿素变化的检测(图2d)。
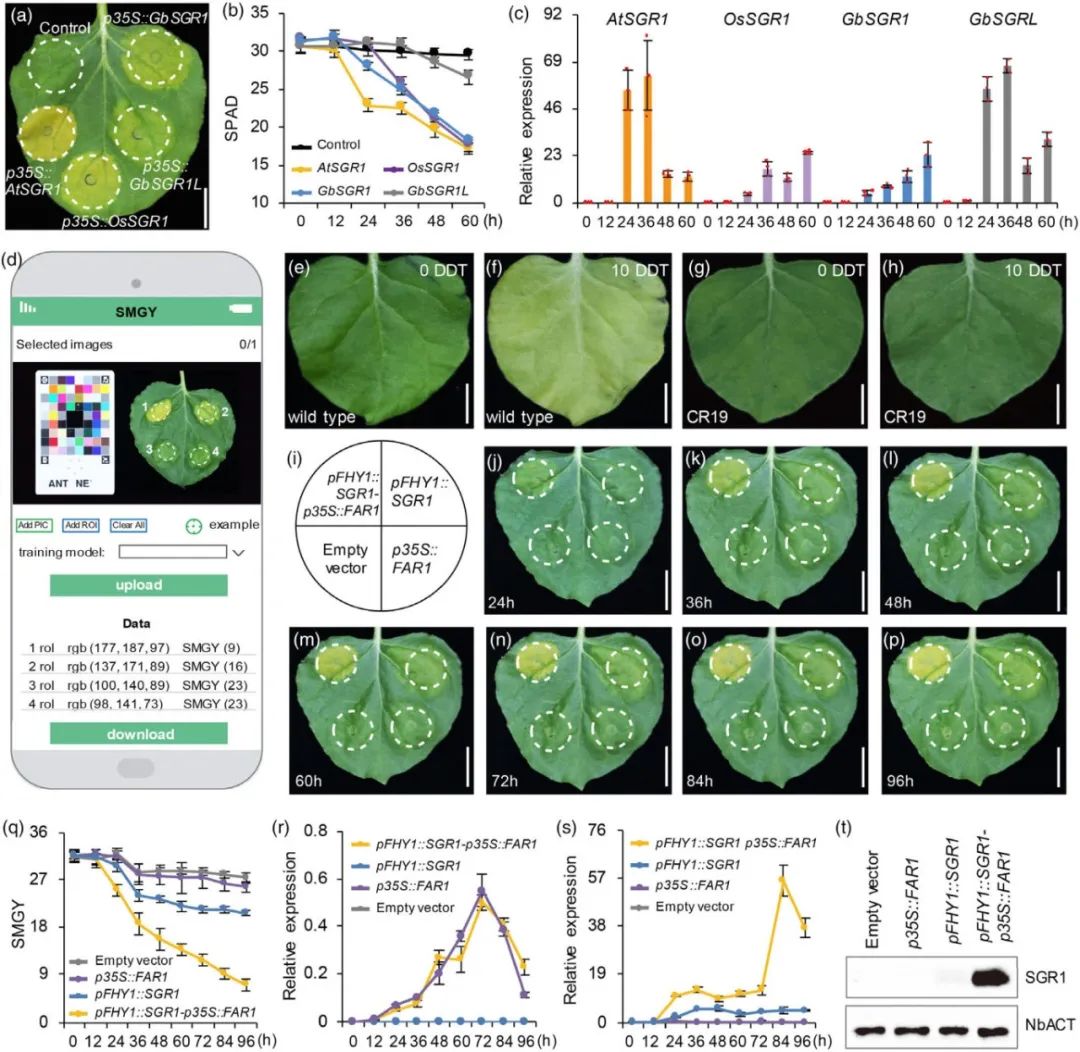 图2 SRS系统的开发和利用(Zhou et al., 2024)。(a)注射不同SGR1基因后本氏烟叶片的表型变化;(b)注射区域Chl含量的动态变化;(c)注射区域中SGR1的相对表达水平;(d)SMGY模型的设计和工作流程;(e-h)黑暗处理10天后野生型和NbSGR1基因编辑叶片的的表型;(i-p)注射pFHY1::SGR1-p35S::FAR1的本氏烟叶片随时间变化的动态表型;(q)注射区域Chl含量的动态变化;(r-s)注射区域中FAR1和SGR1的相对表达水平;(t)注射区域中SGR1的蛋白水平。
为了提高实验操作的便捷性,作者还开发了pTF-SGR1载体(图3)。先前的研究报道表明FAR1和FHY1启动子可以互作(Lin et al., 2007),于是作者将构建好的pFHY1::SGR1-p35S::FAR1载体用于评估SRS系统,在烟草叶片中瞬时表达载体后,与对照组相比,实验组可以观察到显著的颜色变化(图2i-p)。并且检测结果显示FAR1和FHY1启动子同时存在的情况下,SGR1的表达显著升高(图2s、t)。这一结果证实利用SRS系统可以用于研究蛋白和DNA的相互作用。为了验证SRS系统的灵敏性和特异性,作者还使用突变后的FAR1和不与FAR1互作的OsTB1启动子进行测试,结果显示pFHY1::SGR1-p35S::mFAR1和pOsTBI::SGR1-p35S::FAR1与空载相比,均无明显差异。
图2 SRS系统的开发和利用(Zhou et al., 2024)。(a)注射不同SGR1基因后本氏烟叶片的表型变化;(b)注射区域Chl含量的动态变化;(c)注射区域中SGR1的相对表达水平;(d)SMGY模型的设计和工作流程;(e-h)黑暗处理10天后野生型和NbSGR1基因编辑叶片的的表型;(i-p)注射pFHY1::SGR1-p35S::FAR1的本氏烟叶片随时间变化的动态表型;(q)注射区域Chl含量的动态变化;(r-s)注射区域中FAR1和SGR1的相对表达水平;(t)注射区域中SGR1的蛋白水平。
为了提高实验操作的便捷性,作者还开发了pTF-SGR1载体(图3)。先前的研究报道表明FAR1和FHY1启动子可以互作(Lin et al., 2007),于是作者将构建好的pFHY1::SGR1-p35S::FAR1载体用于评估SRS系统,在烟草叶片中瞬时表达载体后,与对照组相比,实验组可以观察到显著的颜色变化(图2i-p)。并且检测结果显示FAR1和FHY1启动子同时存在的情况下,SGR1的表达显著升高(图2s、t)。这一结果证实利用SRS系统可以用于研究蛋白和DNA的相互作用。为了验证SRS系统的灵敏性和特异性,作者还使用突变后的FAR1和不与FAR1互作的OsTB1启动子进行测试,结果显示pFHY1::SGR1-p35S::mFAR1和pOsTBI::SGR1-p35S::FAR1与空载相比,均无明显差异。
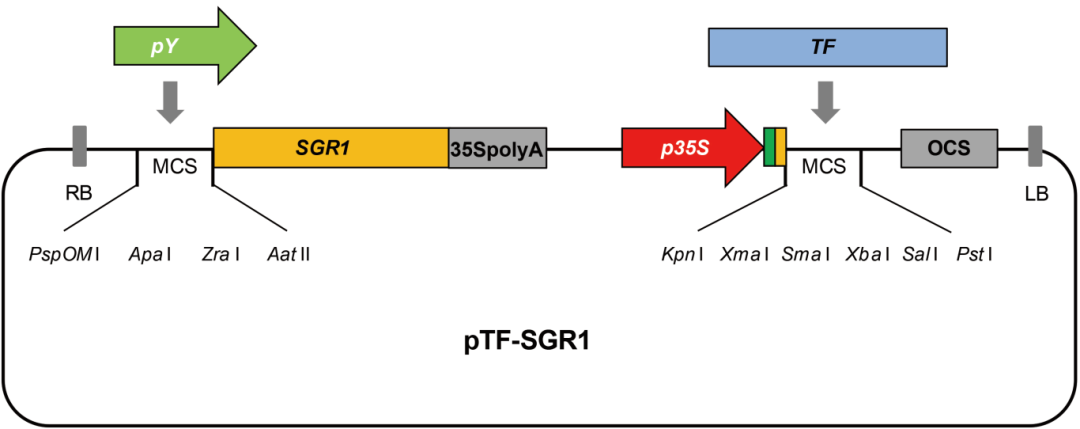 图3 pTF-SGR1载体结构示意图(Zhou et al., 2024)。该载体具有两个独立的表达盒,其中包含多个多克隆位点可以用构建载体。
利用SRS系统,作者成功验证了在拟南芥、水稻、棉花和辣椒中6组已报道的蛋白和DNA的互作,并且进一步测试了该系统在其他物种的适用性,结果显示SRS系统同样适用于油菜和生菜等(图4)。总之,该技术为蛋白-DNA互作研究提供了新的技术方案,有助于基因调控网络的解析。
图3 pTF-SGR1载体结构示意图(Zhou et al., 2024)。该载体具有两个独立的表达盒,其中包含多个多克隆位点可以用构建载体。
利用SRS系统,作者成功验证了在拟南芥、水稻、棉花和辣椒中6组已报道的蛋白和DNA的互作,并且进一步测试了该系统在其他物种的适用性,结果显示SRS系统同样适用于油菜和生菜等(图4)。总之,该技术为蛋白-DNA互作研究提供了新的技术方案,有助于基因调控网络的解析。
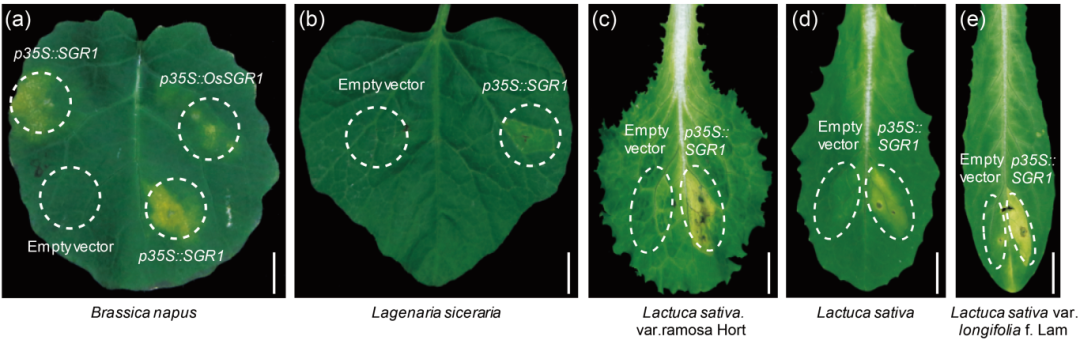 图4 SRS系统在其他物种中的测试结果(Zhou et al., 2024)。
近年来,研究者们陆续开发出了一些新型的可视化报告系统。例如,RUBY系统通过催化合成甜菜红素,可用于可视化基因表达。eYGFPuv作为一种新型绿色荧光蛋白,在紫外光的激发下可表现出明亮的荧光,肉眼就可以观察到其稳定表达。基于这两种报告系统可视化的功能,2023年1月,国际竹藤中心高志民课题组在The Plant Journal杂志上发表了一篇题为“Development of dual-visible reporter assays to determine the DNA-protein interaction”的研究论文,该研究将RUBY和eYGFPuv整合至同一载体中,开发出了一种检测蛋白-DNA互作的新技术,称为Termed dual-visible reporter(DVR)检测。
作者首先在DVR检测中使用了两种RUBY和eYGFPuv的组合形式。方案一是用35S启动子驱动eYGFPuv表达,将DNA顺式作用元件(DE)置于TATA box和RUBY上游。方案二是用35S启动子驱动RUBY表达,将DE置于TATA box和eYGFPuv上游(图5a)。已知ANT1和顺式作用元件ABE结合(Nole-Wilson and Krizek, 2003),通过检测ANT1和ABE的结合情况来测试两个方案。方案一显示,在紫外光下35S::eYGFPuv-ABE::RUBY和35S::ANT1共表达的部位以及单个35S::eYGFPuv-ABE::RUBY表达的区域均观察到了荧光,但只有共表达的区域出现了红色表型。方案二显示,35S::RUBY-ABE::eYGFPuv和35S::ANT1共表达的部位以及单个35S::RUBY-ABE::eYGFPuv表达的区域均观察到了红色表型,在紫外光下可以观察到共表达区域有较弱的荧光(图5b、c)。因此,方案一更适合用于DVR检测。
图4 SRS系统在其他物种中的测试结果(Zhou et al., 2024)。
近年来,研究者们陆续开发出了一些新型的可视化报告系统。例如,RUBY系统通过催化合成甜菜红素,可用于可视化基因表达。eYGFPuv作为一种新型绿色荧光蛋白,在紫外光的激发下可表现出明亮的荧光,肉眼就可以观察到其稳定表达。基于这两种报告系统可视化的功能,2023年1月,国际竹藤中心高志民课题组在The Plant Journal杂志上发表了一篇题为“Development of dual-visible reporter assays to determine the DNA-protein interaction”的研究论文,该研究将RUBY和eYGFPuv整合至同一载体中,开发出了一种检测蛋白-DNA互作的新技术,称为Termed dual-visible reporter(DVR)检测。
作者首先在DVR检测中使用了两种RUBY和eYGFPuv的组合形式。方案一是用35S启动子驱动eYGFPuv表达,将DNA顺式作用元件(DE)置于TATA box和RUBY上游。方案二是用35S启动子驱动RUBY表达,将DE置于TATA box和eYGFPuv上游(图5a)。已知ANT1和顺式作用元件ABE结合(Nole-Wilson and Krizek, 2003),通过检测ANT1和ABE的结合情况来测试两个方案。方案一显示,在紫外光下35S::eYGFPuv-ABE::RUBY和35S::ANT1共表达的部位以及单个35S::eYGFPuv-ABE::RUBY表达的区域均观察到了荧光,但只有共表达的区域出现了红色表型。方案二显示,35S::RUBY-ABE::eYGFPuv和35S::ANT1共表达的部位以及单个35S::RUBY-ABE::eYGFPuv表达的区域均观察到了红色表型,在紫外光下可以观察到共表达区域有较弱的荧光(图5b、c)。因此,方案一更适合用于DVR检测。
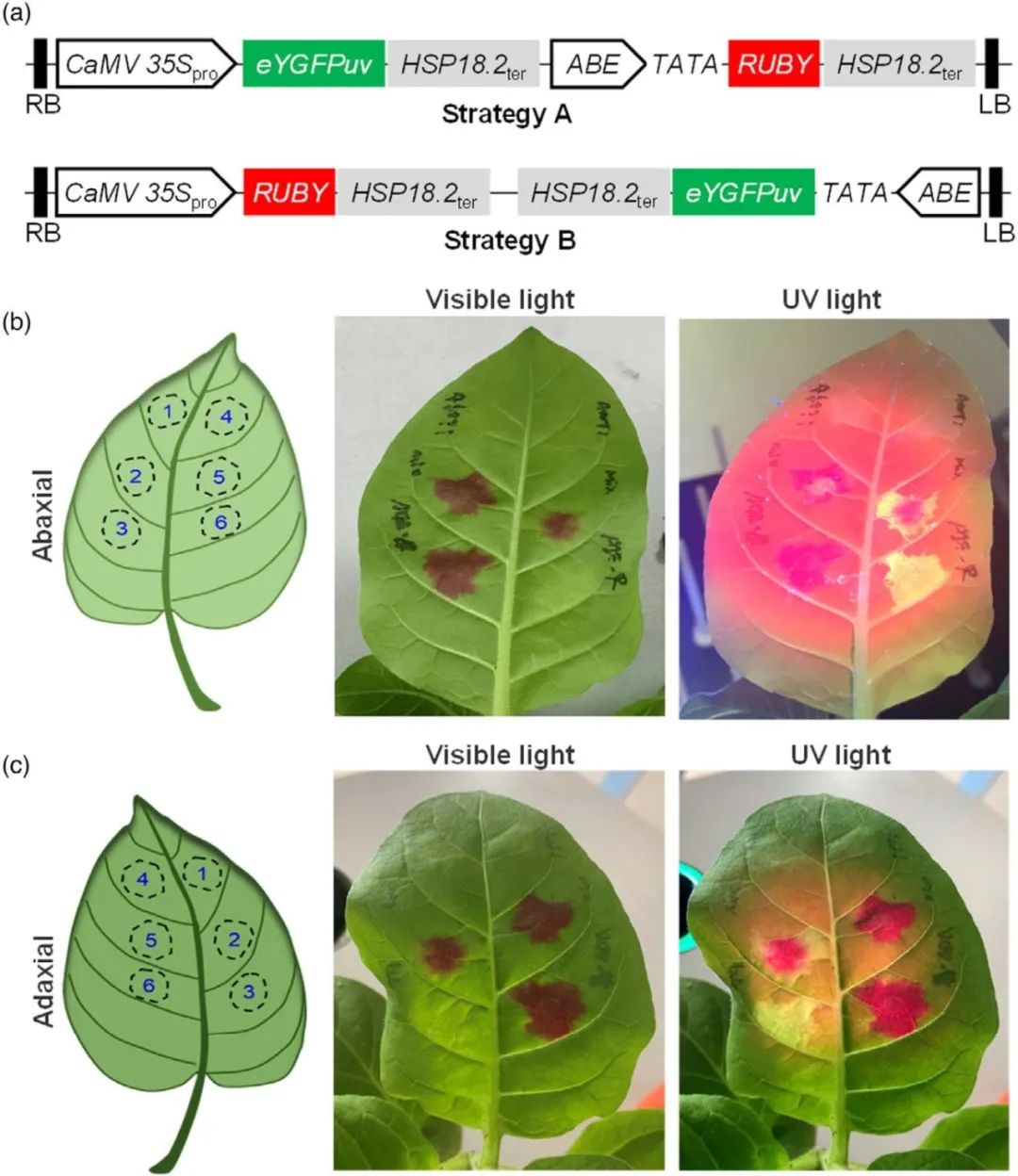 图5 eYGFPuv和RUBY的检测结果反映了烟草叶中ANT1的ABE结合活性(Sun et al., 2023)。(a)两种方案的载体构建示意图;(b、c)左侧为烟草叶片注射区域的示意图,右侧为拍摄的叶片表型,反面(b)和正面(c),1-6是农杆菌悬浮液的注射区域,其中1和4处注射的是35S::ANT1,2是35S::RUBY-ABE::eYGFPuv和35S::ANT1的混合物,3是35S::RUBY-ABE::eYGFPuv,5是35S::eYGFPuv-ABE::RUBY和35S::ANT1的混合物,6是35S::eYGFPuv-ABE::RUBY。
接下来,作者利用DVR检测对先前已发表的一些结果进行验证,来进一步评估该技术。结果显示,ANT1不与EBE结合,AtERF-1对ABE具有弱结合亲和力,对EBE具有强结合亲和力,AtERF-2对EBE具有强结合亲和力但对ABE没有结合亲和力(图6)。这些结果与先前的结论一致(Gong et al., 2008; Krizek, 2003; Nole-Wilson and Krizek, 2000)。随后作者还克隆了毛竹AP2/ERF转录因子家族的基因,利用DVR检测鉴定了这些转录因子与顺式元件ABE、DBE、EBE和RBE的结合情况,总之,该技术的开发为科研工作者提供了一种低成本、便捷的蛋白-DNA互作检测方法。
图5 eYGFPuv和RUBY的检测结果反映了烟草叶中ANT1的ABE结合活性(Sun et al., 2023)。(a)两种方案的载体构建示意图;(b、c)左侧为烟草叶片注射区域的示意图,右侧为拍摄的叶片表型,反面(b)和正面(c),1-6是农杆菌悬浮液的注射区域,其中1和4处注射的是35S::ANT1,2是35S::RUBY-ABE::eYGFPuv和35S::ANT1的混合物,3是35S::RUBY-ABE::eYGFPuv,5是35S::eYGFPuv-ABE::RUBY和35S::ANT1的混合物,6是35S::eYGFPuv-ABE::RUBY。
接下来,作者利用DVR检测对先前已发表的一些结果进行验证,来进一步评估该技术。结果显示,ANT1不与EBE结合,AtERF-1对ABE具有弱结合亲和力,对EBE具有强结合亲和力,AtERF-2对EBE具有强结合亲和力但对ABE没有结合亲和力(图6)。这些结果与先前的结论一致(Gong et al., 2008; Krizek, 2003; Nole-Wilson and Krizek, 2000)。随后作者还克隆了毛竹AP2/ERF转录因子家族的基因,利用DVR检测鉴定了这些转录因子与顺式元件ABE、DBE、EBE和RBE的结合情况,总之,该技术的开发为科研工作者提供了一种低成本、便捷的蛋白-DNA互作检测方法。
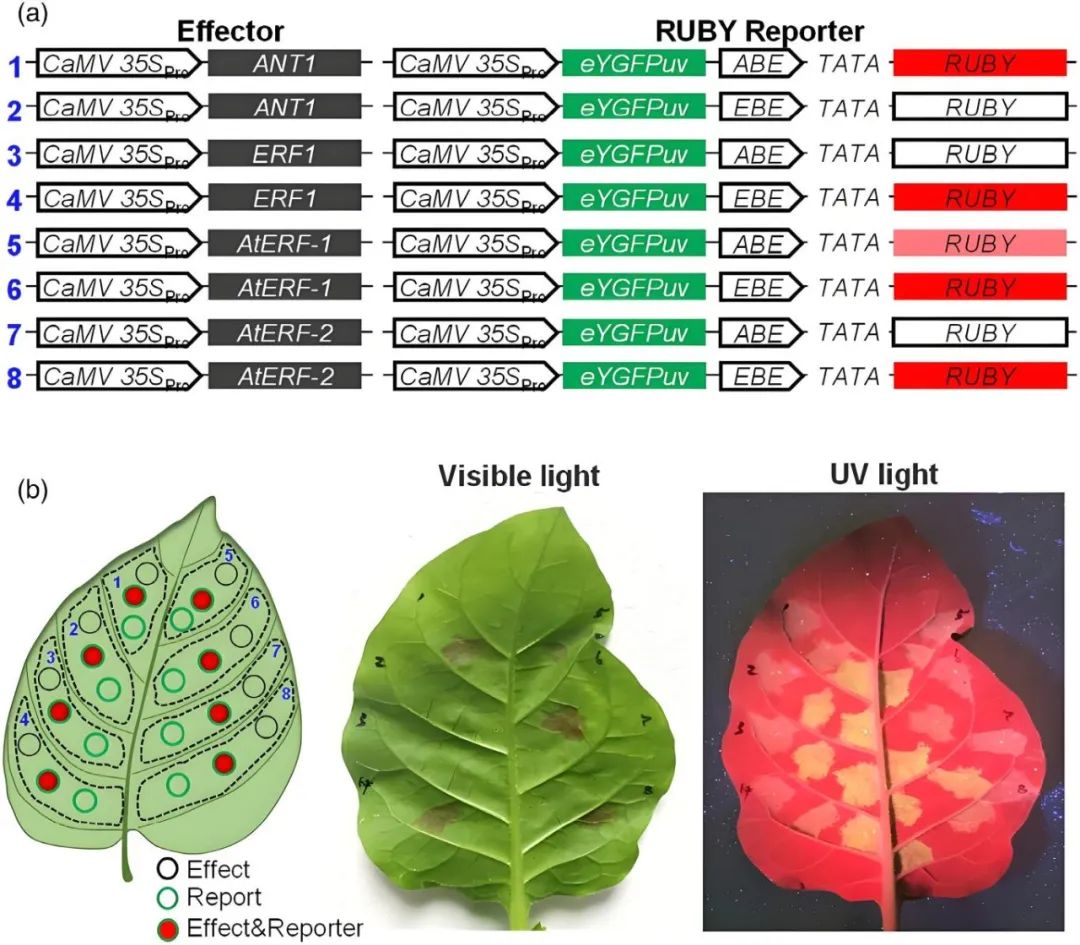 图6 拟南芥AP2/ERF家族被用作DVR检测中DNA结合分析的转录因子模型(Sun et al., 2023)。(a)载体构建示意图,结合活性弱的基因用浅红色表示,结合活性较强的基因用深红色表示;(b)拟南芥AP2/ERF的DNA结合活性的结果。
介绍完蛋白-DNA互作点对点验证的新技术之后,接下来给大家介绍几个蛋白-DNA互作筛选的新技术。
greenCUT&RUN-天然染色质状态下的精细定位
CUT&RUN(Cleavage Under Targets and Release Using Nuclease)是一种用于研究染色质中蛋白和DNA相互作用的高通量技术(图7)。其原理是使用特异性抗体靶向目标蛋白,接着加入核酸酶MNase(Protein A/G-Micrococcal Nuclease),该核酸酶能在抗体结合位点特异性切割DNA,并释放目标蛋白结合的DNA片段。随后通过测序等方法对回收的DNA片段进行分析,可以精准的确定蛋白在基因组上的结合位点(Hainer and Fazzio, 2019)。该技术不需要交联,相较于ChIP-seq方法,能够减少交联引起的假阳性,可在天然染色质状态下鉴定目标蛋白结合的位点(Meers et al., 2019)。
greenCUT&RUN技术是CUT&RUN的改进版本(图7),它进一步提高了CUT&RUN技术的灵敏度,其中GFP特异性纳米抗体直接与MNase融合,可在天然染色质状态下特异性切割GFP标记蛋白结合的DNA区域(Koidl and Timmers, 2021)。
图6 拟南芥AP2/ERF家族被用作DVR检测中DNA结合分析的转录因子模型(Sun et al., 2023)。(a)载体构建示意图,结合活性弱的基因用浅红色表示,结合活性较强的基因用深红色表示;(b)拟南芥AP2/ERF的DNA结合活性的结果。
介绍完蛋白-DNA互作点对点验证的新技术之后,接下来给大家介绍几个蛋白-DNA互作筛选的新技术。
greenCUT&RUN-天然染色质状态下的精细定位
CUT&RUN(Cleavage Under Targets and Release Using Nuclease)是一种用于研究染色质中蛋白和DNA相互作用的高通量技术(图7)。其原理是使用特异性抗体靶向目标蛋白,接着加入核酸酶MNase(Protein A/G-Micrococcal Nuclease),该核酸酶能在抗体结合位点特异性切割DNA,并释放目标蛋白结合的DNA片段。随后通过测序等方法对回收的DNA片段进行分析,可以精准的确定蛋白在基因组上的结合位点(Hainer and Fazzio, 2019)。该技术不需要交联,相较于ChIP-seq方法,能够减少交联引起的假阳性,可在天然染色质状态下鉴定目标蛋白结合的位点(Meers et al., 2019)。
greenCUT&RUN技术是CUT&RUN的改进版本(图7),它进一步提高了CUT&RUN技术的灵敏度,其中GFP特异性纳米抗体直接与MNase融合,可在天然染色质状态下特异性切割GFP标记蛋白结合的DNA区域(Koidl and Timmers, 2021)。
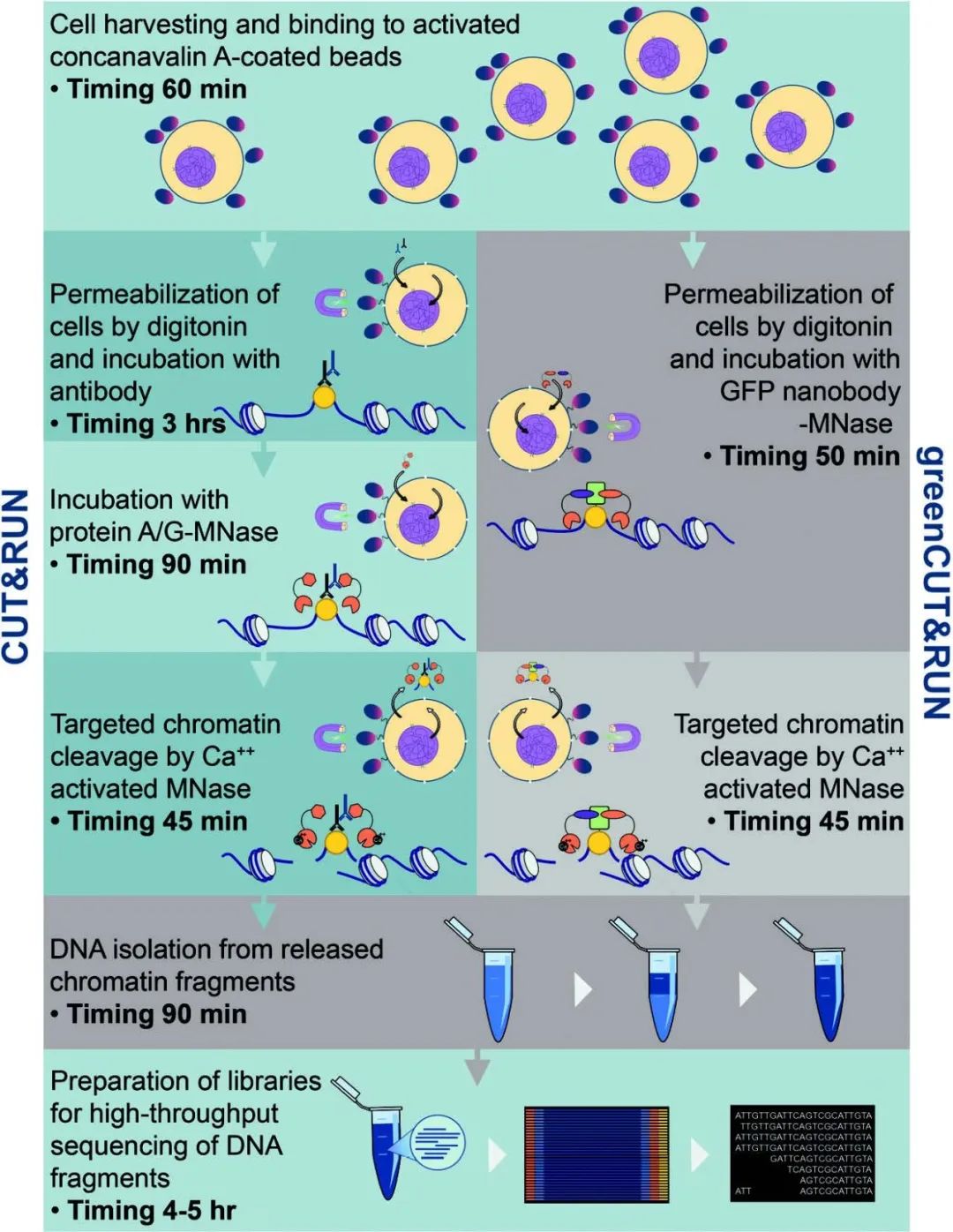 图7 流程图包括CUT&RUN(左)和greenCUT&RUN(右)方案中各个步骤所需的时间(Koidl and Timmers, 2021)。
2024年10月,杜克大学董欣年课题组在Molecular Plant杂志上发表了一篇题为“Next-generation mapping of the salicylic acid signaling hub and transcriptional cascade”的研究论文,报道了水杨酸(SA)信号转导枢纽中新组分的发现和相关信号转导途径的调控机制。在这篇文章中作者为了进一步探索NPR1的转录靶标,使用greenCUT&RUN技术对SA诱导下的NPR1进行了分析。结果显示,NPR1主要结合在靶基因的启动子区域,且TGA结合的as-1元件TGACG为最显著富集的基序,这一结果进一步证实了NPR1通过TGA转录因子与启动子结合的观点(图8)。
图7 流程图包括CUT&RUN(左)和greenCUT&RUN(右)方案中各个步骤所需的时间(Koidl and Timmers, 2021)。
2024年10月,杜克大学董欣年课题组在Molecular Plant杂志上发表了一篇题为“Next-generation mapping of the salicylic acid signaling hub and transcriptional cascade”的研究论文,报道了水杨酸(SA)信号转导枢纽中新组分的发现和相关信号转导途径的调控机制。在这篇文章中作者为了进一步探索NPR1的转录靶标,使用greenCUT&RUN技术对SA诱导下的NPR1进行了分析。结果显示,NPR1主要结合在靶基因的启动子区域,且TGA结合的as-1元件TGACG为最显著富集的基序,这一结果进一步证实了NPR1通过TGA转录因子与启动子结合的观点(图8)。
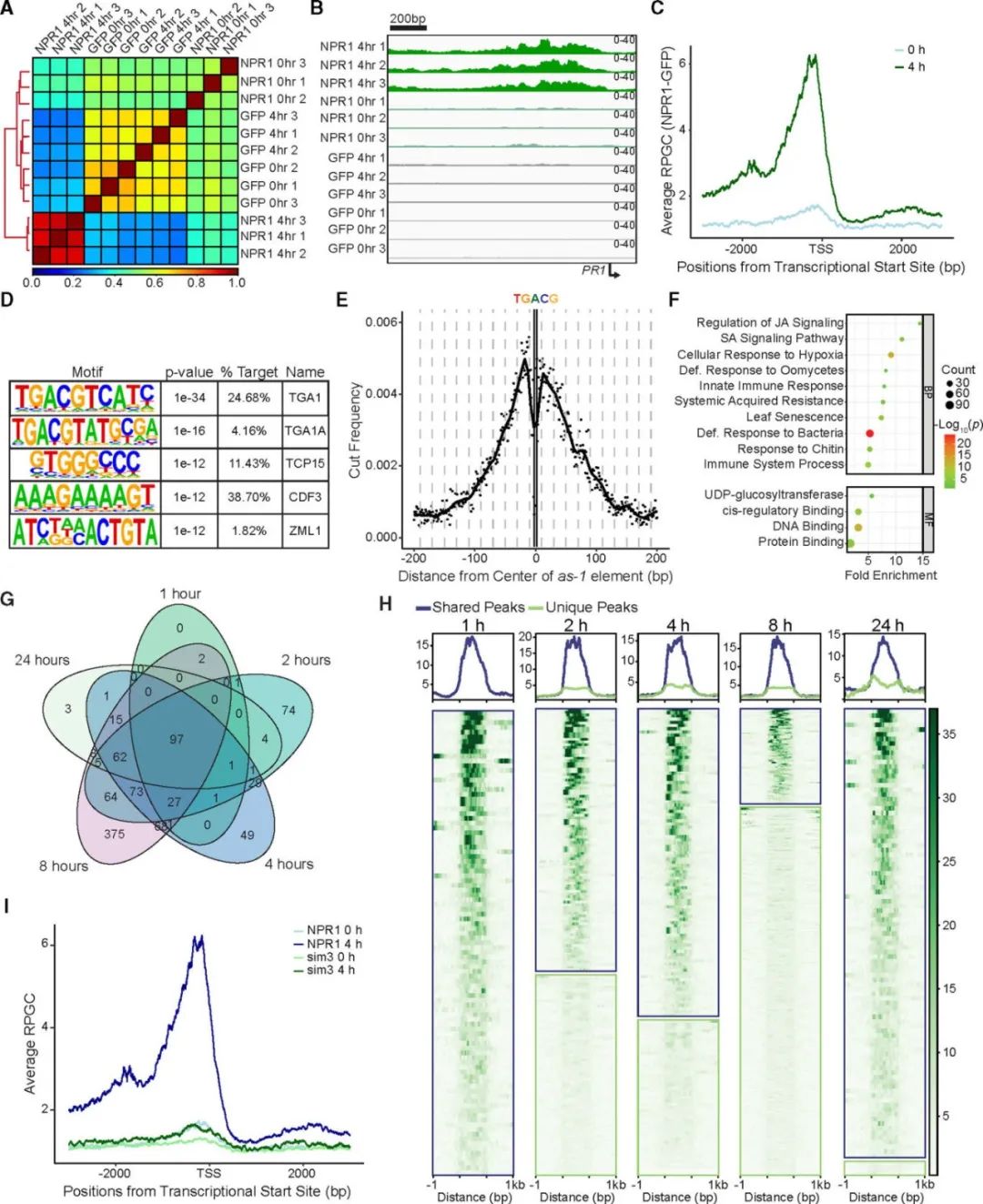 图8 NPR1通过与TGA转录因子结合靶向靶基因启动子(Powers et al., 2024)。(A)表达NPR1-GFP(NPR1)和GFP的植物在1mM SA处理4小时前后的greenCUT&RUN数据的皮尔逊相关性分析;(B)PR1启动子的IGV视图;(C)SA处理前后NPR1-GFP在NPR1靶基因处的平均读段密度(RPGCs)分布;(D)1mM SA处理4小时后,NPR1-GFP峰下富集的基序;(E)1mM SA处理4小时后,GFP纳米抗体MNase在所有NPR1-GFP峰中对as-1元件(TGACG)的切割频率;(F)NPR1靶基因富集的生物过程和分子功能;(G)1mM SA处理后1、2、4、8和24小时共享NPR1位点的维恩图;(H)1mM SA处理后1、2、4、8和24小时,共享位点(蓝色框)和独特位点(绿色框)处NPR1结合的标准化(RPGC)热图和平均分布;(I)SA处理前后,NPR1-GFP和npr1sim3-GFP(sim3)在NPR1靶基因处的RPGC分布。
传统的实验技术,如ChIP-seq通常只能解析单一蛋白与DNA的结合,这导致我们只能捕捉到基因调控网络的局部信息。那么如何能够同时捕捉多个蛋白和DNA的相互作用呢?
2024年12月,加州理工学院Mitchell Guttman课题组在Nature Genetics杂志上发表了一篇题为“ChIP-DIP maps binding of hundreds of proteins to DNA simultaneously and identifies diverse gene regulatory elements”的研究论文,该论文报道的ChIP-DIP(Chromatin immunoprecipitation done in parallel)技术通过单次实验即可对数百种调控蛋白与DNA的结合情况进行检测,从而生成这些蛋白的全基因组图谱,为全面解析基因调控的动态变化提供了有力的工具。
图8 NPR1通过与TGA转录因子结合靶向靶基因启动子(Powers et al., 2024)。(A)表达NPR1-GFP(NPR1)和GFP的植物在1mM SA处理4小时前后的greenCUT&RUN数据的皮尔逊相关性分析;(B)PR1启动子的IGV视图;(C)SA处理前后NPR1-GFP在NPR1靶基因处的平均读段密度(RPGCs)分布;(D)1mM SA处理4小时后,NPR1-GFP峰下富集的基序;(E)1mM SA处理4小时后,GFP纳米抗体MNase在所有NPR1-GFP峰中对as-1元件(TGACG)的切割频率;(F)NPR1靶基因富集的生物过程和分子功能;(G)1mM SA处理后1、2、4、8和24小时共享NPR1位点的维恩图;(H)1mM SA处理后1、2、4、8和24小时,共享位点(蓝色框)和独特位点(绿色框)处NPR1结合的标准化(RPGC)热图和平均分布;(I)SA处理前后,NPR1-GFP和npr1sim3-GFP(sim3)在NPR1靶基因处的RPGC分布。
传统的实验技术,如ChIP-seq通常只能解析单一蛋白与DNA的结合,这导致我们只能捕捉到基因调控网络的局部信息。那么如何能够同时捕捉多个蛋白和DNA的相互作用呢?
2024年12月,加州理工学院Mitchell Guttman课题组在Nature Genetics杂志上发表了一篇题为“ChIP-DIP maps binding of hundreds of proteins to DNA simultaneously and identifies diverse gene regulatory elements”的研究论文,该论文报道的ChIP-DIP(Chromatin immunoprecipitation done in parallel)技术通过单次实验即可对数百种调控蛋白与DNA的结合情况进行检测,从而生成这些蛋白的全基因组图谱,为全面解析基因调控的动态变化提供了有力的工具。
ChIP-DIP技术的实验流程是首先将多种抗体与带有独特寡核苷酸标签的磁珠结合,形成抗体-磁珠池并用于ChIP实验中,以捕获与目标蛋白质相结合的染色质区域。接着将DNA-目标蛋白-抗体-磁珠-寡核苷酸标签复合物分到96孔板中,每个孔内所有目标蛋白结合的DNA和寡核苷酸标签末端会被连上相同的barcode,之后将每个孔中的复合物收集到一起混合均匀后再次分到不同的96孔板中连接barcode,如此至少重复5次。在后续的测序过程中,所有具有相同barcode的DNA读段(reads)会被归为同一簇,这样可以将DNA区域与其对应的抗体进行匹配,从而为每个目标蛋白生成定位图谱(图9a)。
为了验证该技术的准确性和高效性,作者选择了4种典型的蛋白质在人类K562细胞中进行ChIP-DIP测试。结果显示,ChIP-DIP与传统的ChIP-seq生成的蛋白-DNA结合图谱基本一致,表现出良好的准确性和高效性(图9b-d)。此外,ChIP-DIP技术能高效地检测低丰度蛋白质的结合,尤其是转录因子。并且该技术在较少的样品中即可产生可靠的数据,可应用于稀有样本的分析。该技术还能够揭示基因调控的动态过程等。总之,ChIP-DIP技术的开发有望成为解析基因调控网络的关键工具。
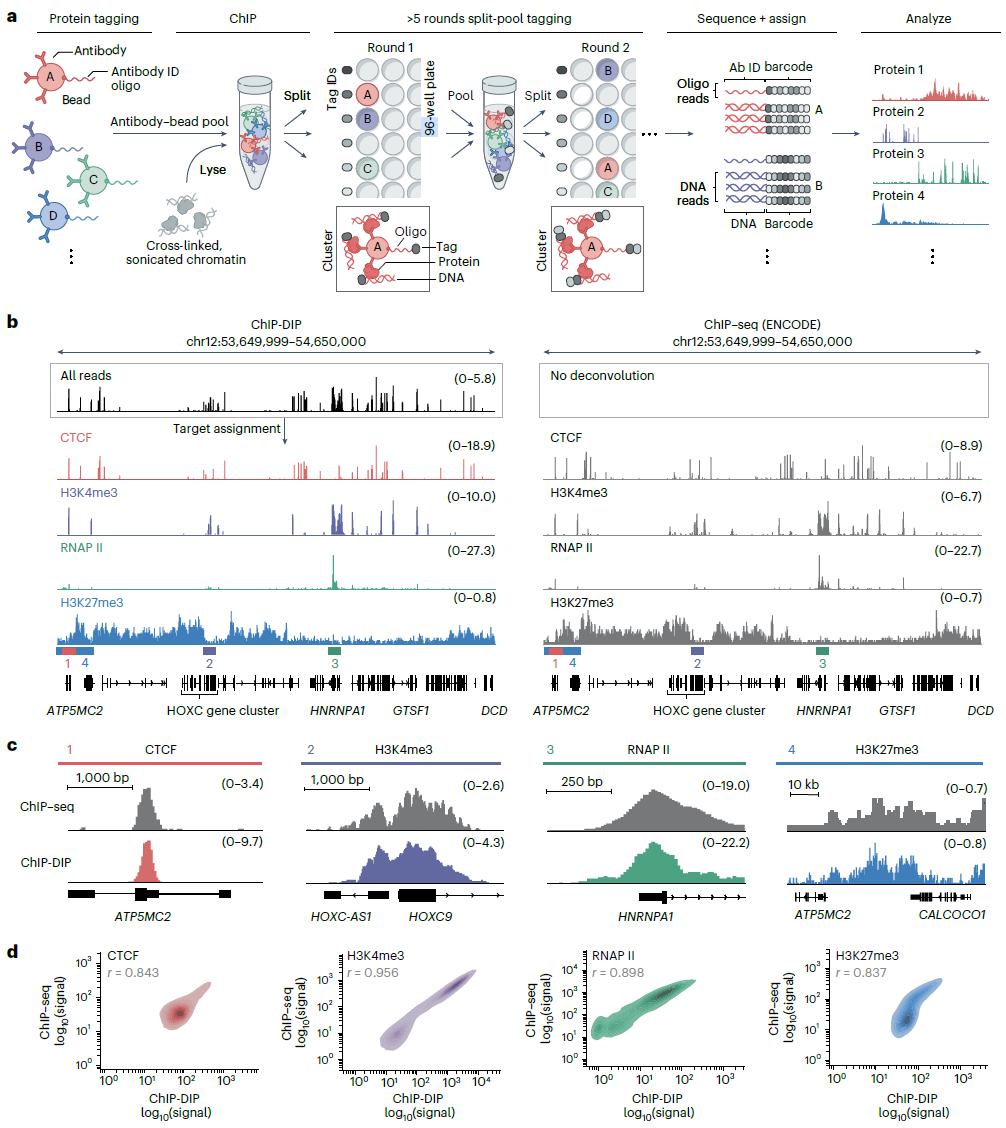
图9 ChIP-DIP的实验流程和测试结果(Perez et al., 2024)。(a)ChIP-DIP的实验流程示意图;(b)四种蛋白(CTCF、H3K4me3、RNAP II、H3K27me3)在特定人类基因组区域(hg38,12号染色体:53,649,999-54,650,000)的定位图谱,左侧ChIP-DIP的结果,右侧是ChIP-seq的结果;(c)ChIP-DIP与ChIP-seq在局部区域的定位图谱对比;(d)每个蛋白质通过ChIP-DIP(x轴)或ChIP-seq(y轴)测量的定位之间的全基因组比较(信号相关性的密度图)。
目前对于生物体中低丰度的蛋白质,以及动态、微弱的蛋白质-DNA复合物的分析仍极具挑战。2023年4月,中国科学院上海药物研究所陈小华课题组与谭敏佳课题组联合在Nature Chemistry杂志上发表了一篇题为“Spatiotemporal and global profiling of DNA-protein interactions enables discovery of low-affinity transcription factors”的研究论文,该研究报道了一种具有赖氨酸选择性的蛋白质-DNA交联方法LIKE-XL(Light-Induced Lysine (K) Enabled Crosslinking),可对蛋白-DNA互作进行时空和全局性分析。该方法能够捕获低亲和力的转录因子(TF)-DNA互作,发现TF及其结合位点。
在先前的研究中,作者开发了光诱导的伯胺与邻硝基苄醇(o-NBA)环化(PANAC)光点击反应(Hu et al., 2019; Guo et al., 2020)。在该反应中,o-NBA官能团在光激活后会转化为芳基-亚硝基中间体,并以快速动力学选择性捕捉伯胺。在生理条件下,芳基-亚硝基中间体具有可靠的化学选择性、高反应活性、良好的生物相容性和较长的半衰期。因此,o-NBA官能团作为一种理想且易于获得的交联剂,可通过偶联到感兴趣的寡核苷酸和DNA序列中,用于在复杂生物环境中以共价方式捕捉蛋白-DNA相互作用。在该论文中,作者设计合成了含光交联基团的DNA探针,该探针能够在低浓度(微摩尔浓度)、短时间(5-10分钟)高效交联相互作用的蛋白质,结合质谱分析即可鉴定到与目标探针结合的互作蛋白(图10)。
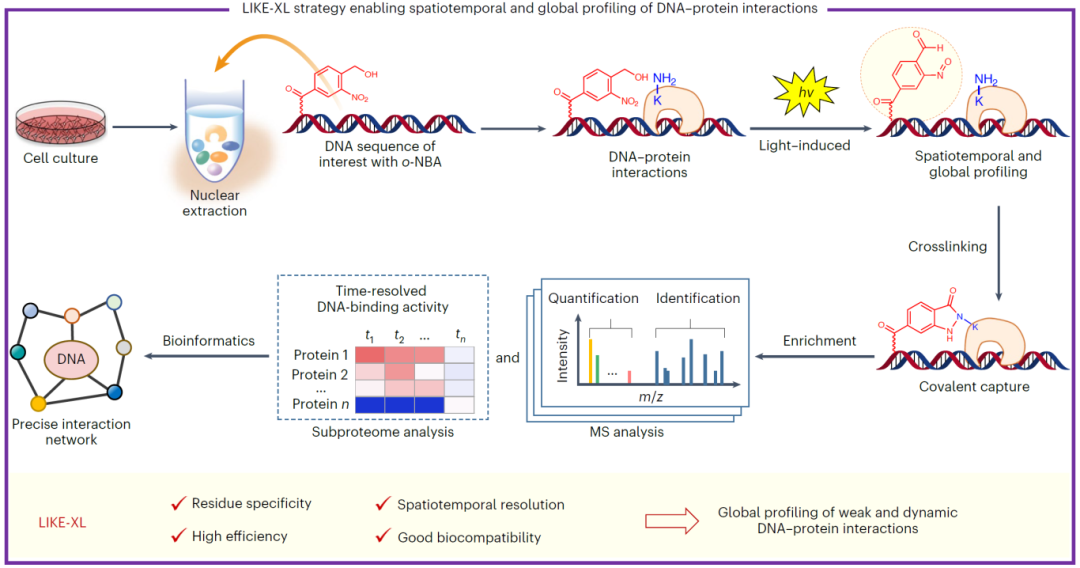 图10 LIKE-XL的实验流程图(Guo et al., 2023)。
为了探索LIKE-XL方法交联蛋白的可行性和效率,作者以双链DNA探针(dsDNAo-NBA)与富含赖氨酸的重组组蛋白交联的反应为例进行了实验。结果显示,光诱导后,dsDNA1o-NBA可以与组蛋白H3偶联,而无光诱导组则不会发生偶联反应。SDS-PAGE结果显示,在低浓度dsDNA1o-NBA(1equiv.)和短时间(3分钟)光诱导条件下,偶联反应便可发生。此外,作者还探究了序列特异性DNA探针对转录因子蛋白MAX的特异性,结果显示,光诱导后,o-NBA探针可与MAX蛋白偶联,而无光诱导组则不会发生偶联反应,并且相同条件下对照组牛血清蛋白(BSA,含有60个赖氨酸)中未观察到偶联(图11)。
图10 LIKE-XL的实验流程图(Guo et al., 2023)。
为了探索LIKE-XL方法交联蛋白的可行性和效率,作者以双链DNA探针(dsDNAo-NBA)与富含赖氨酸的重组组蛋白交联的反应为例进行了实验。结果显示,光诱导后,dsDNA1o-NBA可以与组蛋白H3偶联,而无光诱导组则不会发生偶联反应。SDS-PAGE结果显示,在低浓度dsDNA1o-NBA(1equiv.)和短时间(3分钟)光诱导条件下,偶联反应便可发生。此外,作者还探究了序列特异性DNA探针对转录因子蛋白MAX的特异性,结果显示,光诱导后,o-NBA探针可与MAX蛋白偶联,而无光诱导组则不会发生偶联反应,并且相同条件下对照组牛血清蛋白(BSA,含有60个赖氨酸)中未观察到偶联(图11)。

图11 LIKE-XL方法用于共价捕获DNA-组蛋白以及特异性DNA-TF相互作用(Guo et al., 2023)。(a)通过LIKE-XL对DNA探针和组蛋白的捕获示意图;(b)不同浓度的dsDNA1o-NBA探针与组蛋白H3蛋白的交联情况;(c)dsDNA1o-NBA探针与组蛋白H3蛋白交联的DNA光激活时间的优化;(d)通过LIKE-XL对o-NBA探针和MAX蛋白的捕获示意图;(e)o-NBA探针与MAX的交联情况;(f)o-NBA探针与BSA的交联情况。
接下来,作者探究了在复杂的生物环境下,LIKE-XL方法是否可以对蛋白-DNA互作进行全局分析。通过实验总共获得了129种目标结合蛋白,包括16种HD-TF和26种bHLH-TF,并且这些TF中包含几个低亲和力TF(图12)。此外,作者还通过o-NBA探针对表观遗传药物(如组蛋白去乙酰化酶抑制剂SAHA)调控后,相关TF亚蛋白质组的动态进行了时间分辨的全局分析。总之,该技术的开发为蛋白-DNA互作研究提供了一种新方法。
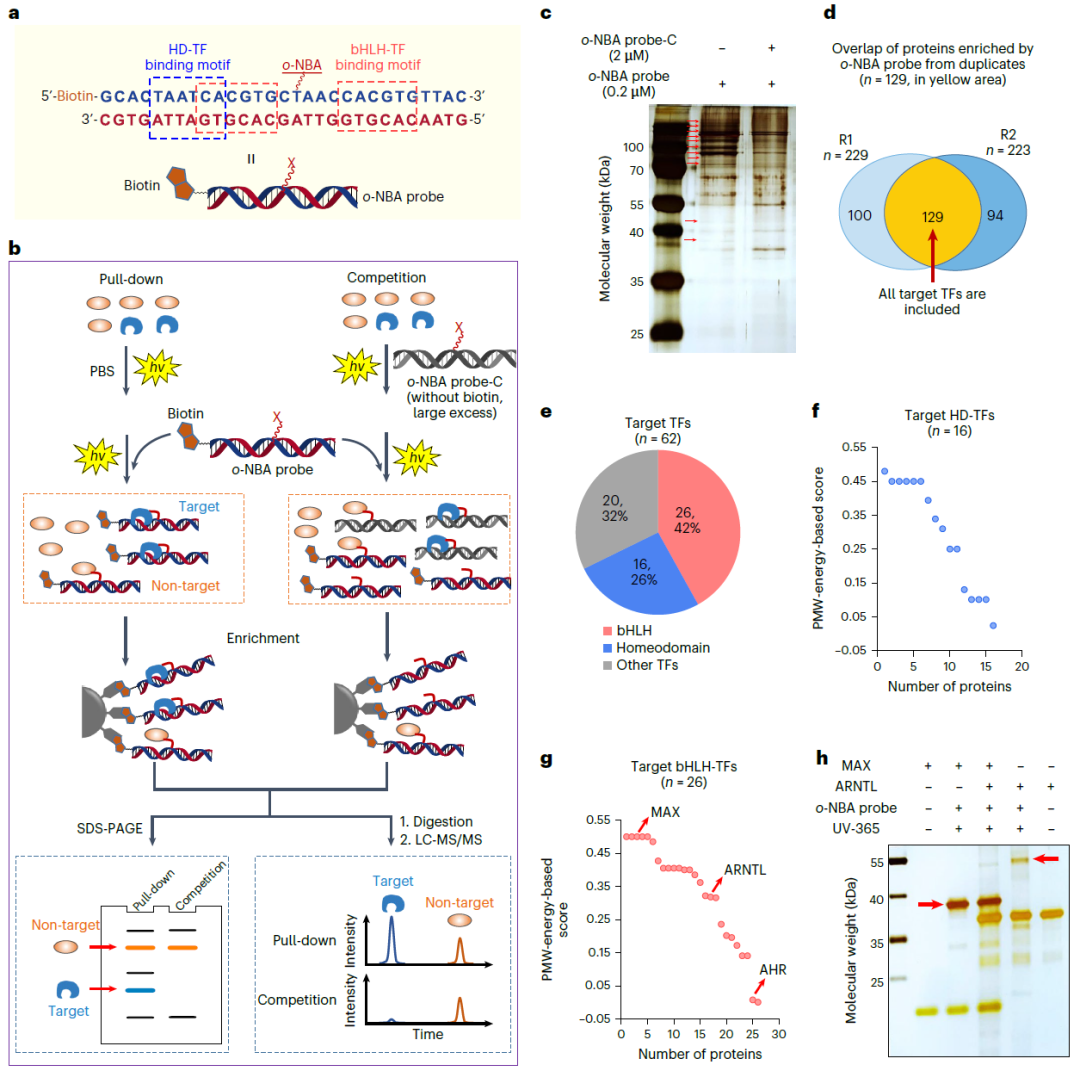 图12 基于o-NBA探针的竞争性交联下拉法来分析特定的DNA-TF的相互作用(Guo et al., 2023)。(a)o-NBA探针的结构和序列;(b)竞争交联下拉法的实验流程;(c)下拉组和竞争组的银染结果;(d)在两次重复实验中均富集到的蛋白质被认为是o-NBA探针的靶标结合蛋白;(e)不同类型靶标TFs的占比;(f、g)HD-TFs(f)和bHLH-TFs(g)基于PWM的能量分数;(h)验证低亲和力ARNTL蛋白与o-NBA探针之间的直接交联。
在本期的推文中,小远先带大家回顾了研究蛋白-DNA互作的传统实验方法,包括如何寻找蛋白-DNA互作以及如何验证两者的互作关系。接着重点介绍了近些年发表的一些新技术,首先是用于在植物体内验证蛋白-DNA互作的可视化技术SRS系统和DVR检测,这两种技术相比于传统的双荧光素酶报告基因实验,在观察蛋白-DNA互作时,无需使用仪器和药品,具有实验操作简单、便捷的特点。接着介绍的实验技术主要是用来筛选蛋白-DNA互作的。其中,greenCUT&RUN技术是CUT&RUN的改进版本,它进一步提高了CUT&RUN技术的灵敏度,并已经在植物中有相关的应用案例。ChIP-DIP技术可以一次性完成数百种DNA结合蛋白的ChIP-seq,为基因调控的动态变化提供了更全面的视角。LIKE-XL技术实现了对弱和动态蛋白-DNA相互作用的全局分析。不过,ChIP-DIP和LIKE-XL这两种技术目前在植物研究领域小远并未找到相关的应用案例,大家今后如果看到了相关的应用文献欢迎留言告诉小远哦~
图12 基于o-NBA探针的竞争性交联下拉法来分析特定的DNA-TF的相互作用(Guo et al., 2023)。(a)o-NBA探针的结构和序列;(b)竞争交联下拉法的实验流程;(c)下拉组和竞争组的银染结果;(d)在两次重复实验中均富集到的蛋白质被认为是o-NBA探针的靶标结合蛋白;(e)不同类型靶标TFs的占比;(f、g)HD-TFs(f)和bHLH-TFs(g)基于PWM的能量分数;(h)验证低亲和力ARNTL蛋白与o-NBA探针之间的直接交联。
在本期的推文中,小远先带大家回顾了研究蛋白-DNA互作的传统实验方法,包括如何寻找蛋白-DNA互作以及如何验证两者的互作关系。接着重点介绍了近些年发表的一些新技术,首先是用于在植物体内验证蛋白-DNA互作的可视化技术SRS系统和DVR检测,这两种技术相比于传统的双荧光素酶报告基因实验,在观察蛋白-DNA互作时,无需使用仪器和药品,具有实验操作简单、便捷的特点。接着介绍的实验技术主要是用来筛选蛋白-DNA互作的。其中,greenCUT&RUN技术是CUT&RUN的改进版本,它进一步提高了CUT&RUN技术的灵敏度,并已经在植物中有相关的应用案例。ChIP-DIP技术可以一次性完成数百种DNA结合蛋白的ChIP-seq,为基因调控的动态变化提供了更全面的视角。LIKE-XL技术实现了对弱和动态蛋白-DNA相互作用的全局分析。不过,ChIP-DIP和LIKE-XL这两种技术目前在植物研究领域小远并未找到相关的应用案例,大家今后如果看到了相关的应用文献欢迎留言告诉小远哦~
References:
Gong W, He K, Covington M, et al. The development of protein microarrays and their applications in DNA-protein and protein-protein interaction analyses of Arabidopsis transcription factors[J]. Molecular Plant, 2008, 1(1): 27-41.
Guo AD, Wei D, Nie HJ, et al. Light-induced primary amines and o-nitrobenzyl alcohols cyclization as a versatile photoclick reaction for modular conjugation. Nature Communications, 2020, 11(1): 5472.
Guo AD, Yan KN, Hu H, et al. Spatiotemporal and global profiling of DNA-protein interactions enables discovery of low-affinity transcription factors. Nature Chemistry, 2023, 15(6): 803-814.
Hainer S J, Fazzio T G. High-resolution chromatin profiling using CUT&RUN[J]. Current protocols in molecular biology, 2019, 126(1): e85.
Hu W, Yuan Y, Wang CH, et al. Genetically Encoded Residue-Selective Photo-Crosslinker to Capture Protein-Protein Interactions in Living Cells. Chem, 2019, 5: 2955-2968.
Krizek BA. AINTEGUMENTA utilizes a mode of DNA recognition distinct from that used by proteins containing a single AP2 domain. Nucleic Acids Res[J]. Nucleic Acids Research, 2003 31(7): 1859-68.
Koidl S, Timmers H T M. greenCUT&RUN: Efficient genomic profiling of GFP-tagged transcription factors and chromatin regulators[J]. Current protocols, 2021, 1(10): e266.
Lin R, Ding L, Casola C, et al. Transposase-derived transcription factors regulate light signaling in Arabidopsis[J]. Science, 2007, 318(5854): 1302-1305.
Meers M P, Bryson T D, Henikoff J G, et al. Improved CUT&RUN chromatin profiling tools[J]. eLife, 2019, 8: e46314.
Nole-Wilson S, Krizek BA. DNA binding properties of the Arabidopsis floral development protein AINTEGUMENTA[J]. Nucleic Acids Research, 2000, 28(21): 4076-82.
Powers J, Zhang X, Reyes A V, et al. Next-generation mapping of the salicylic acid signaling hub and transcriptional cascade[J]. Molecular plant, 2024, 17(10): 1558-1572.
Perez AA, Goronzy IN, Blanco MR, et al. ChIP-DIP maps binding of hundreds of proteins to DNA simultaneously and identifies diverse gene regulatory elements. Nature Genetics, 2024 56(12): 2827-2841.
Sun H, Wang S, Yang K, et al. Development of dual-visible reporter assays to determine the DNA-protein interaction[J]. The Plant Journal, 2023, 113(5): 1095-1101.
Zhou Q, Ye Y, He H, et al. SRS: An intelligent and robust approach for confirmation of plant transcription factor-DNA interactions[J]. Plant Biotechnology Journal, 2024, 23(1): 198.














