近日,Plant Biotechnology Journal 杂志在线发表了由北京林业大学孙丽丹课题组题为“Haplotype-resolved genome assembly provides new insights into the genomic origin of purple colour in Prunus mume”论文。该研究工作利用Pacbio、Hi-C等测序技术,完成了‘美人’梅单倍型基因组构建,通过比较基因组学在单倍型C(Haplotype C)亚基因组上鉴定到一个约1.8 Mb的倒位(INV)变异,利用群体遗传学分析揭示INV与紫色性状共分离,并在INV的远端断点发现了PmMYB10.5b转录因子。利用梅花毛状根遗传转化、双荧光素酶和EMSA等实验验证PmMYB10.5b通过与花青素合成等位基因启动子结合,促进花青素积累,解析了‘美人’梅紫色性状的基因组起源与分子调控机制,为观赏木本植物彩色性状的遗传改良和分子育种工作提供重要的理论依据。
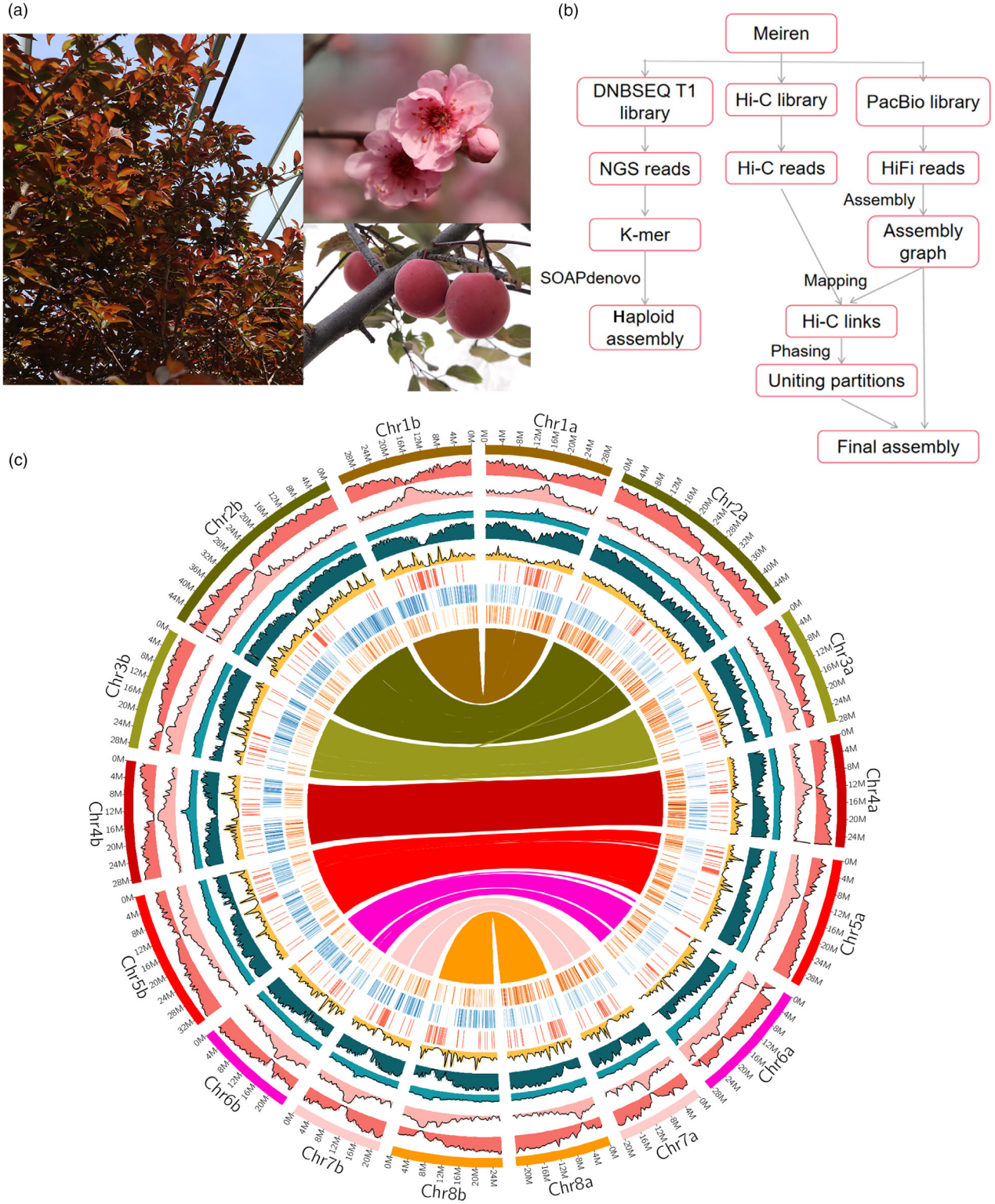
研究团队通过Pacbio和Hi-C等测序技术,利用Hifiasm软件,构建‘美人’梅高质量的单倍型基因组(图1a,b)。该基因组大小为499.47 Mb,其中来源于梅花HaplotypeM(HM)为250.66 Mb,来源于李子HaplotypeC(HC)为248.79 Mb,约95.42%的序列锚定到16条染色体(图1c)。通过近缘物种的同源注释、ab initio基因预测和‘美人’梅叶、茎、花不同发育时期的全长转录组等方法在基因组共注释到了59980个蛋白质编码基因,利用MCScanX和blastp软件,通过共线性分析和序列比对鉴定了42985个等位基因,其中HM亚基因组和HC亚基因组分别有21571个和21414个。通过比较基因组学分析,发现‘美人’梅的单倍型HM和HC分别来自梅花和李,且在杂交过程中几乎不存在染色体重排事件。
图1‘美人’梅基因组组装和单倍型染色体构建
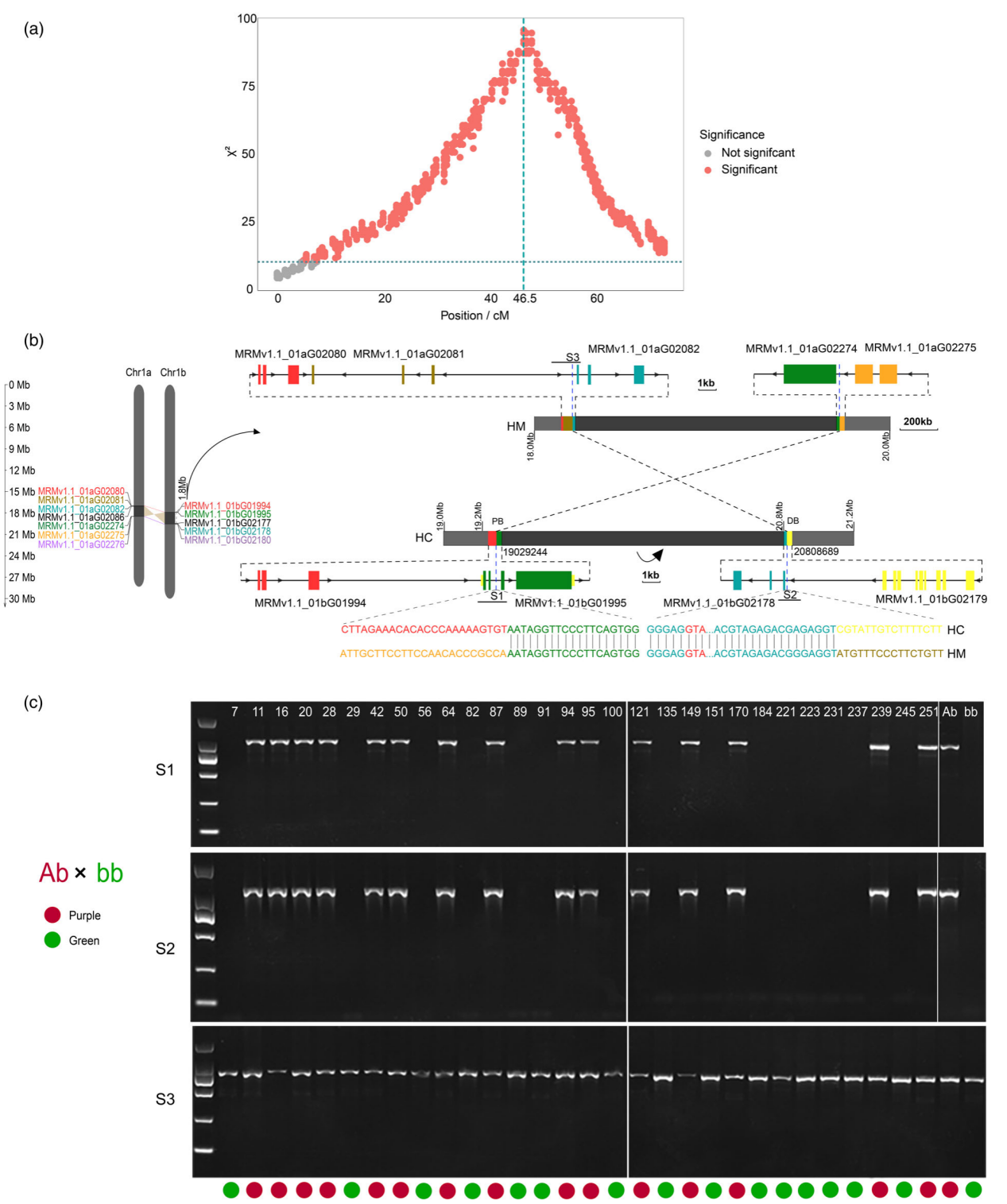
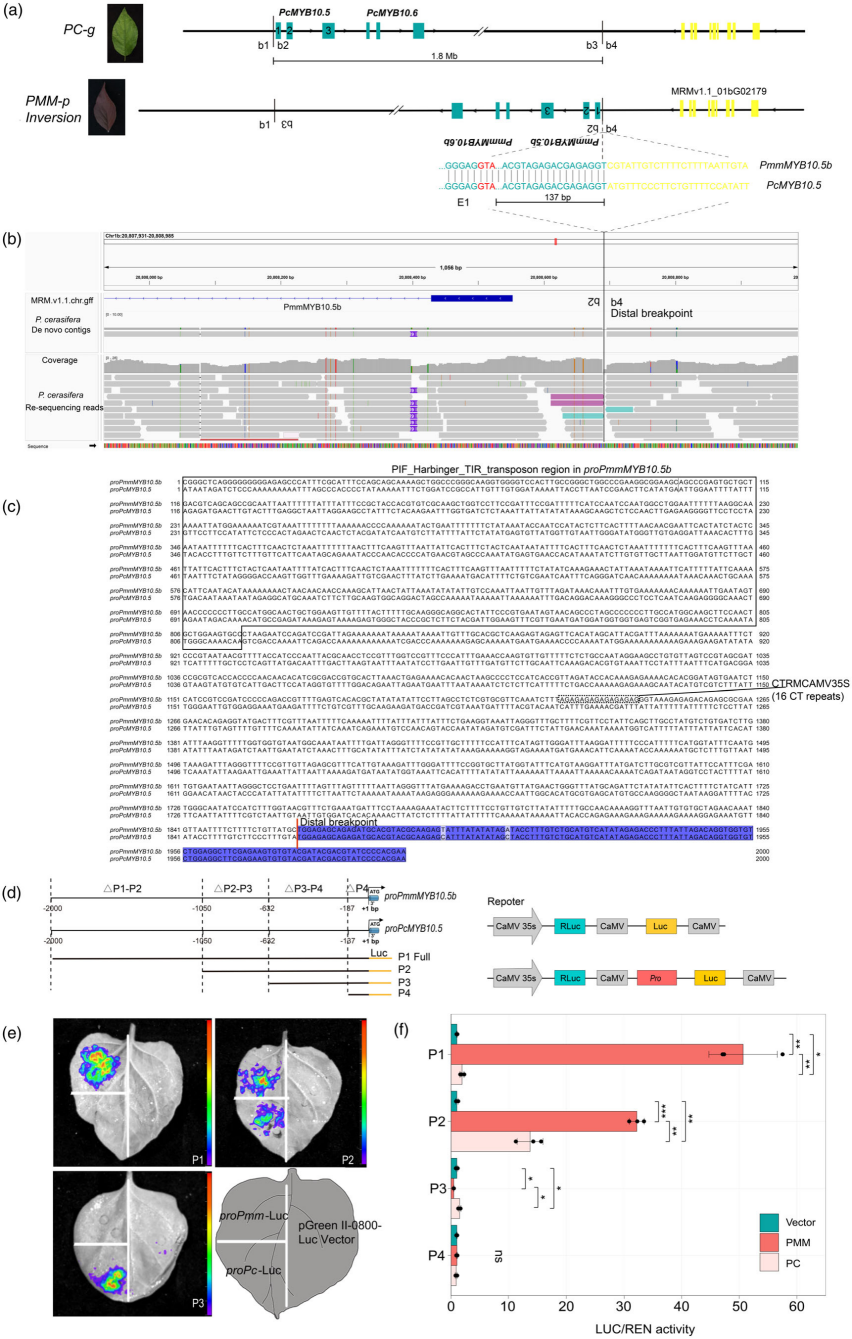
研究团队利用比较基因组学分析,在HC单倍型1号染色体上发现一个1.8Mb的大片段INV变异。通过与亲本基因组共线性分析,发现该INV存在于亲本紫叶李。为深入探究INV与紫色性状的关联,研究人员构建了以‘美人’梅为母本,‘香雪宫粉’为父本的F1杂交群体,对子代个体进行重测序,通过QTL定位分析发现INV与紫色性状紧密相关(图2a)。在INV断点处设计特异性引物,利用杂交子代群体进行PCR扩增,并将其重测序数据回帖至单倍型基因组,结果发现含有INV的个体呈紫色,不含则为绿色。以上研究结果均证明INV与‘美人’梅紫色性状完全共分离(图2b,c)。
图2 1.8 Mb大片段染色体倒位与紫色性状完全共分离
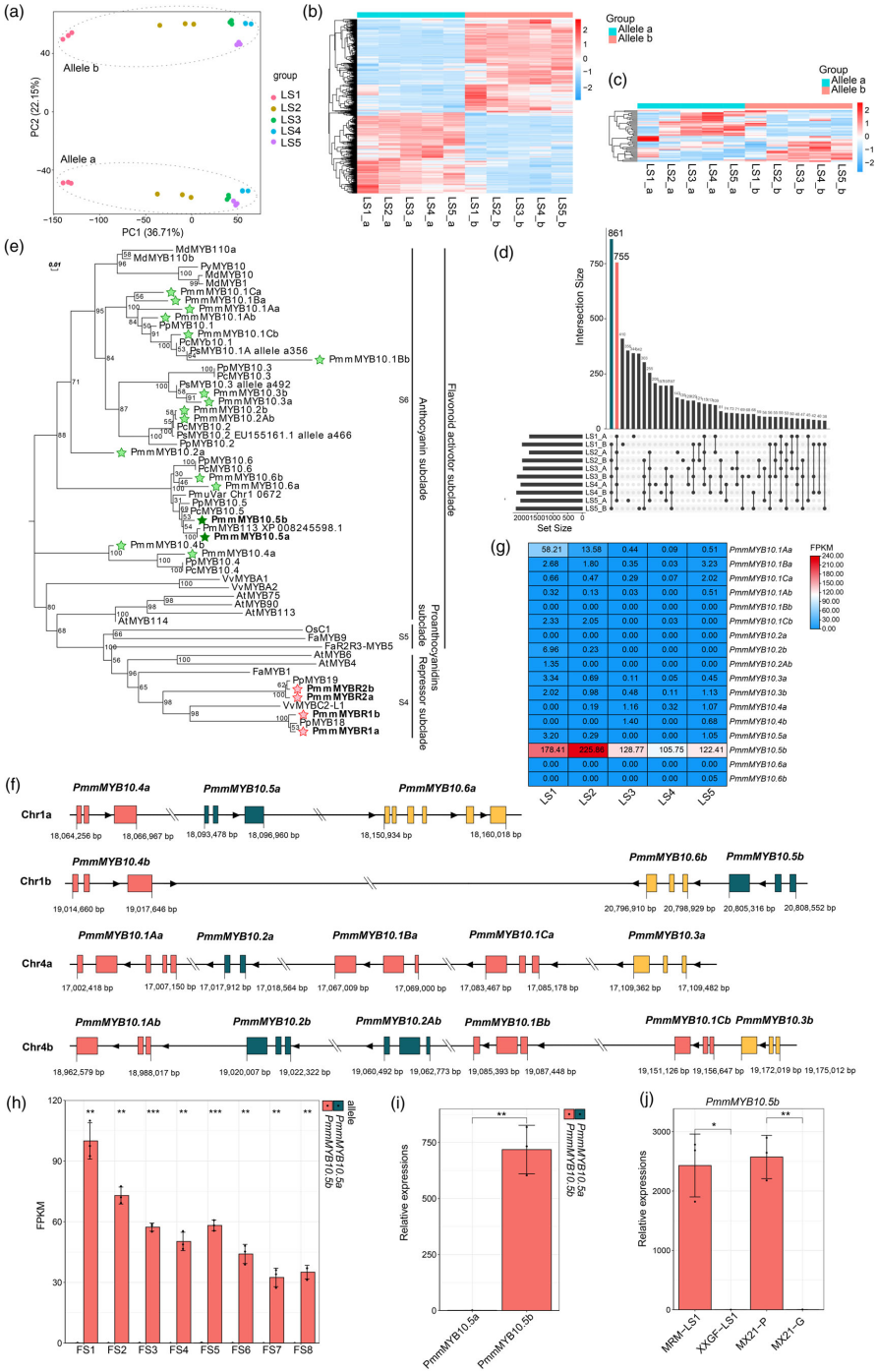
基于单倍型基因组,研究团队选择了叶片和果实发育不同阶段探究了‘美人’梅的 ASE模式。‘美人’梅HC亚基因组中ASE基因数量均高于HM亚基因组,存在微弱的ASE不平衡现象。为了明确ASE对性状的影响,开展了一致性和非一致性ASE的分析,发现在叶片和果实中一致性ASE基因数量均多于非一致性ASE基因。其中,叶片中共有1616对一致性ASE基因,HC-biased 861个,HM-biased 755个,这些一致性ASE基因在黄酮类化合物生物合成等多种代谢过程中功能富集,可能参与调控‘美人’梅紫色性状的的形成(图3a-d)。本研究对位于INV及侧翼的区域基因进行了注释,在INV断点处发现了MYBs簇共包含6个等位基因,与AtMYB114和PpMYB10基因同源。通过系统进化分析,发现该基因簇为花青素合成调控关键分支S6(图3e)。在‘美人’梅基因组中共鉴定到17个MYB10同源基因,仅MRMv1.1_01bG02178(命名为PmmMYB10.5b)是位于INV远端断点的基因且表现为一致性ASE,在叶和果中高表达(图3f-j),推测该基因是‘美人’梅紫色性状形成的核心激活基因。
图3PmmMYB10.5b是花青素生物合成的核心激活子
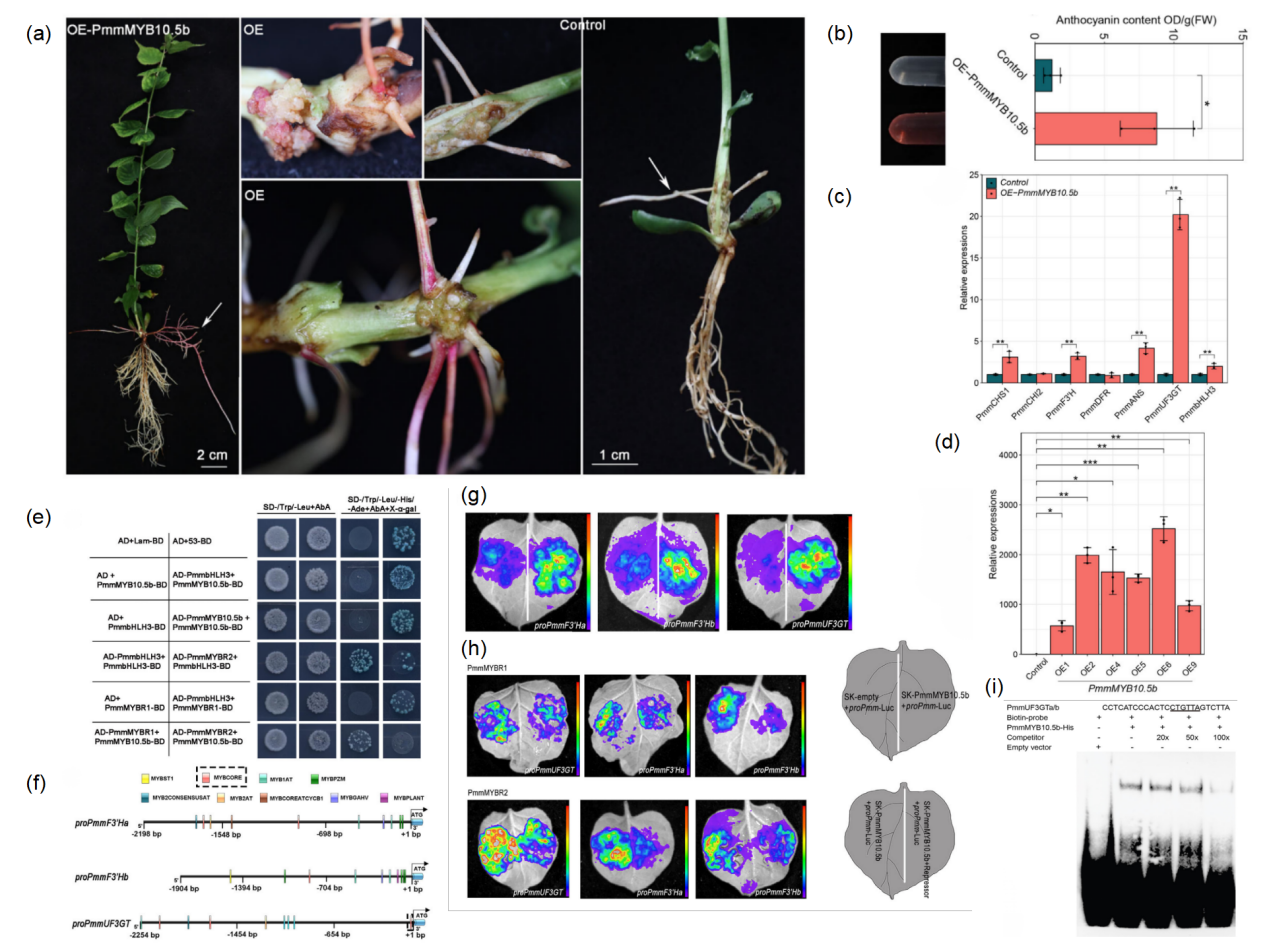
为了验证PmmMYB10.5b基因的调控功能,首先在烟草叶片中瞬时过表达PmmMYB10.5a/b两个等位基因,发现均有花青素的大量积累,说明PmmMYB10.5基因编码序列不存在功能差异。其次,梅花毛状根遗传转化和拟南芥过表达实验表明PmmMYB10.5b能够促进花青素合成积累(图4a-d)。再者,通过Y2H实验发现PmmMYB10.5b与PmmbHLH3相互作用,形成蛋白复合体发挥功能(图4e)。最后,利用双荧光素酶和EMSA等实验发现PmmMYB10.5b可直接靶向结合花青素合成等位基因启动子,激活其表达,促进花青素的合成积累(图4f-i)。
图4. PmmMYB10.5b基因促进花青素生物合成积累
为确定‘美人’梅紫色性状的基因组起源,研究团队选择野生樱桃李等李属绿色叶物种的重测序数据比对到‘美人’梅HC亚基因,判断INV是否存在。分析发现这些物种均不存在INV,尤其是紫叶李的原种樱桃李中也不存在INV,推测其在紫叶李形成时产生。为了探究INV如何改变MYB10.5基因的启动子,对樱桃李的重测序数据进行了组装,获得了INV附近的contigs序列,与‘美人’梅HC亚基因组比对发现INV改变了MYB10.5启动子序列(图5a,b)。为了探究INV如何激活MYB10.5基因表达,本研究分析了倒位前后樱桃李和‘美人’梅HC中MYB10.5的启动子序列差异(图5c),并利用LUC报告基因检测了倒位前后MYB10.5的启动子激活活性,发现倒位后启动子活性显著增强,引起其表达激活,导致花青素的生物合成(图5d-f)。
图5. 源自紫叶李的INV导致PmmMYB10.5b的表达激活
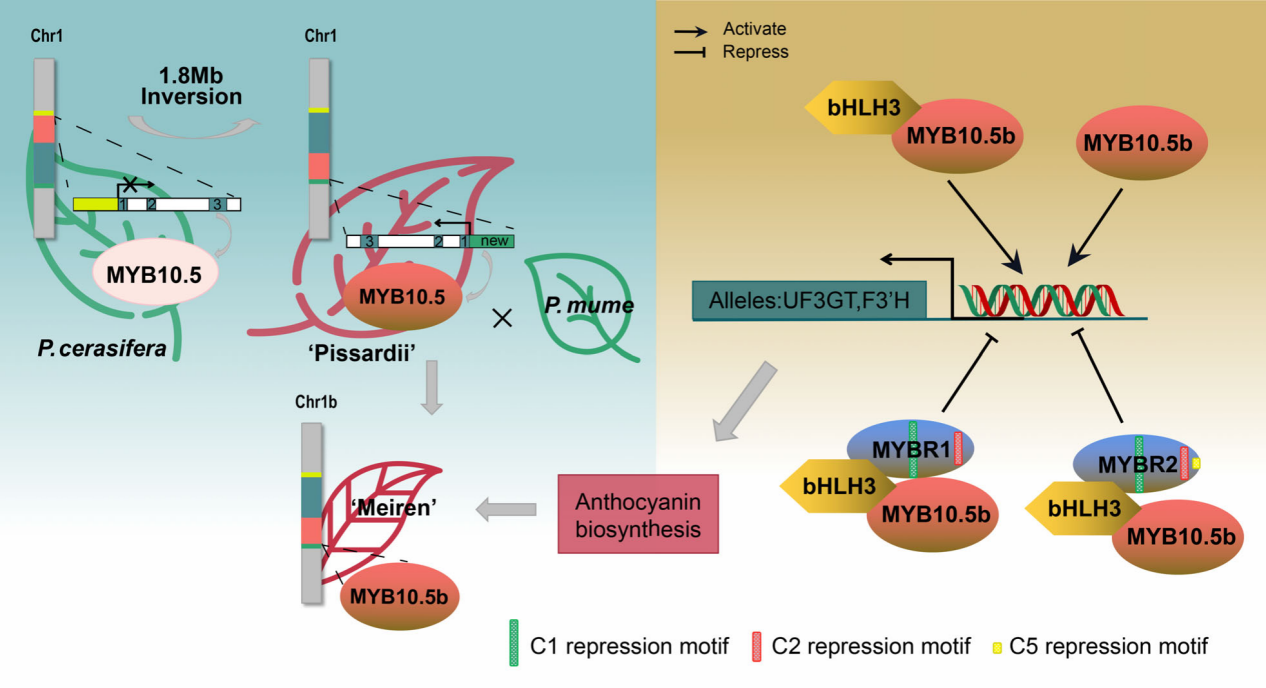
图6. 紫色叶形成的基因组起源与调控模式
http://doi.org/10.1111/pbi.14595
Dai D, Chen J, Du C, Liang M, Wu M, Mou T, Zhang H and Ma L (2022) A 2-Mb Chromosome Inversion Interrupted Transcription of LAX2-4 and Generated Pleiotropic Phenotypes in Rice. J PLANT GROWTH REGUL. 41,2328-2337.
Fang ZZ, Lin-Wang K, Zhou DR, Lin YJ, Jiang CC, Pan SL, Espley RV, Andre CM and Ye XF (2021) Activation of PsMYB10.2 Transcription Causes Anthocyanin Accumulation in Flesh of the Red-Fleshed Mutant of ‘Sanyueli’ (Prunus salicina Lindl.). FRONT PLANT SCI. 12,680469.
Fiol A, García-Gómez BE, Jurado-Ruiz F, Alexiou K, Howad W and Aranzana MJ (2021) Characterization of japanese plum (Prunus salicina) PsMYB10 alleles reveals structural variation and polymorphisms correlating with fruit skin color. FRONT PLANT SCI. 12.
Hughes NM and Lev-Yadun S (2023) Review: Why do some plants have leaves with red or purple undersides? ENVIRON EXP BOT. 205,105126.
Jaakola L (2013) New insights into the regulation of anthocyanin biosynthesis in fruits. TRENDS PLANT SCI. 18,477-483.
Surmacz B (2023) Spatial patterns of flower colour variation in native and introduced ranges of Convolvulus arvensis (Convolvulaceae) revealed by citizen-science data and machine learning. Plant Biol (Stuttg). 25,681-686.
Xu W, Grain D, Bobet S, Le Gourrierec J, Thevenin J, Kelemen Z, Lepiniec L and Dubos C (2014) Complexity and robustness of the flavonoid transcriptional regulatory network revealed by comprehensive analyses of MYB-bHLH-WDR complexes and their targets in Arabidopsis seed. NEW PHYTOL. 202,132-144.
Zhou Y, Zhou H, Lin-Wang K, Vimolmangkang S, Espley RV, Wang L, Allan AC and Han Y (2014) Transcriptome analysis and transient transformation suggest an ancient duplicated MYB transcription factor as a candidate gene for leaf red coloration in peach. BMC PLANT BIOL. 14,388.
Zhou H, Ma R, Gao L, Zhang J, Zhang A, Zhang X, Ren F, Zhang W, Liao L, Yang Q, Xu S, Otieno Ogutu C, Zhao J, Yu M, Jiang Q, Korban SS and Han Y (2021) A 1.7‐Mb chromosomal inversion downstream of a PpOFP1 gene is responsible for flat fruit shape in peach. PLANT BIOTECHNOL J. 19,192-205.
图文来源:植物生物技术Pbj