本文内容速览:
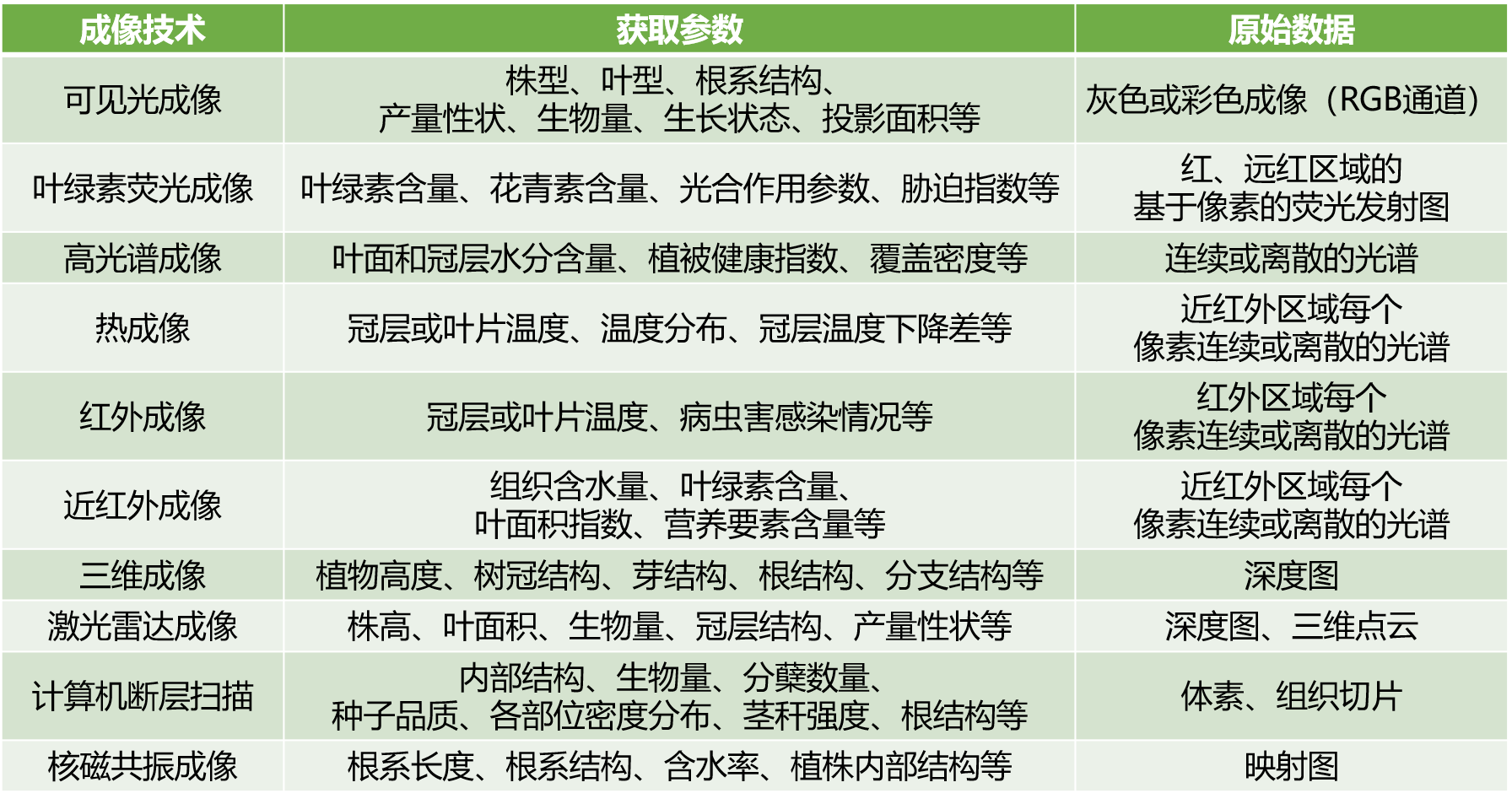
图像采集技术按照图像的性质可分为二维成像技术和三维成像技术,不同的图像采集技术对应采集不同的表型参数,在实践中需要考虑应用场景及成本等因素来选择合适的成像技术,表1列举了常用的图像成像技术及常用的参数(杨文庆等, 2022)。
表1 常用的植物表型成像技术,改编自(杨文庆等, 2022)。
其中三维成像技术由于能够更直接地测量植物的形态,因而得到了广泛应用。在表征对象的三维结构时,主要有以下两种方法:主动方法使用诸如激光雷达之类的主动传感器来直接捕获三维点云,该点云代表植物在三维空间中每个部分的坐标;被动方法使用被动传感器(相机等),从多角度采集二维图像来推导出三维点云。
在对植物的三维点云进行分析时,一个必要的步骤是将点云转换为几何图形,进而提取植物器官的几何特征。2021年4月,庆应义塾大学的Ryuhei Ando、东京电机大学的Yuko Ozasa和东京大学的Wei Guo在Plant Phenomics杂志上发表了一篇题为“Robust Surface Reconstruction of Plant Leaves from 3D Point Clouds”的研究论文,该研究开发了一种叶片表面重建的方法。利用大豆和甜菜进行该方法性能评估,发现能够成功采集到三维点云并对叶片表面进行重建(图1)。
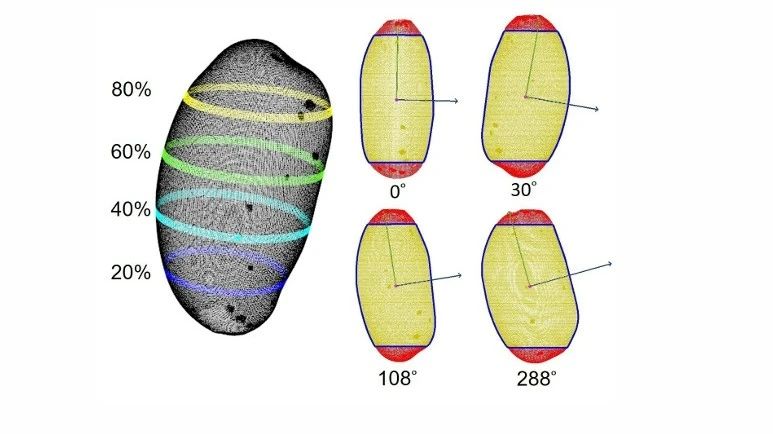
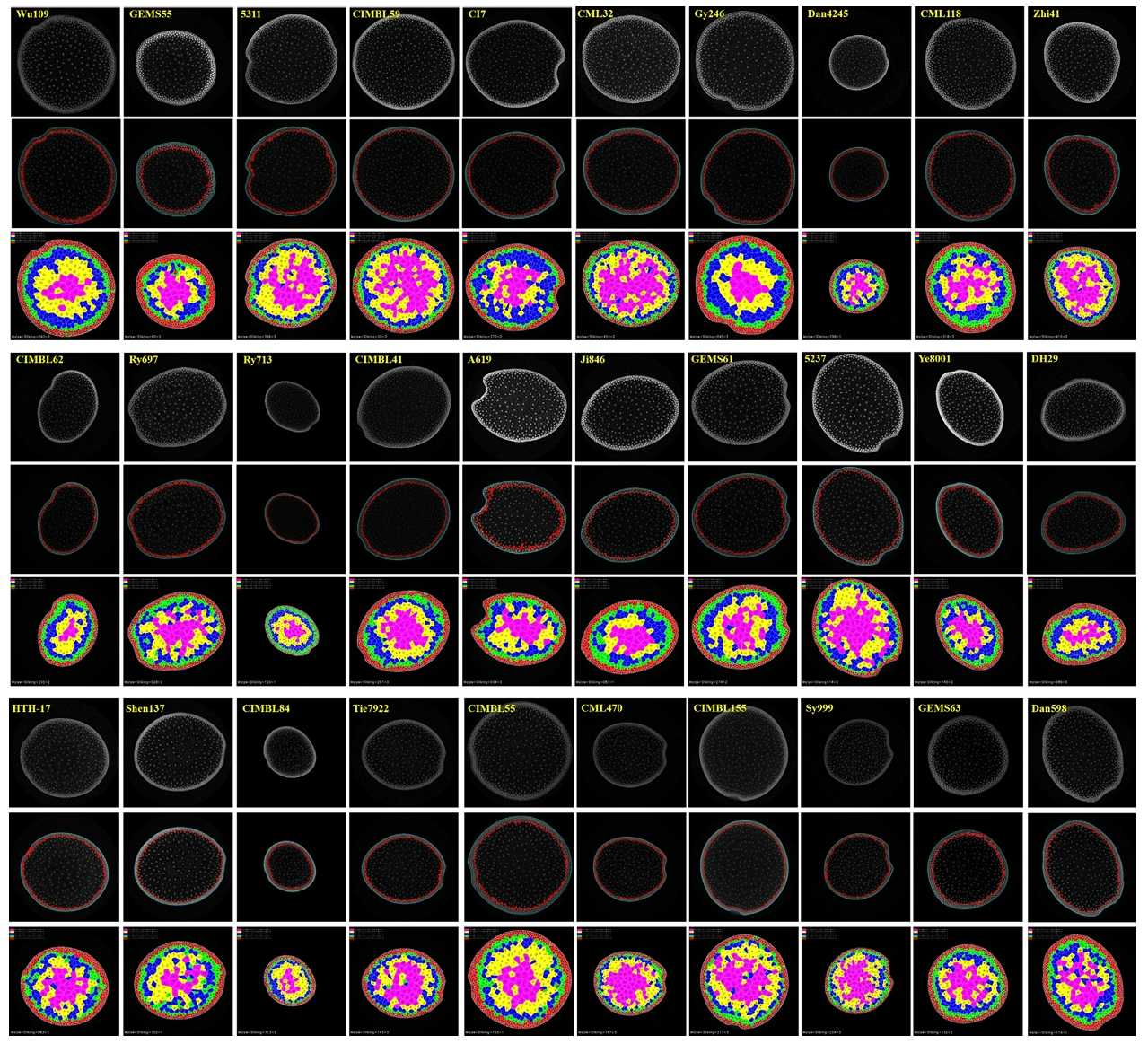
通过对6个马铃薯品种的6个形状一致性的相关性状及其形状指数进行了测定,证明了三维图像分析技术可以高精度地估计土豆芽眼的数量和深度。此外,3个形状一致性特征可以为区分品种提供有力的支撑。
图2 点云生成的马铃薯块茎表面的三维网格(a)和标记识别芽眼(b)(Liu et al., 2021)。
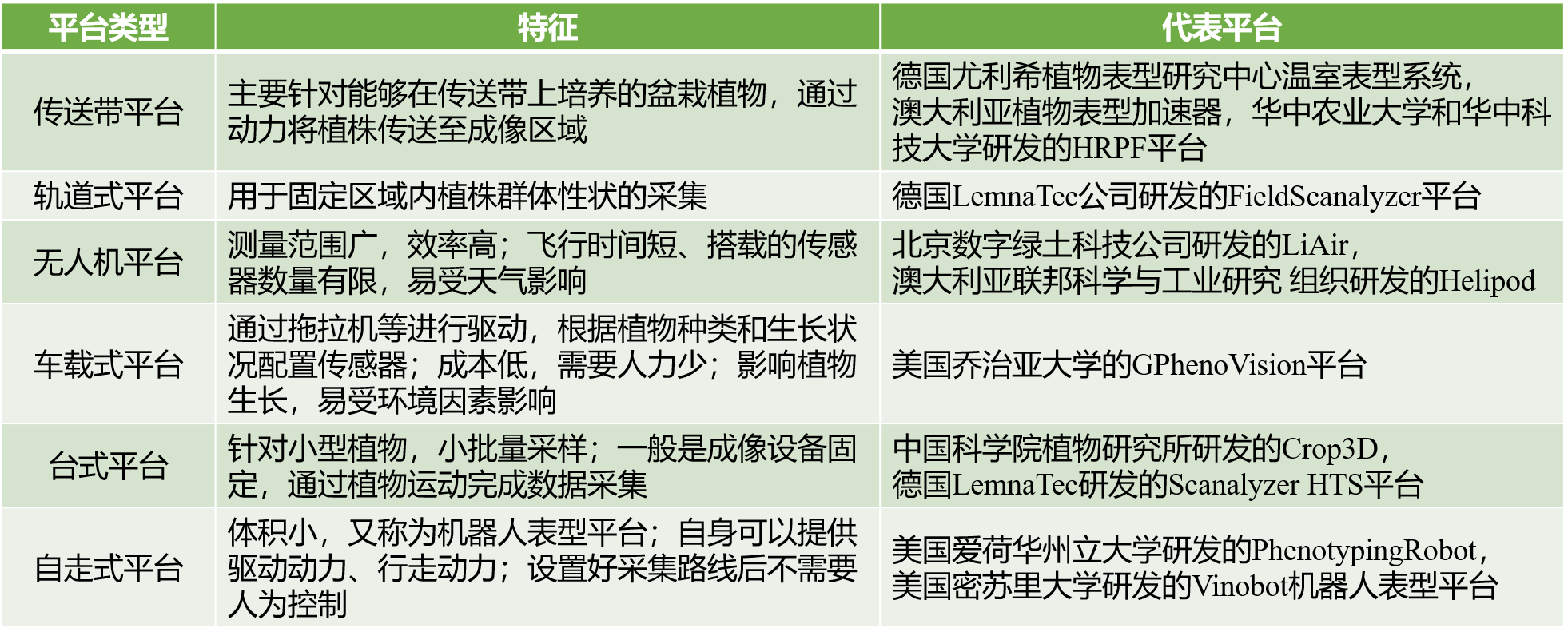
植物表型采集平台主要包括田间型和温室型。田间作物位置固定,一般通过移动传感器的方式获取表型信息,常用的平台包括车载式平台、自走式平台、无人机平台以及飞机、卫星等。传送带平台和轨道式平台是比较常见的温室表型采集平台,传送带平台通过传送带将作物传送到成像区域成像,轨道式平台通过移动传感器实现在作物原位置上采集表型数据。表2给大家汇总了不同平台类型的特征。
表2 常见植物表型信息采集平台,改编自(杨文庆等, 2022)。
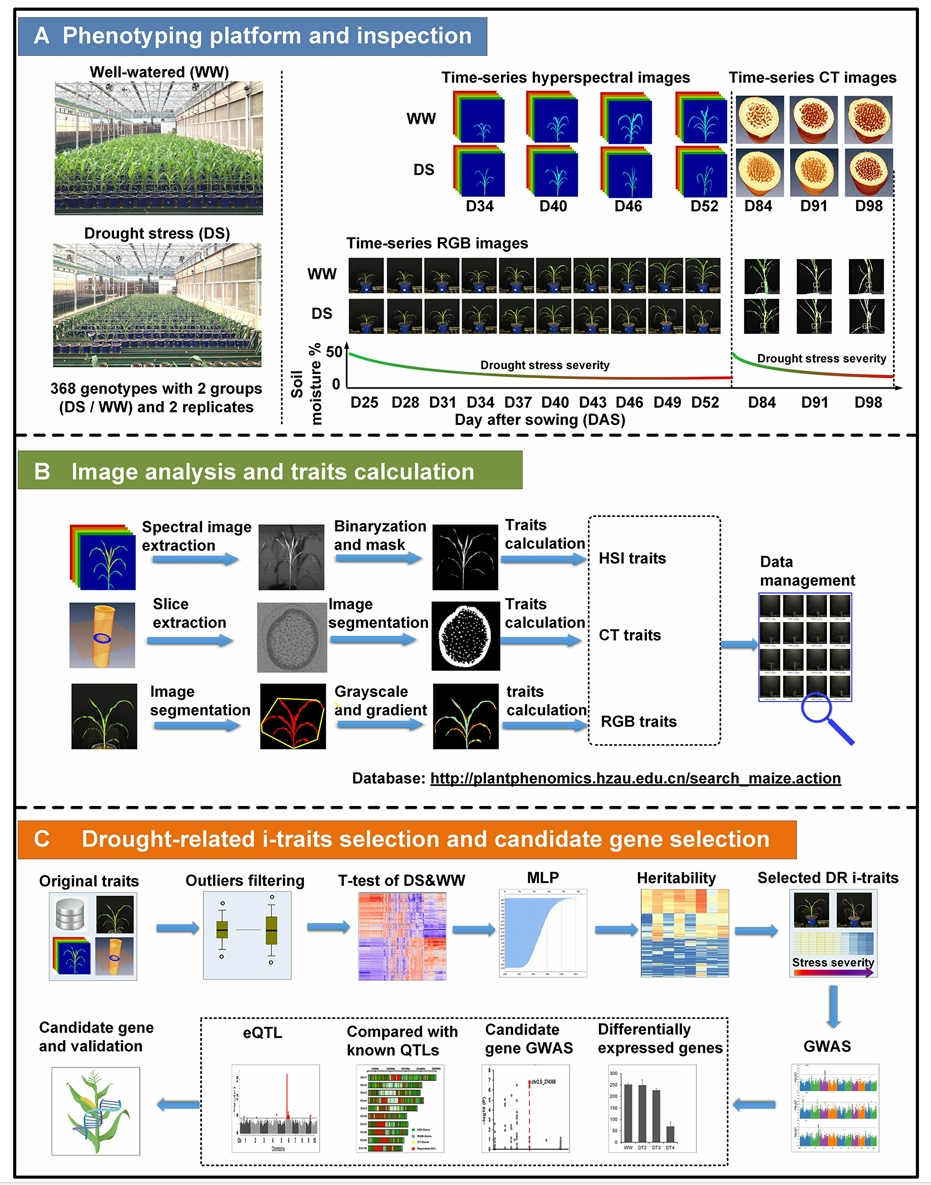
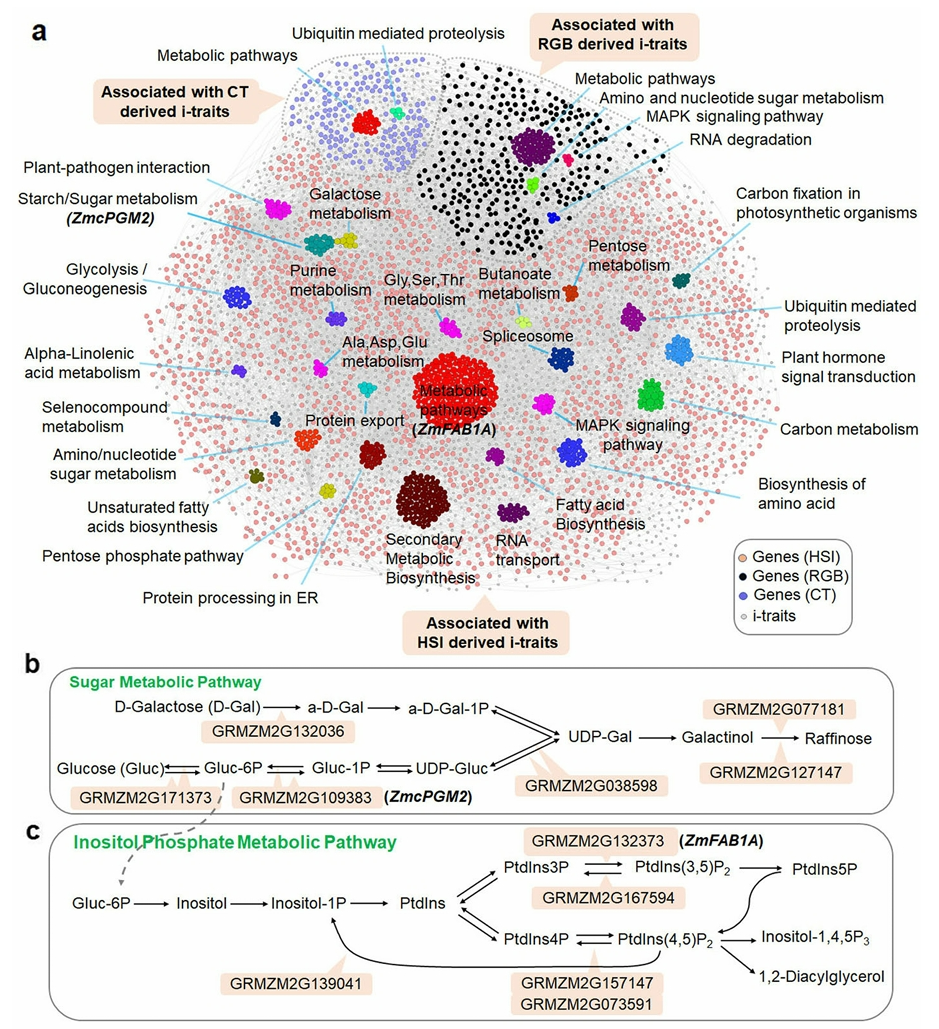
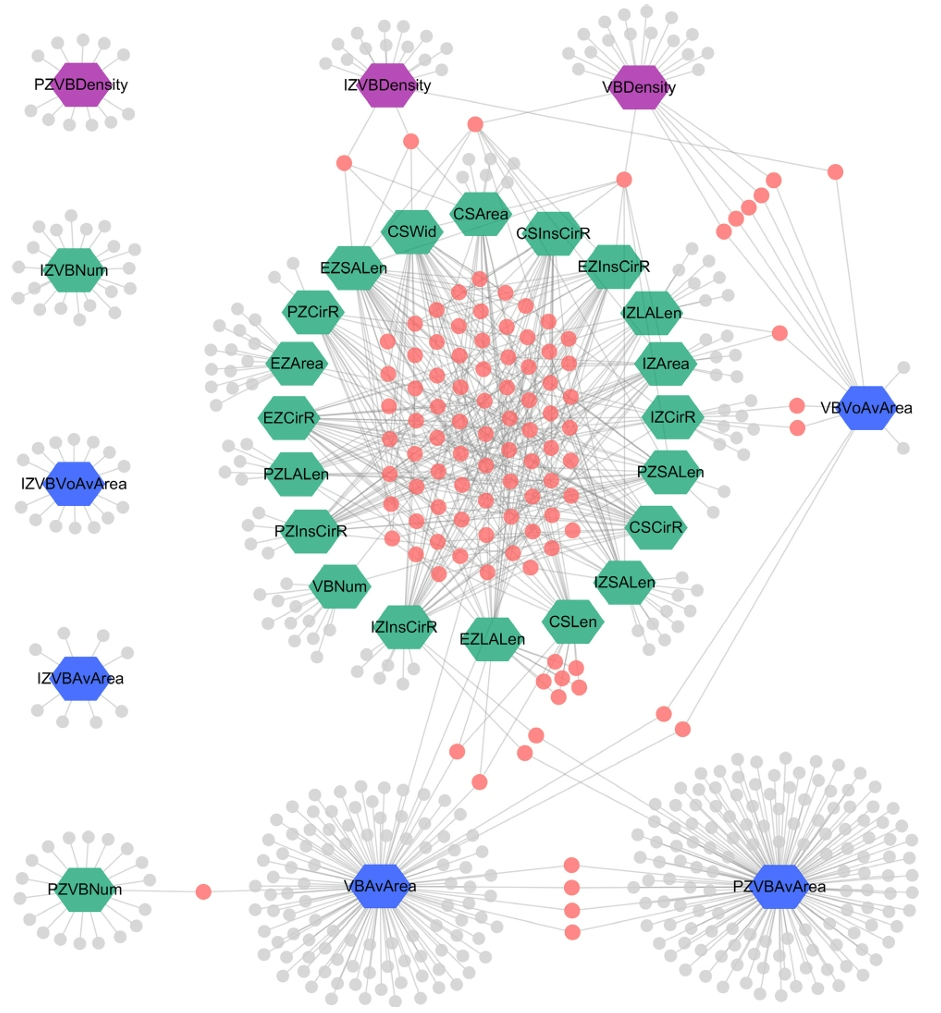
依托以上植物表型信息采集平台,众多科研发现已经进入我们的视野,为研究工作带来了新的启示和方向。2021年6月,华中农业大学作物遗传改良全国重点实验室代明球课题组联合杨万能课题组在Genome Biology杂志上发表了一篇题为“Using high-throughput multiple optical phenotyping to decipher the genetic architecture of maize drought tolerance”的研究论文。该研究就是利用华中农业大学和华中科技大学合作开发的HRPF表型平台,并结合高光谱、微型CT及RGB多光学成像技术对368份玉米自然群体材料在多个生长时期、正常浇水和干旱胁迫下的表型进行连续无损检测,获得了丰富的与干旱胁迫响应相关的图像性状(图4),结合全基因组关联(GWAS)分析鉴定到大量与干旱胁迫相关的候选基因和QTLs,构建了基因-表型关联网络(图5)。
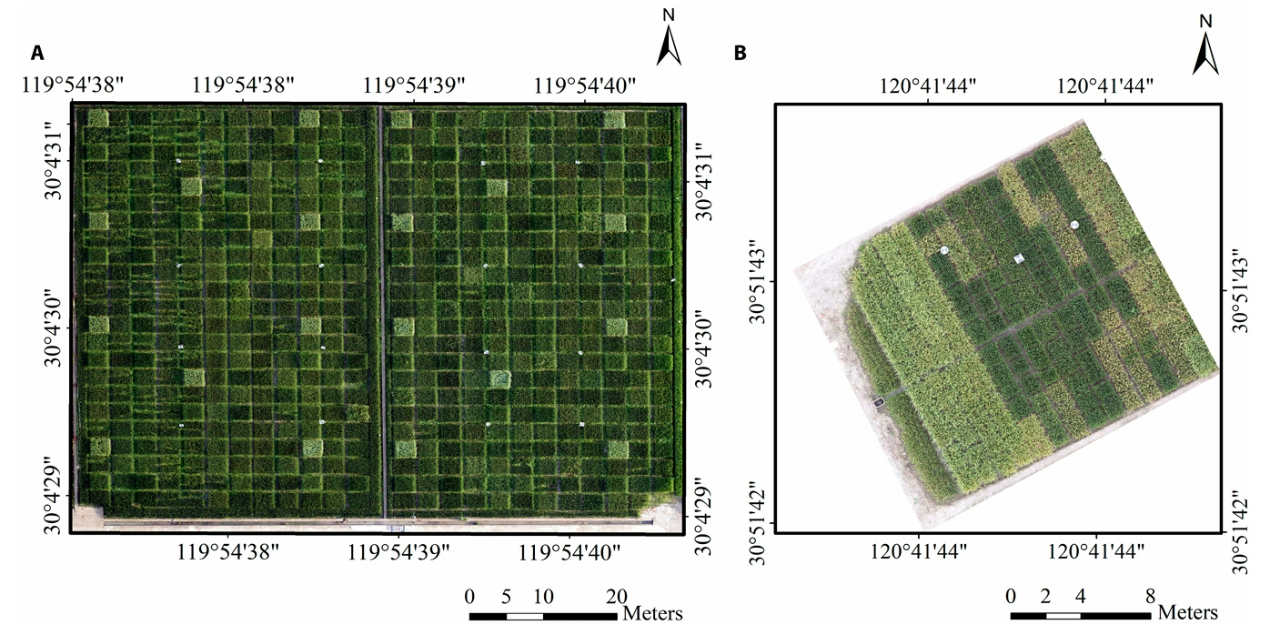
2024年10月,浙江大学生物系统工程与食品科学学院岑海燕课题组在Plant Phenomics杂志上发表了一篇题为“Phenotyping of Panicle Number and Shape in Rice Breeding Materials Based on Unmanned Aerial Vehicle Imager”的研究论文。研究团队对以往的无人机平台进行改良,开发出了一种基于无人机图像的全自动化、高通量的田块分割和稻穗计数方法,并且能够同时对不同稻穗进行弯曲度分类,为育种学家提供数字化的稻穗表型信息。
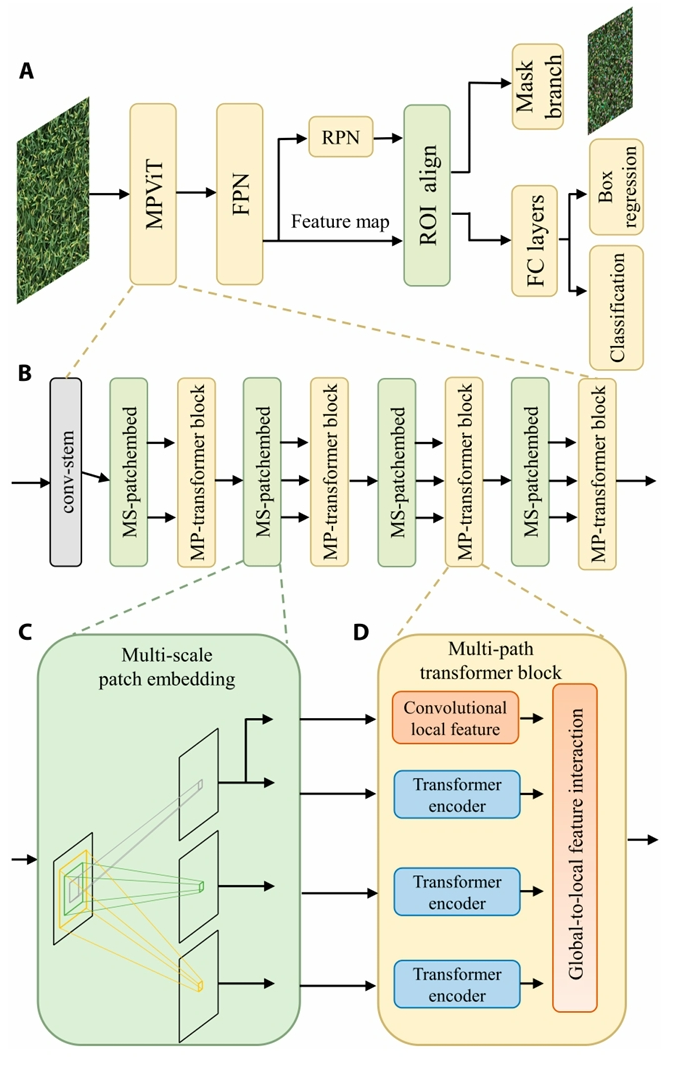
研究团队在15m的高度拍摄水稻冠层图像(图6),图像拼接后使用基于Mask R-CNN的田块边界自动识别算法Plot-Seg将大田图像分割成小区尺度的子图(图7),并根据齐穗-成熟时间将其分为3个生长阶段,而后根据集成多路径Transformer的稻穗识别网络Panicle-ViT准确检测单位面积稻穗数目(图8)。
图6 位于中国浙江省杭州市中国水稻研究所,水稻播种后98天(A)和中国浙江省嘉兴市农业科学院,水稻播种后104天(B)的水稻冠层图像(Lu et al., 2024)。
图7 小区田块分割流程图,通过 Mask R-CNN在农业遥感图像中进行批次分割(Lu et al., 2024)。
2020年6月,北京市农林科学院赵春江课题组联合郭新宇课题组在Plant Biotechnology Journal杂志上发表了一篇题为“Dissecting the phenotypic components and genetic architecture of maize stem vascular bundles using high-throughput phenotypic analysis”的研究论文。该研究利用显微电子计算机断层扫描(CT)表型技术结合GWAS分析,在玉米中鉴定到调控维管束数目、维管束分布密度等性状的特异候选基因。
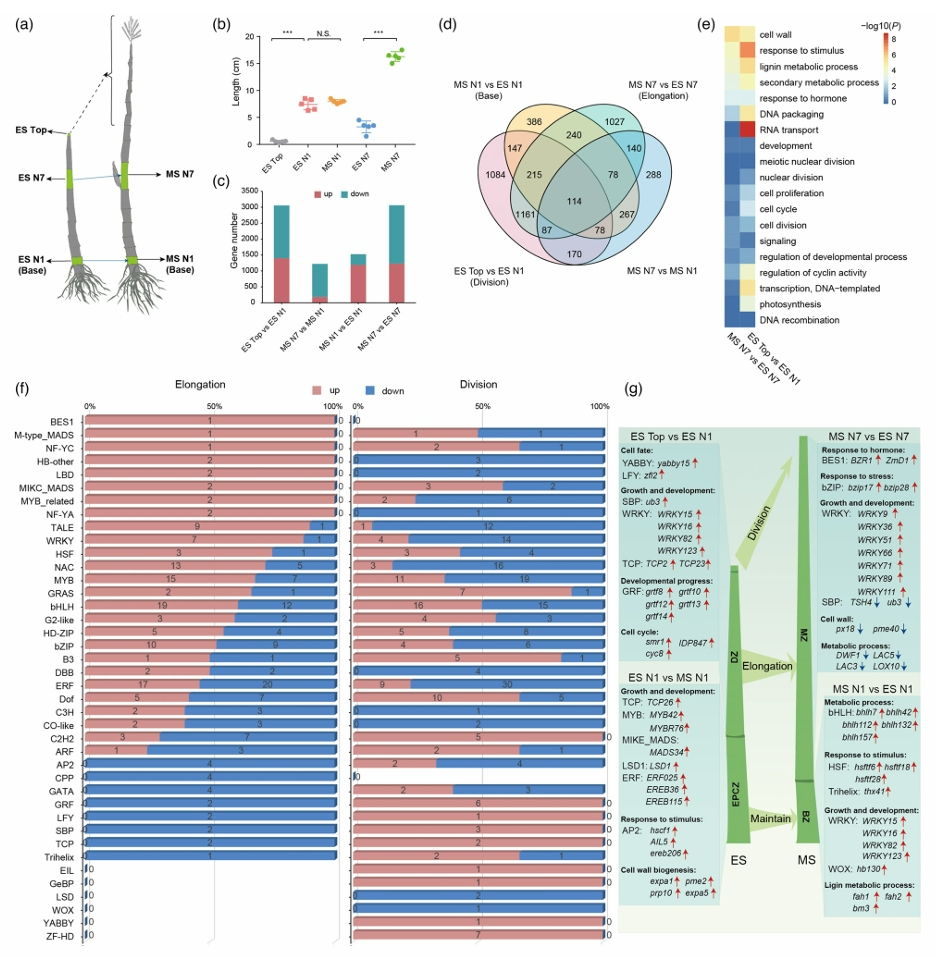
为了鉴定玉米快速伸长期的关键调节基因,研究团队选择5个代表性的节间进行深入的差异基因表达分析,分别鉴定了分生阶段和延长阶段的关键转录因子,例如BES1、NF-YC在节间延长阶段表达上调等。进一步结合节间长、直径、周长和重量等数据进行WGCNA分析与表型预测,构建了第一个玉米时空特异全节间基因表达调控网络,鉴定了一批与表型高度相关的基因,主要参与激素调控、信号转导等生物学过程,预示着它们在玉米的株型改良育种中可能具有重要的应用价值,可作为株高形成相关的潜在生物标记。
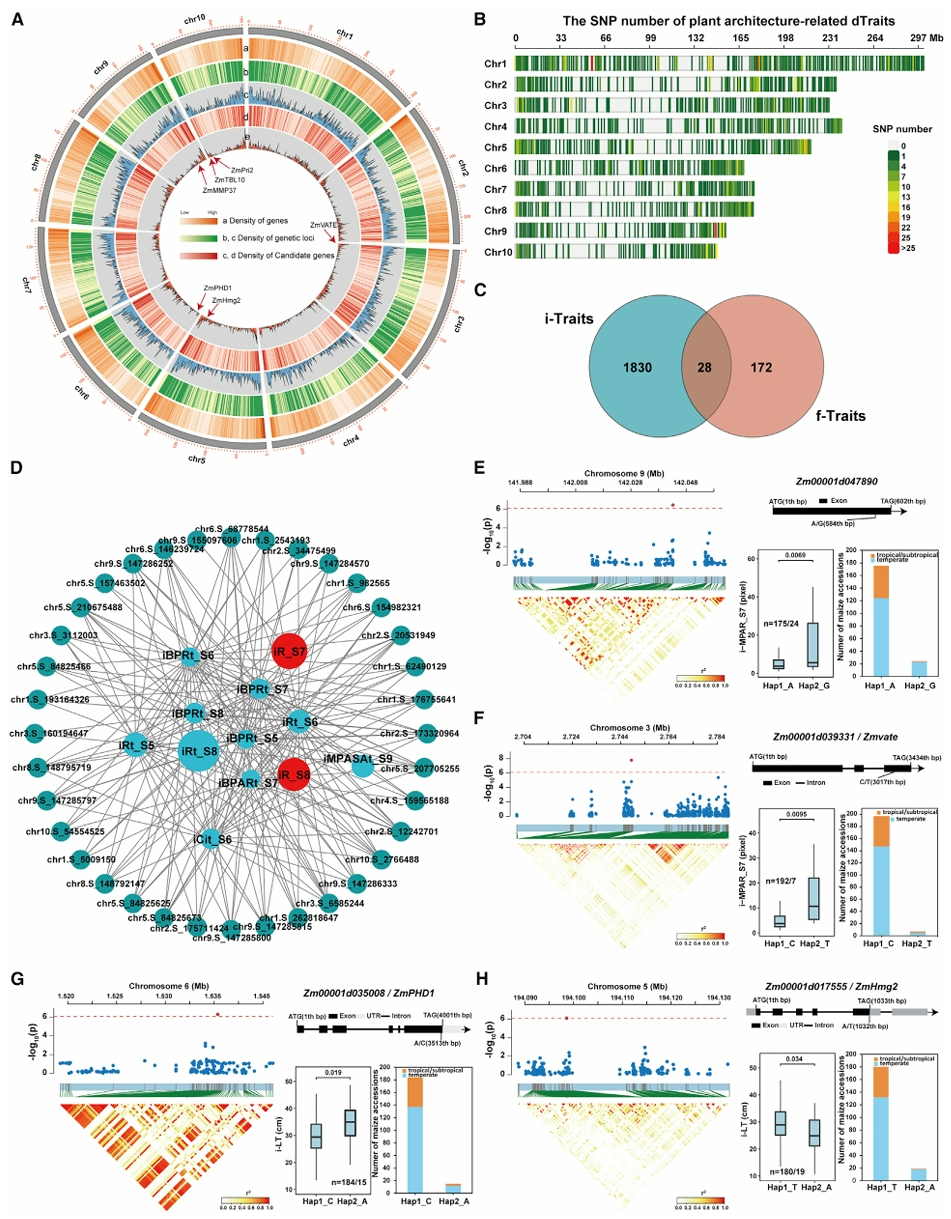
作者首先搭建了玉米高通量表型组平台(HTP),并对228份玉米自然群体材料全生育期株高相关性状变化进行动态检测,共获得了24.4万张图像,通过自主研发的图像处理程序提取出77个图像性状i-traits(图12),其中包括49个形态相关性状、20个生物量相关性状和8个生长速率相关性状。
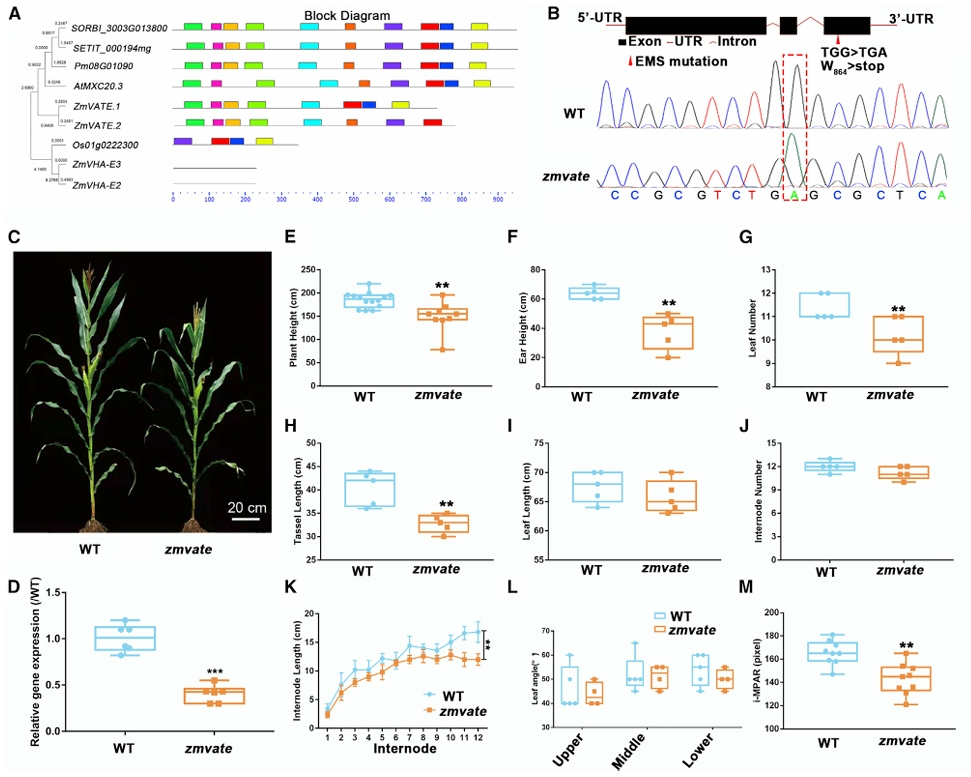
图14 ZmVATE突变导致玉米矮化(Wang et al., 2023)。
References:
周济, Tardieu F, Pridmore T等.植物表型组学:发展、现状与挑战[J].南京农业大学学报, 2018,41(4): 580-588.
杨文庆, 刘天霞, 唐兴萍,等.智慧农业背景下的植物表型组学研究进展[J].河南农业科学,2022,51(7): 1-12.
胡伟娟, 凌宏清, 傅向东.植物表型组学研究平台建设及技术应用[J].遗传, 2019,41(11): 1060-1066.
Ryuhei Ando, Yuko Ozasa, Wei Guo. Robust Surface Reconstruction of Plant Leaves from 3D Point Clouds. Plant Phenomics. 2021.
Liu J., Xu X., Liu Y., et al. Quantitative potato tuber phenotyping by 3D imaging[J]. Biosystems Engineering, 2021, 210(6):48-59.
Wu X, Feng H, Wu D, et al. Using high-throughput multiple optical phenotyping to decipher the genetic architecture of maize drought tolerance. Genome Biol. 2021;22(1):185.
Lu X, Shen Y, Xie J, et al. Phenotyping of Panicle Number and Shape in Rice Breeding Materials Based on Unmanned Aerial Vehicle Imagery. Plant Phenomics. 2024;6:0265.
Zhang Y, Wang J, Du J, et al. Dissecting the phenotypic components and genetic architecture of maize stem vascular bundles using high-throughput phenotypic analysis. Plant Biotechnol J. 2021;19(1):35-50.
Wang W, Guo W, Le L, et al. Integration of high-throughput phenotyping, GWAS, and predictive models reveals the genetic architecture of plant height in maize. Mol Plant. 2023;16(2):354-373.
Le L, Guo W, Du D, et al. A spatiotemporal transcriptomic network dynamically modulates stalk development in maize. Plant Biotechnol J. 2022;20(12):2313-2331.
图文来源:伯远生物公众号