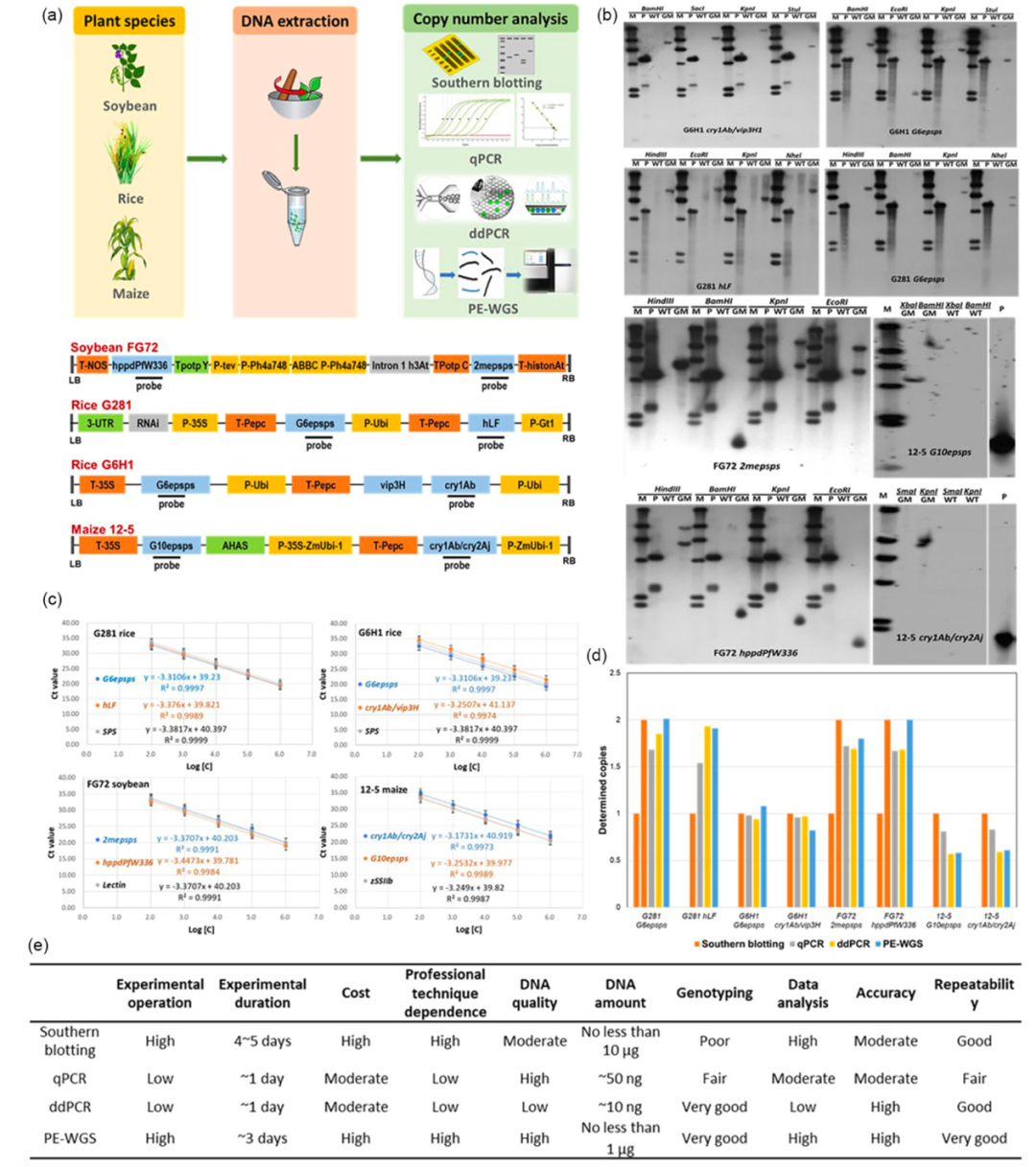
现代农业生产中,转基因和基因组编辑技术的应用为生物育种产业带来了革命性的进步,使得作物更加容易获得抗病、抗逆、高产等优良特性。然而,在转基因和基因编辑生物育种产品研发和产业化过程中,清晰的分子特征识别是其通过安全评价和商业化应用的前提。转基因生物的分子特征识别一般包括插入位点、旁侧序列、拷贝数等。特别是外源基因整合拷贝数的准确分析有助于确认转基因插入的稳定性、预期功能的正常表达,以及规避因意外插入导致的潜在风险等。同时,基因组上特定基因的拷贝数变异(CNVs)通常会影响作物的产量抗逆性和代谢能力等关键性状。因此,准确评估目的基因的拷贝数在转基因生物安全评价和作物育种中都十分重要。
目前,研究人员已经开发了多种技术方法来定量分析目的基因的拷贝数,包括Southern杂交(SB)、实时定量PCR (qPCR)、数字PCR (dPCR)、以及全基因组测序(WGS)。然而,这些技术方法在实际转基因作物的拷贝数测定时的准确性和适用性等性能如何尚不清晰?研究团队选取了3种转基因作物的4个转基因事件(FG72大豆、12-5玉米、G6H1水稻和G281水稻)作为案例,从准确性、实验操作、时间、成本、效率等多个方面系统评估和比较了每种技术方法的优缺点(图 1)。
分析结果表明,Southern杂交(SB):该方法耗费时间也消耗大量DNA, 在检测多拷贝基因时,容易受到复杂排列时不完全消化和交叉杂交的影响,导致结果偏高。实时定量PCR(qPCR)在单拷贝基因检测中表现更为准确,操作简便,但在多拷贝数基因检测时存在一定的定量偏差。虽然,后期实验可以通过优化引物设计和反应条件,提高其检测精度。数字PCR(dPCR),凭借其多分区独立计数的优势,能够实现基因拷贝数的精确测定,特别是对于多拷贝数的基因的测定具有非常高的分辨率和精确性。双端全基因组重测序(PE-WGS),通过对全基因组的高覆盖率测序和数据统计分析,同样能够精确定量目的基因的拷贝数。特别是,PE-WGS可以同时测定多个目的基因的拷贝数,还可以提供目的基因插入位点、侧翼序列等信息,是转基因作物分子特征识别的最有效技术手段之一。
在目的基因拷贝数测定的实际应用中,目前四种方法都能够准确检测出单拷贝样本;dPCR和PE-WGS综合性能更加优越,被推荐首选方法。其中dPCR适用于小样本量、特定基因的高精度分析,而PE-WGS则更加适合多个基因拷贝数的同时测定。特别是针对具有商业化应用前景的转基因作物的目的基因的拷贝数测定时,建议使用PE-WGS方法,获得包括拷贝数在内的比较全面的分子特征数据,为后期安全评价提供可靠的实验数据。
参考文献
Xu, W., Liang, J., Wang, F., & Yang, L. (2024). Comparative evaluation of gene copy number estimation techniques in genetically modified crops: insights from Southern blotting, qPCR, dPCR and NGS. Plant Biotechnology Journal. DOI:10.1111/pbi.14466.
文章来源:植物生物技术Pbj