本文内容速览:
蛋白质是执行生命体功能的基本单元,往往以各种复合物的形式存在,构成生物体内错综复杂的互作网络,从而行使生物学功能。因此,许多生物学问题需要在蛋白质层面上进行研究。蛋白质组学(Proteomics)是研究一种细胞或者一种生物体表达的全部蛋白质的学科,已广泛应用于植物生长发育、逆境胁迫等领域,是后基因组时代的研究热点之一。
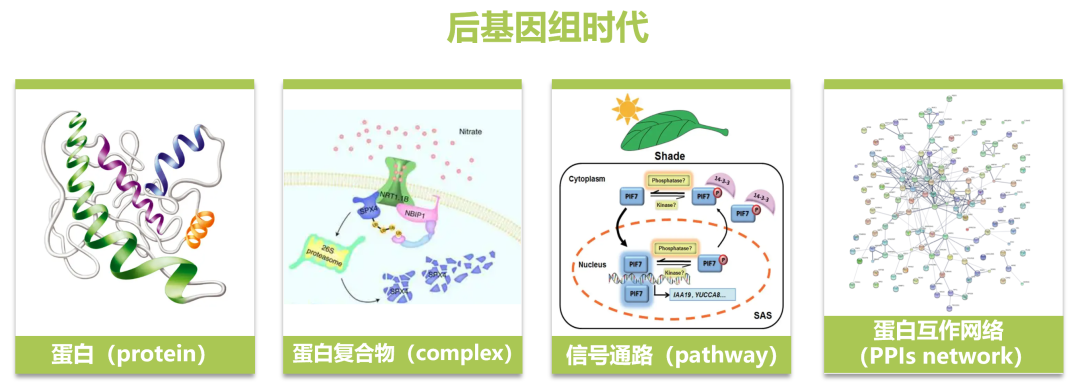
图1 蛋白质组学是后基因组时代的研究热点之一。图片来源:伯远生物。
植物蛋白质组具有功能的多样性和时空的特异性,也受到多种因素的影响,这些因素不断地改变蛋白质的丰度、修饰、相互作用、定位和活性,以满足植物的动态需求。分析植物蛋白质组及其潜在的遗传变异引起了越来越多研究者的关注。在这篇文章中,伯小远将给大家介绍蛋白质组学的相关内容。
最早应用于蛋白质组学的技术是双向凝胶电泳(2-DE),它基于蛋白质等电点和分子量的不同将混合物中的蛋白质分离。但这项技术独立使用时存在通量低、重叠率高等问题,因此通常被用来在质谱检测前分离蛋白质。随着质谱技术的发展,研究人员利用该技术能高效鉴定和定量生物样本中的蛋白质,揭示其在生物体内的表达水平、修饰状态和相互作用关系。接下来,伯小远主要为大家讲讲基于质谱的蛋白质组学分析策略。
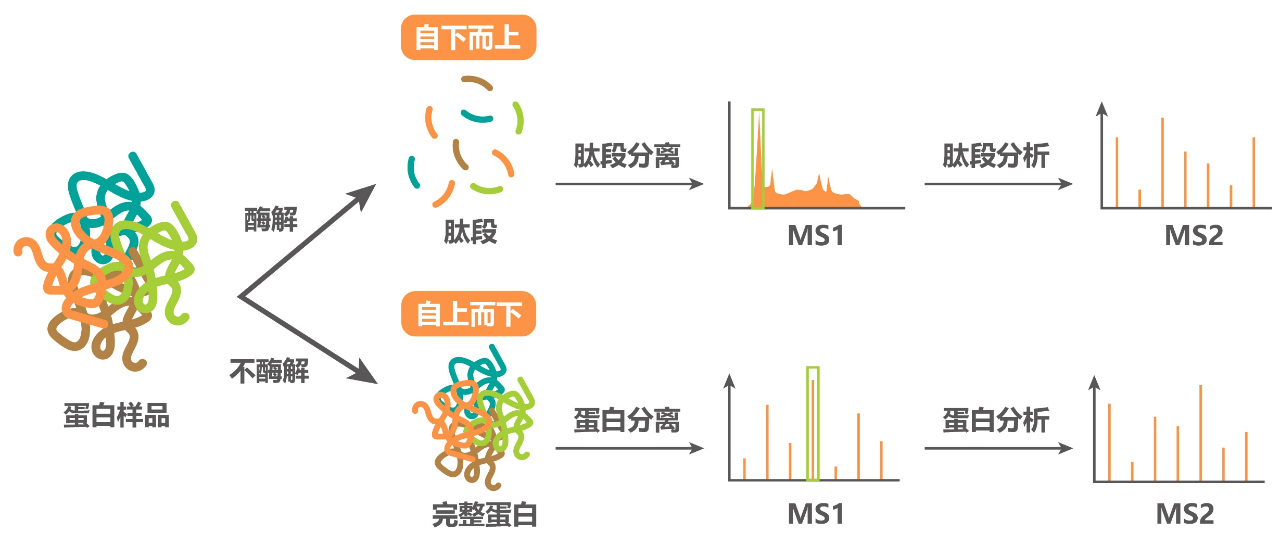
蛋白质组学的分析策略包括自上而下(Top-down)和自下而上(Bottom-up)。自上而下(Top-down)指的是针对特定的蛋白质,通过质谱碎片处理后检测碎片离子(也称子离子),从而推导出蛋白的序列;而自下而上(Bottom-up)是目前研究蛋白质组学的主流思路,也常被称为鸟枪法(Shotgun),该方法充分利用了蛋白质在特定的位点能够被酶解的特性,用蛋白酶对样本中全部蛋白质进行酶解,质谱鉴定酶解后的肽段,最后根据这些肽段的序列再推导出蛋白的序列。
蛋白质组学研究的基本技术流程包括样本准备、蛋白提取、蛋白酶解、肽段色谱分级、质谱检测和数据分析。
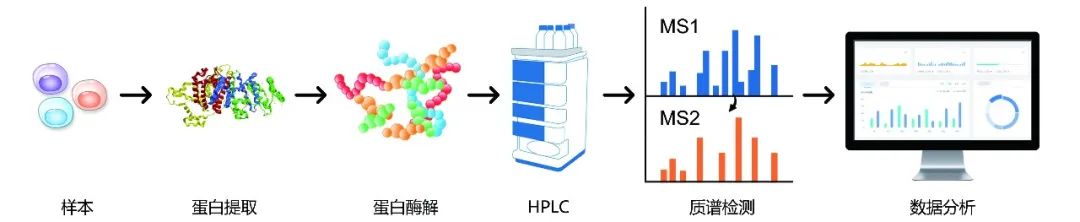 图3 蛋白质组学研究的基本技术流程。图片来源:伯远生物。
样本准备:根据具体实验需求,获取相应的样本材料;
图3 蛋白质组学研究的基本技术流程。图片来源:伯远生物。
样本准备:根据具体实验需求,获取相应的样本材料;
蛋白提取:根据样本类型,选择合适的方法来提取蛋白质;
蛋白酶解:利用序列特异性的酶对蛋白进行酶解;
肽段色谱分级:酶解后的肽段进入高效液相色谱仪(HPLC),肽段会因为在色谱柱填料上的保留时间不同而得到预分离;
质谱检测:肽段经分离后,在质谱仪上进行检测,得到一级谱图和二级谱图的数据信号;
数据分析:使用搜库工具对质谱图进行自动化的分析,得到肽段及蛋白序列信息。
03 质谱仪
讲完蛋白质组学研究的基本技术流程,大家可能对质谱仪还不太了解。质谱仪的应用非常广泛,不仅可以用于蛋白质组学研究,还广泛用于化学物质、代谢物等的检测。虽然不同的应用领域使用不同类型的质谱仪,但是其工作原理基本相同。质谱仪一般由五部分构成,包括进样系统、离子源、质量分析器、检测器、数据采集处理系统。
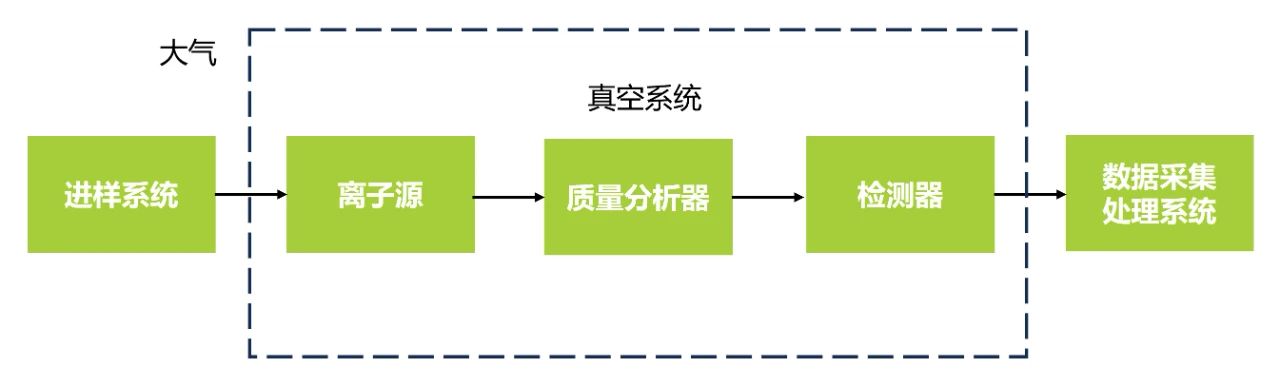
以检测蛋白质为例,质谱仪的工作原理是肽段在离子源中发生电离,产生带上电荷的肽段(也称母离子),经加速电场作用,形成离子束,进入质量分析器中,质量分析器根据质荷比(m/z)对母离子进行分离,记录各种母离子的质量数和丰度,即一级质谱(MS1或MS);再将部分或全部母离子进行二级碎裂,得到子离子,进入质量分析器根据m/z对子离子进行分离,记录各种离子的质量数和丰度,即二级质谱(MS2或MS/MS);由检测器负责进行数据信号转换和放大;最终由数据采集处理系统将数据信号处理成一级谱图和二级谱图,通过谱图来推测肽段的序列,再反推出蛋白质的序列,为我们提供所需要的定性及定量结果。
质量分析器是质谱仪中至关重要的组成部分,所以在这里拿出来单独说一说。各种类型质谱仪通常是根据质量分析器的类型来命名的,比如四级杆质谱、飞行时间质谱、四级杆离子阱质谱、轨道阱质谱等。质量分析器的性能主要由质量范围和分辨率来衡量。质量范围是指所能测定的质荷比的范围,决定了能检测到的离子种类和质量范围。分辨率则是更为重要的指标,可以表征两个相邻的谱峰在质谱中被区分开的能力,质谱分辨率越高,质谱图中的谱峰就会越尖越细。不同的质量分析器有不同的分辨率,但随着分辨率的提高,质谱仪的造价也会相应增加。因此,在选择质谱仪时,需要根据实验需求和预算综合考虑。
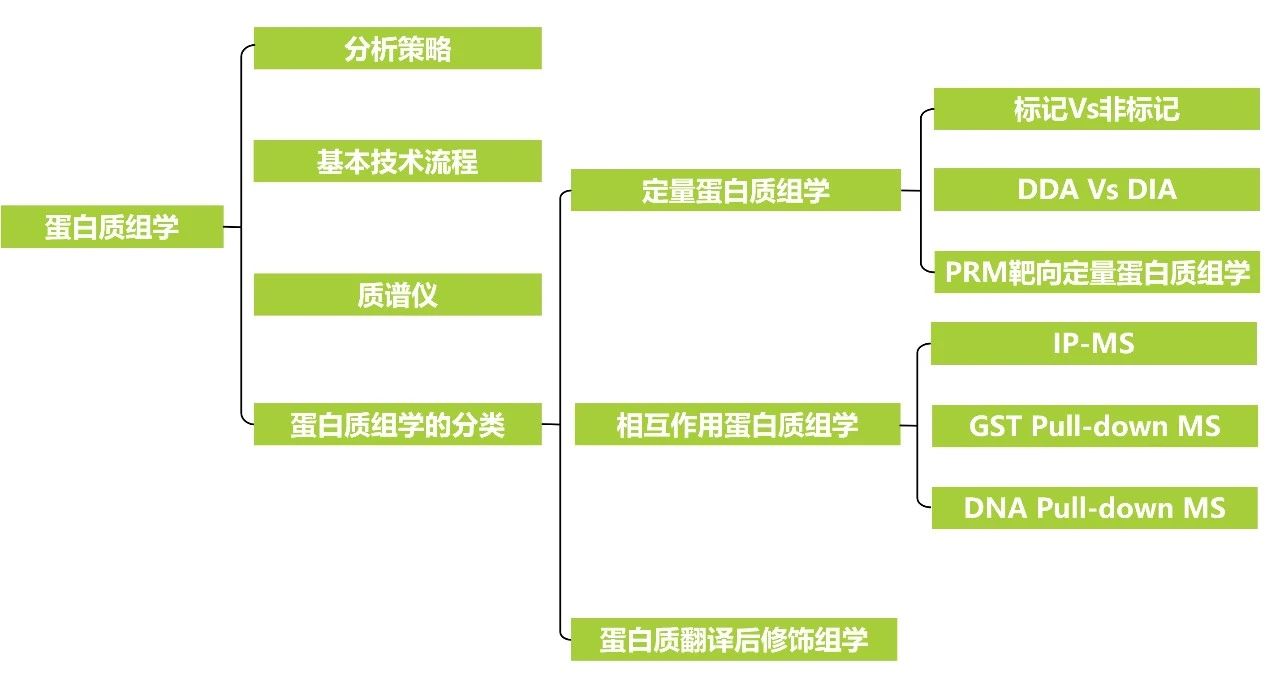
根据蛋白质组学能够揭示蛋白质的表达水平、修饰状态和相互作用关系等功能,可将蛋白质组学主要分为定量蛋白质组学、相互作用蛋白质组学、蛋白质翻译后修饰组学三个部分。
4.1 定量蛋白质组学
定量蛋白质组学可根据样本制备是否加入标记以及数据采集模式,进一步细分为Label Free非标记定量蛋白质组学、DIA定量蛋白质组学、TMT/iTRAQ标记定量蛋白质组学、PRM靶向蛋白质组学。
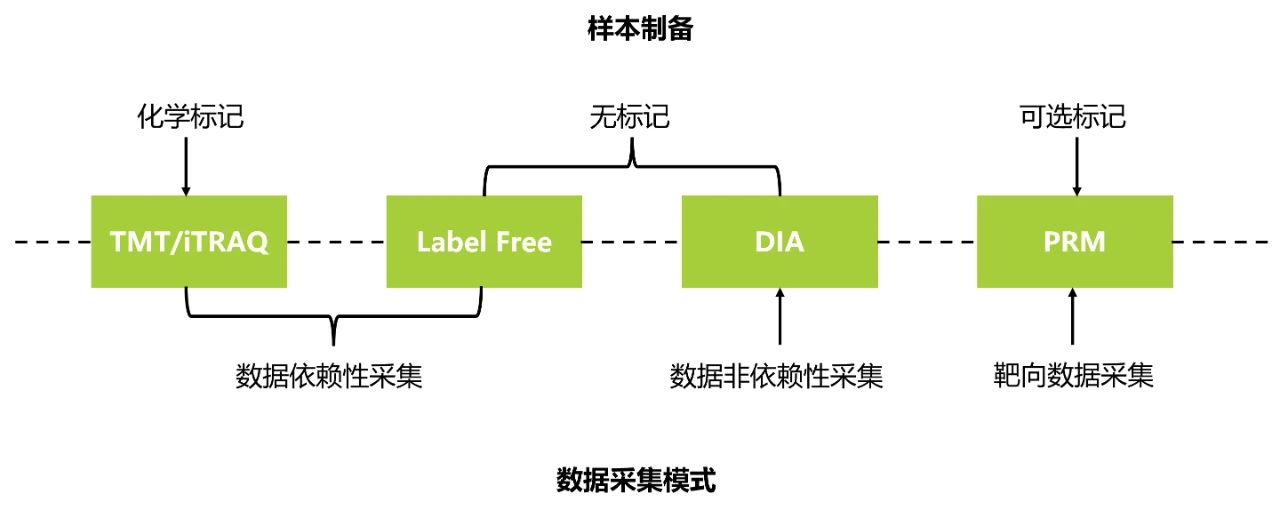
定量蛋白质组学可分为标记定量和非标记定量两大类。标记定量蛋白质组学可以同时检测多个样本,而非标记定量蛋白质组学一次只能检测一个样本,但标记定量蛋白质组学需要使用标记试剂,价格较为昂贵。目前基于标记的定量蛋白质组学方法可分为代谢标记和化学标记。代谢标记通常指SILAC,通过在培养基中添加稳定同位素标记的氨基酸,细胞在生长代谢过程中蛋白质原有的氨基酸会被同位素标记的氨基酸替换,不同同位素标记的同一种氨基酸存在质量差,因此能够被质谱仪检测区分出来。
在这里主要介绍TMT和iTRAQ两种化学标记方法。TMT和iTRAQ为两种不同的化学标签,其标签均由报告基团、平衡基团和肽反应基团三部分构成。报告基团用来指示蛋白质的丰度,平衡基团保证标记的不同样本的同一肽段的m/z相同,而肽反应基团则是与肽段的N-term或末端赖氨酸的侧链基团结合达到标记肽段的作用。
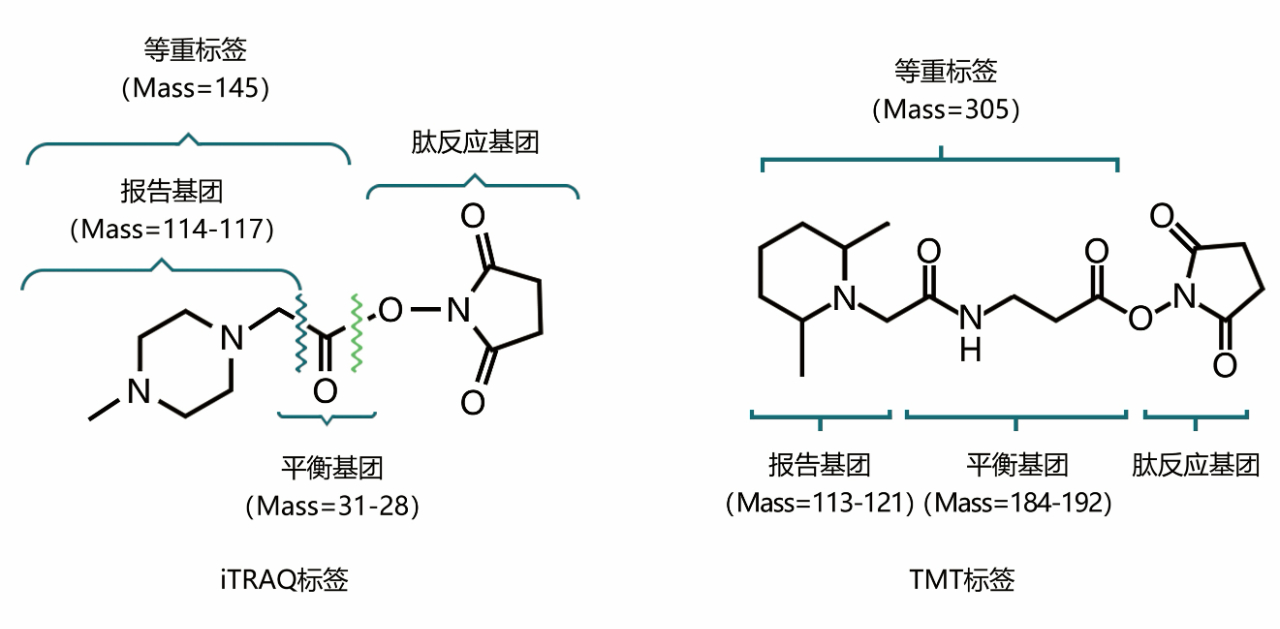
图6 iTRAQ和TMT化学标签结构示意图。图片来源:伯远生物。
基于不同样本中同一肽段经TMT/iTRAQ试剂标记后具有相同的质量数,并在MS1中表现为同一个质谱峰。当此质谱峰被选定进行碎裂后,在MS2中,不同的报告基团被释放,它们各自的质谱峰的信号强弱代表着来源于不同样本的该肽段及其所对应的蛋白的表达量的高低。同时,根据肽段的谱图结果可以鉴定出相应的蛋白种类。目前,iTRAQ标记蛋白质组学最多可同时比较8个样本,TMT标记蛋白质组学最多可同时比较18个样本,一旦超过最大样本个数,需要加入内参标签二次上机检测。由于TMT和iTRAQ标记定量蛋白质组学两者的原理相似,也可统称为TMT/iTRAQ标记定量蛋白质组学。
前面在提到质谱仪的工作原理时,讲到MS1后还需要对部分或全部母离子进行MS2,选择部分母离子还是选择全部母离子,这是质谱仪的两种数据采集模式,即数据依赖性采集(DDA)和数据非依赖性采集(DIA)。DDA是根据MS1中母离子的富集程度选择部分母离子,对选中的母离子进行碎裂得到的二级谱图进行定性和定量分析;DIA是按照m/z的不同将MS1的扫描范围分成多个窗口,每个窗口中包含多种母离子,对窗口中全部的母离子进行碎裂,采集所有母离子的子离子信息,生成二级谱图进行定性和定量分析。
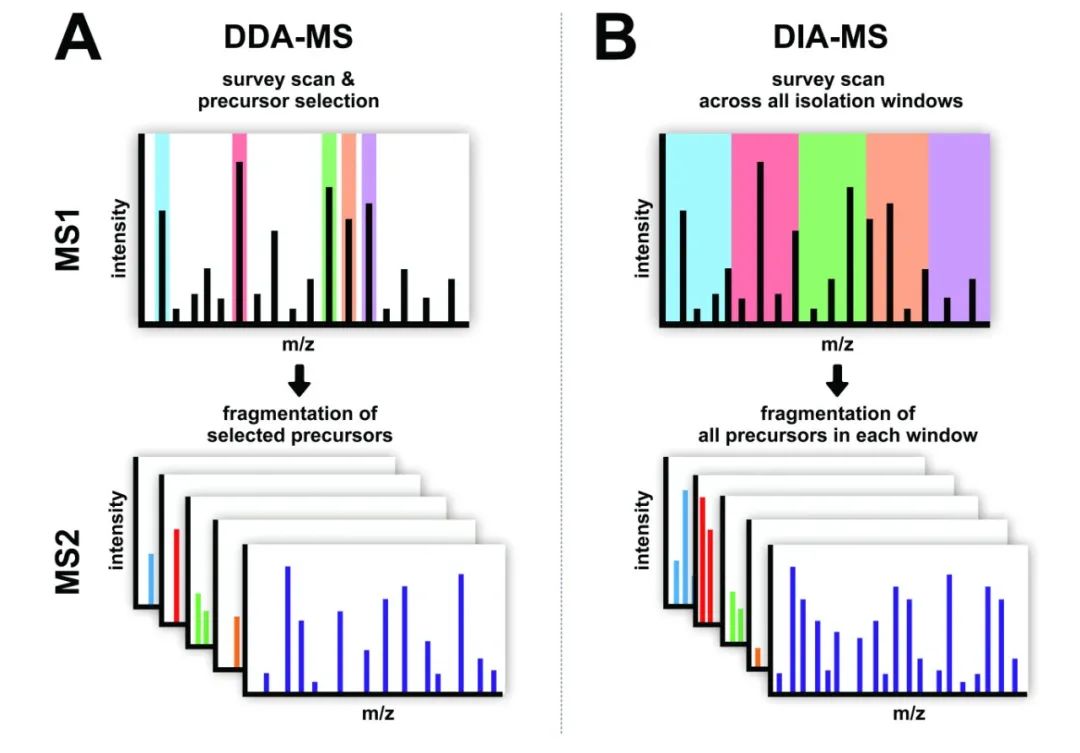
图7 蛋白质组学数据采集模式(改编自Krasny et al., 2021)。
Label Free非标记定量蛋白质组学采用DDA模式,易丢失低丰度蛋白的信息,数据重现性相对差,适合较多样本的初步检测。DIA定量蛋白质组学采用DIA模式,检测通量高、灵敏度高,可鉴定到低丰度蛋白,更适用于检测血浆、血清、尿液等样本中的低丰度蛋白。
靶向蛋白质组学技术是针对选中的目标蛋白或肽段进行监测和定量,被认为是“质谱领域的WB”。SRM/MRM靶向蛋白质组学只选择一个母离子,碰撞后,从形成的子离子中也只选一个离子进行检测。因为两步都只选单离子,针对性很强,可以排除噪音和干扰的影响。随着高分辨质谱在蛋白质组学领域普及,基于高分辨Orbitrap质谱仪的PRM靶向蛋白质组学被开发出来,PRM靶向蛋白质组学只需要选定母离子,而不需要选定子离子,比SRM/MRM靶向蛋白质组学简便很多。
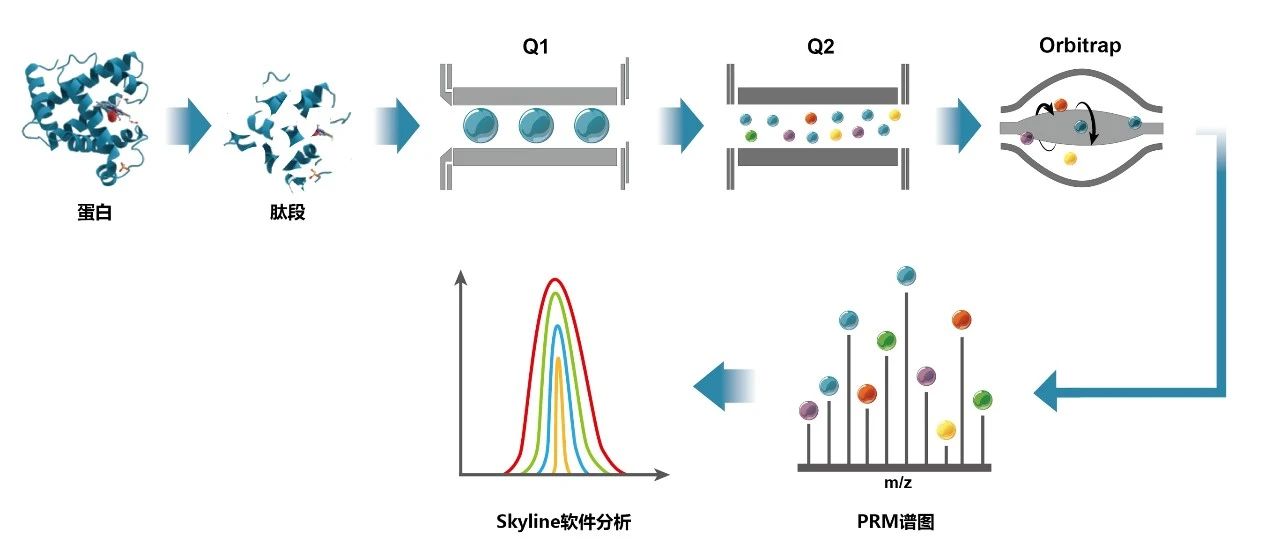
图8 PRM靶向蛋白质组学的实验流程图。图片来源:伯远生物。
定量蛋白质组学可以帮助大家寻找与特定研究领域密切相关的目的基因。通过比较不同样本间的定量蛋白质组学数据,大家能够得到差异表达蛋白,接着利用GO、KEGG、COG富集分析,获得差异表达蛋白的功能注释信息,从中选择与研究领域密切相关的某个差异表达蛋白,编码这个差异表达蛋白的基因即是目的基因,大家可以进行后续的基因功能研究。同时,比较转基因和野生型材料的定量蛋白质组学数据,可以从蛋白层面来解析基因功能。下面一起来看具体的案例吧!
2023年1月,四川农业大学王文明课题组在Nature Plants杂志上在线发表了一篇题为“A natural allele of proteasome maturation factor improves rice resistance to multiple pathogens”的研究论文,该文章报道了一个编码蛋白酶体成熟因子的天然等位基因,该基因能够赋予水稻对稻瘟病、稻曲病、纹枯病、白叶枯病等多种病害的抗性,同时不影响水稻产量。
作者首先通过转录组学筛选到了具有广谱抗性的蛋白酶体成熟因子OsUMP1,OsUMP1在R2115中高表达。为了验证R2115水稻中OsUMP1基因的广谱抗性,作者将R2115的OsUMP1等位基因转入至TP309水稻中,得到UMP1R2115-1和UMP1R2115-13两个转基因株系,病变区域和稻瘟菌生长量均减少。作者研究发现水稻中的UMP1也发挥了蛋白酶体形成的功能,实验分析中UMP1R2115水稻与TP309相比,表现出更高水平的蛋白酶体丰度和活性(图9a-e)。作者采用iTRAQ标记定量蛋白质组学的方式检测UMP1R2115-1水稻和野生型TP309水稻之间蛋白质水平的变化(图9f),研究发现在UMP1R2115-1中三种抗血酸过氧化物酶B8AU10(POD)、A3A7Y3(OsAPX8)、B9FV80(POD)和CAT A0A0R7FMX6(OsCatB)蛋白水平均有所降低(图9g),ELISA实验进一步证实了UMP1R2115-1水稻中的参与抑制植物免疫和负调控水稻抗病性的POD和CAT蛋白表达水平的降低,活性也降低(图9h-k),而H2O2水平增加(图9l)。这表明UMP1R2115水稻中可能存在蛋白酶体-POD/CAT-H2O2的防御途径。

图9 OsUMP1调节蛋白酶体和ROS清除酶的丰度以实现H2O2稳态(Hu et al., 2023)。(a-e)实验分析中UMP1R2115水稻与TP309相比,表现出更高水平的蛋白酶体丰度和活性,而敲除OsUMP1基因的水稻(ko-11)与TP309相比,蛋白酶体丰度和活性下降;(f)TP309和UMP1R2115-1两个样本之间的差异表达蛋白(DAP)的数量;(g)UMP1R2115-1与TP309相比,下调DAP的热图。色标表示蛋白质的丰度;(h-i)ELISA测定接种稻瘟菌GZ8后TP309、ko-11和UMP1R2115-1水稻叶片中POD(h)和CAT(i)蛋白的丰度;(j)接种GZ8后TP309、ko-11和UMP1R2115-1水稻叶片中POD(j)和CAT(k)的酶活性;(l)接种GZ8后TP309、ko-11和UMP1R2115-1水稻叶片中H2O2的定量。
免疫共沉淀(Co-IP)和GST拉下实验(GST Pull-down)是验证蛋白互作的经典方法,如果两个实验技术分别与质谱结合可以筛选出多个可能与目的蛋白相互作用的未知蛋白,分别被称为IP-MS和GST Pull-down MS,IP-MS是更接近植物生理状态的体内实验,而GST Pull-down MS是体外实验。DNA拉下实验(DNA Pull-down)是验证核酸和蛋白互作的方法,质谱与之联用,可以筛选出多个可能与目的基因相互作用的未知蛋白,被称为DNA Pull-down MS。
IP-MS的基本原理是基于特异性抗体与靶标蛋白的亲和作用,通过免疫共沉淀将与目的蛋白相互作用的未知蛋白从复杂的样本中获取出来,然后进行质谱检测以筛选出可能与目的蛋白相互作用的蛋白。理想的IP-MS实验是通过特异性抗体靶向目的蛋白以获得更接近生理状态下的相互作用蛋白,但这种检测准确性很大程度上会受到目的蛋白内源表达水平低或抗体效价低等问题的影响。因此,过表达目的蛋白并带有融合标签(例如Flag、GFP等)获得稳定转化材料是一种很好的选择。但是,对于一些没有稳定转化体系的物种,也可以通过瞬时转化来进行实验。
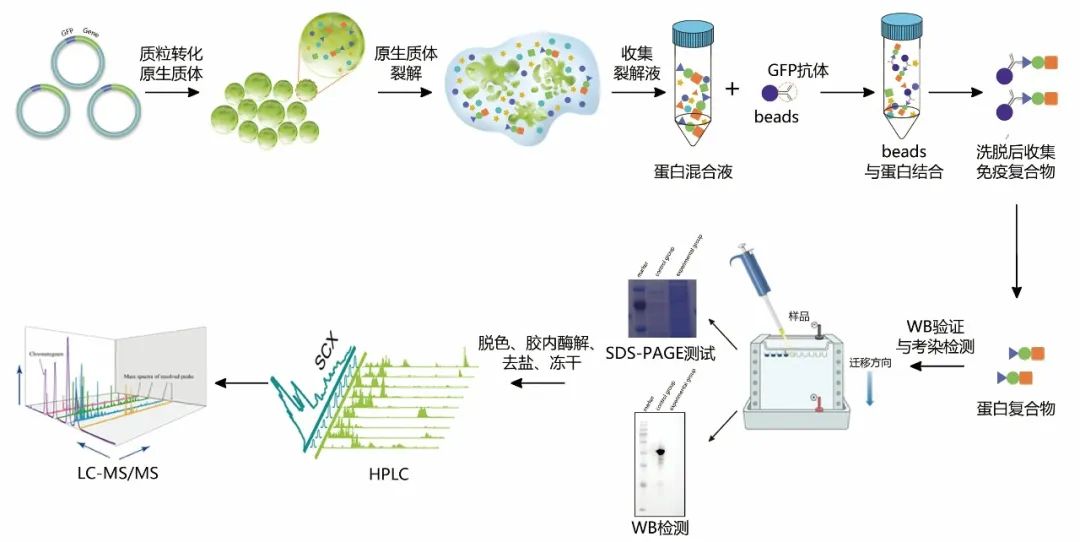
图10 基于原生质体瞬转体系的IP-MS实验流程图。图片来源:伯远生物。
GST Pull-down MS的基本原理是将目的蛋白与GST标签的融合蛋白亲和固化在谷胱甘肽亲和树脂上,充当“诱饵蛋白”,将提取的总蛋白与“诱饵蛋白”孵育,可捕获与“诱饵蛋白”相互作用的“猎物蛋白”,洗脱结合物后通过质谱分析,从而筛选出可能的互作蛋白。
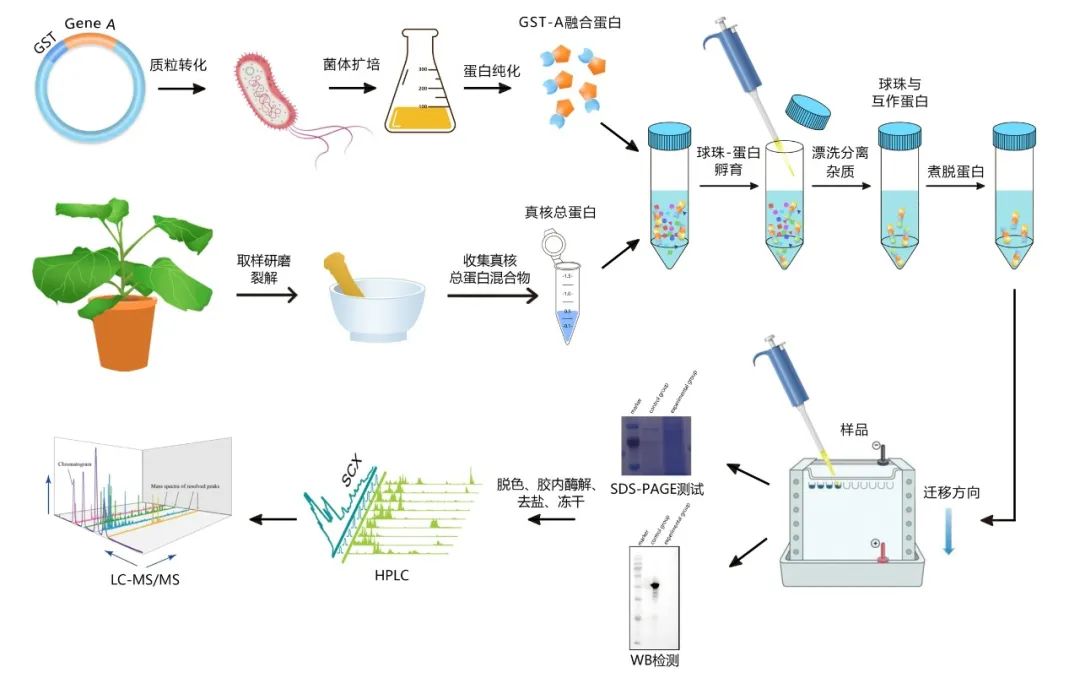
图11 GST Pull-down MS实验流程。图片来源:伯远生物。
DNA Pull-down MS的基本原理是将生物素标记的DNA片段结合在链霉亲和素磁珠上,形成磁珠-DNA探针复合体,再与植物总蛋白溶液孵育,这可以将与DNA结合的蛋白吸附在磁珠上。洗涤去除未结合及非特异性结合蛋白分子后,将与DNA结合的蛋白洗脱下来,通过MS鉴定与靶DNA片段结合的蛋白。
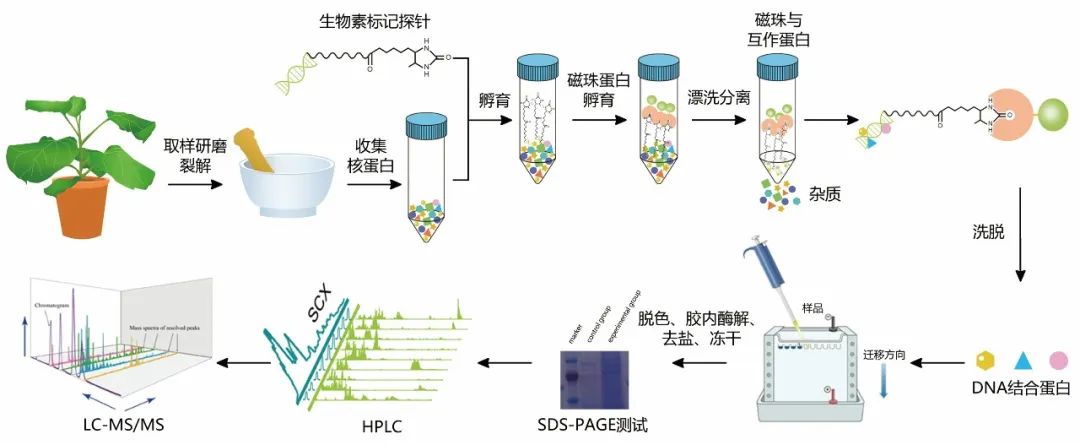
图12 DNA Pull-down MS流程图。图片来源:伯远生物。
通过相互作用蛋白质组学,大家能够寻找与目的蛋白或目的基因相互作用的蛋白,然后再利用蛋白-蛋白和蛋白-核酸互作实验进行点对点验证。
2024年1月,西北农林科技大学郭军课题组在Plant, Cell & Environment杂志上发表了一篇题为“Glycine-serine-rich effector PstGSRE4 in Puccinia striiformis f. sp. tritici targets and stabilizes TaGAPDH2 that promotes stripe rust disease”的研究论文。该文章报道了小麦条锈菌富含甘氨酸/丝氨酸效应子PstGSRE4劫持感病因子TaGAPDH2以调控植物免疫的新机制。作者通过IP-MS技术筛选出了PstGSRE4的互作蛋白甘油醛-3-磷酸脱氢酶TaGAPDH2,并通过酵母双杂实验(Y2H)、GST拉下实验(GST Pull-down)和免疫共沉淀(Co-IP)证实两者存在相互作用(图13)。沉默TaGAPDH2后,小麦对条锈菌的抗性增强,而过表达TaGAPDH2能够显著提高条锈菌致病力,表明TaGAPDH2为小麦感条锈病因子。进一步发现TaGAPDH2能够阻止活性氧(ROS)积累进而抑制细胞坏死,而效应子PstGSRE4能够增强TaGAPDH2的稳定性从而促进病原菌侵染。
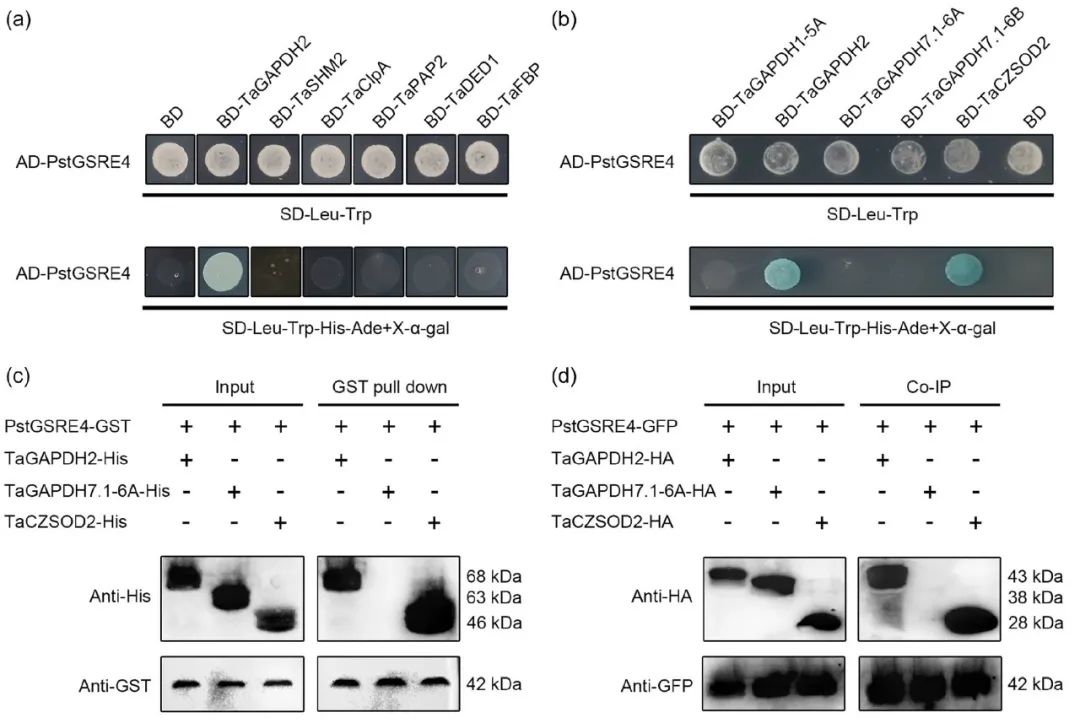
图13 PstGSRE4在体外和体内与小麦甘油醛-3-磷酸脱氢酶TaGAPDH2相互作用(Liu et al., 2024)。(a、b)Y2H实验验证TaGAPDH2与PstGSRE4相互作用;(c)GST Pull-down实验验证PstGSRE4与TaGAPDH2在体外相互作用;(d) Co-IP实验验证PstGSRE4与TaGAPDH2在体内相互作用。
4.3 蛋白质翻译后修饰组学
蛋白质翻译后修饰(Protein translational modifications, PTMs)是指在蛋白质氨基酸残基上添加或移除特定的基团。这种修饰可以改变蛋白质的结构、活性和稳定性,因此在细胞信号转导调控、蛋白质定位和细胞功能维持中发挥着重要作用。植物中常见的PTMs有磷酸化、糖基化、泛素化、乙酰化、S-亚硝基化和脂质化等。这些修饰的发生和去除通常由相应的修饰酶和去修饰酶所介导,修饰酶或去修饰酶需要与下游被修饰蛋白发生互作以实现修饰基团的添加或移除,研究这种互作关系可以解析PTMs对蛋白的调控,明确PTMs对生物体的意义,是蛋白研究的热点之一。蛋白质翻译后修饰组学可以鉴定PTMs,此外还可以准确定位修饰的位点。通过结合标记和非标记的定量方法,大家能够实现对修饰事件的定量分析,从而揭示修饰的丰度变化与生物过程的相关性。往期对蛋白质翻译后修饰已经进行了系统的介绍,这里就不再叙述了。蛋白质翻译后修饰组学的文献案例可以查看往期公众号文章“蛋白翻译后修饰研究套路”。
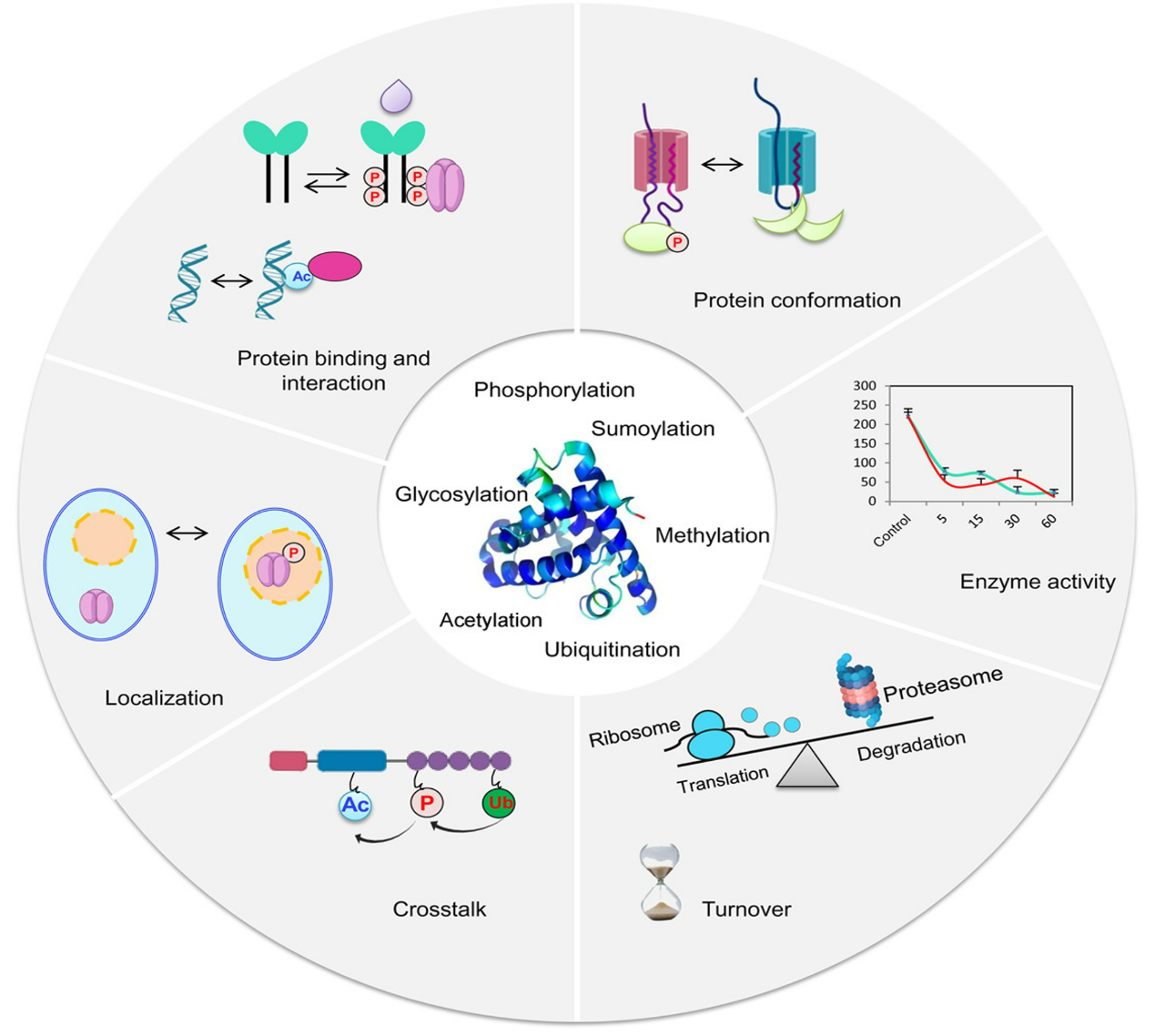
图14 PTMs功能的多样性和复杂性(Chen et al., 2021)。
最后来回顾一下今天所讲的内容,小远为大家介绍了蛋白质组学的分析策略、基本技术流程、质谱仪的结构以及工作原理,还系统地阐述定量蛋白质组学、相互作用蛋白质组学、蛋白质翻译后修饰组学的概念和特点。往期的公众号文章也有许多关于蛋白质组学的内容,对蛋白质组学感兴趣的小伙伴可以找来阅读,进行系统地学习。
我司从国外引进了Orbitrap Astral高分辨率质谱仪、Orbitrap Exploris 120质谱仪和TSQ Altis Plus三重四极杆质谱仪,构成了伯远生物“表观组-转录组-蛋白组-代谢组”四位一体的多组学平台,有需要蛋白质组学或多组学技术服务的小伙伴,欢迎致电详询哦!
Chen Y, Wang Y, Yang J, et al. Exploring the diversity of plant proteome[J]. Journal of Integrative Plant Biology, 2021, 63(7): 1197-1210.
Hu X H, Shen S, Wu J L, et al. A natural allele of proteasome maturation factor improves rice resistance to multiple pathogens[J]. Nature Plants, 2023, 9(2): 228-237.
Krasny L, Huang P H. Data-independent acquisition mass spectrometry (DIA-MS) for proteomic applications in oncology[J]. Molecular omics, 2021, 17(1): 29-42.
Liu C, Wang Y, Du Y, et al. Glycine‐serine‐rich effector PstGSRE4 in Puccinia striiformis f. sp. tritici targets and stabilizes TaGAPDH2 that promotes stripe rust disease[J]. Plant, Cell & Environment, 2024, 47(3): 947-960.
















