本文内容速览:
表观遗传修饰是通过化学修饰改变染色体上的DNA和蛋白质,这种修饰可以影响基因的转录、剪接、翻译、核小体组装和染色质结构等多个层面,从而影响植物的生长发育以及生物或非生物胁迫响应过程。表观遗传修饰主要包括DNA修饰、组蛋白修饰和染色质重塑等。伯小远在往期推文“生命科学研究热点——组蛋白修饰”、“蛋白翻译后修饰研究套路”中介绍了组蛋白修饰以及组蛋白修饰的研究方法。在这篇文章中,伯小远将给大家介绍DNA修饰的相关内容,DNA修饰主要是指DNA甲基化(DNA methylation),DNA甲基化与植物的基因表达、生长发育和抵御逆境胁迫等重要的生命过程有着密切的联系。研究DNA甲基化是当今表观遗传学的热点之一。全基因组重亚硫酸盐测序(Whole genome bisulfite sequencing, WGBS/BS-seq)是DNA甲基化研究的金标准。今天就跟着伯小远一起学习一下DNA甲基化以及BS-seq技术是如何运用到植物DNA甲基化研究的吧。
1 DNA甲基化的定义
DNA甲基化是指在DNA甲基转移酶的催化下,以S-腺苷甲硫氨酸(S-adenosyl methionine, SAM)为甲基供体,将甲基基团共价结合到DNA分子上的一种修饰形式(Lister et al., 2008)。DNA甲基化修饰可以发生在多个位点,包括胞嘧啶的C-5位、腺嘌呤的N-6位及鸟嘌呤的G-7位。一般我们所关注的DNA甲基化主要是指发生在CpG二核苷酸中胞嘧啶上第5位碳原子的甲基化,其产物称为5-甲基胞嘧啶(5-mC)。这是动植物等真核生物中DNA甲基化的主要形式。
2 植物DNA甲基化的模式
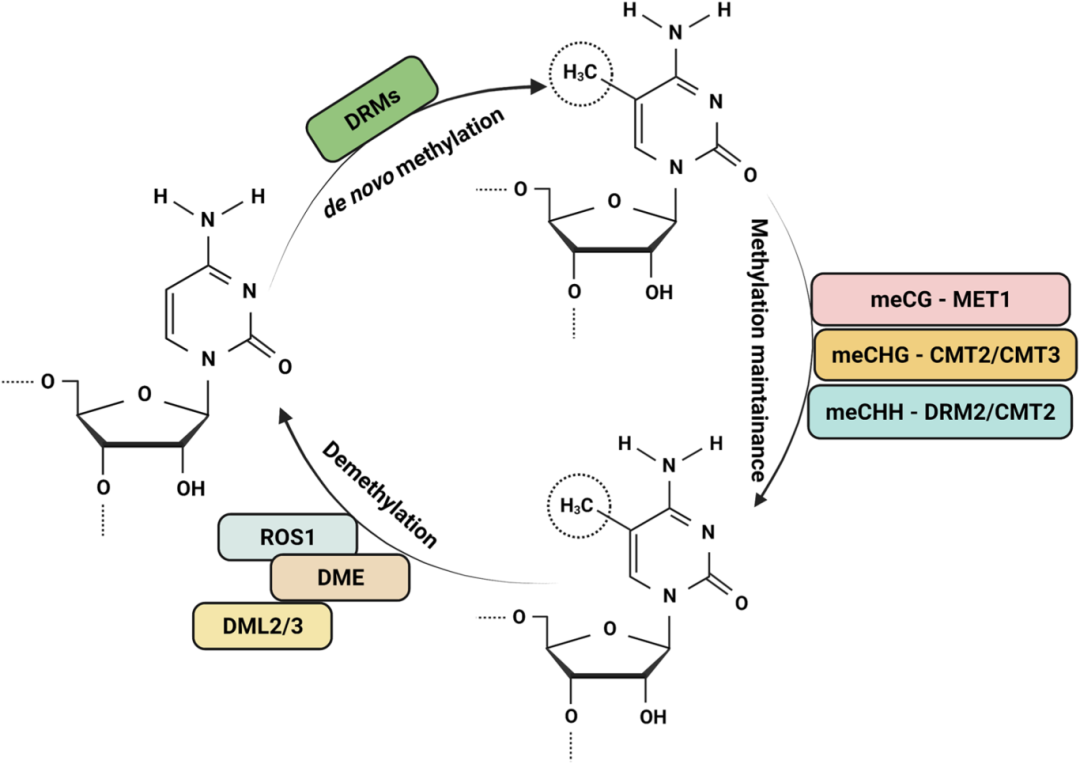
在植物中,DNA甲基化是从头甲基化(De novo methylation)、维持甲基化(Methylation maintainance)和去甲基化(Demethylation)三者动态调节的结果,并且这些调节途径受到不同酶的催化(图1)。
2.1从头甲基化
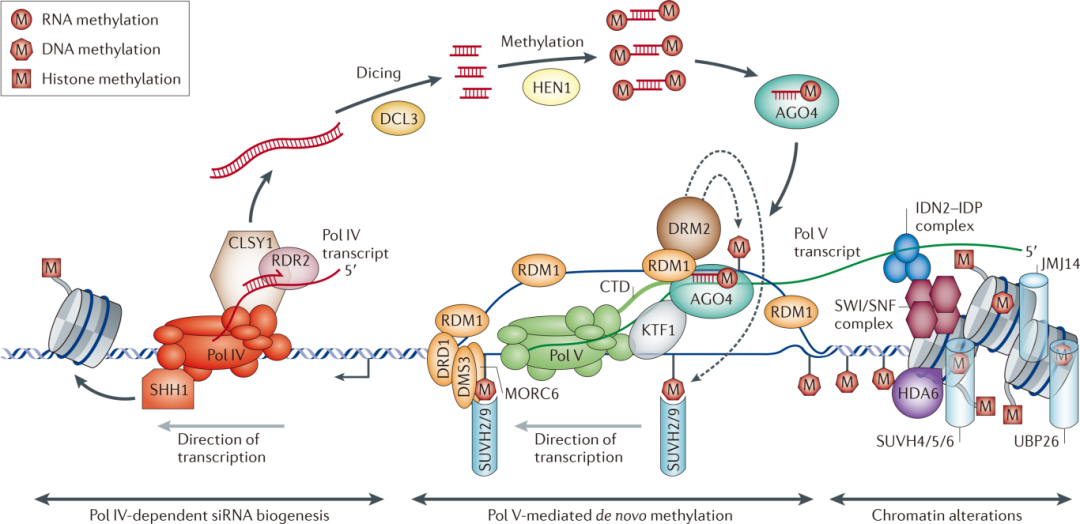
在植物中,从头甲基化是通过RNA介导的DNA甲基化(RNA-directed DNA methylation, RdDM)途径介导完成的甲基化修饰过程(Matzke et al., 2014)。RdDM途径的过程主要概括为以下3个步骤:
(1)依赖RNA聚合酶Pol IV的小干扰RNA(Small interfering RNAs, siRNAs)的产生;
(2)依赖RNA聚合酶Pol V的长非编码RNA(Long intergenic noncoding RNAs, lincRNAs)的产生;
(3)siRNAs和AGO蛋白结合并与lincRNAs互补配对,招募甲基转移酶2(Domain rearranged methyltransferase 2, DRM2)至靶标位点建立DNA甲基化。
图2 植物RdDM模型(Matzke et al., 2014)。在Pol IV的参与下,通过富集转座子和重复元件相关区域,转录产生单链RNA(ssRNA),之后由RNA依赖的RNA聚合酶2(RNA-dependent RNA polymerase 2, RDR2)以单链RNA为模板复制合成双链RNA(dsRNA),再通过Dicer-like 3(DCL3)蛋白将双链RNA切割成长度为24nt的siRNA。成熟的siRNA与AGO4蛋白结合形成复合体,并与Pol V转录的scaffold RNAs配对募集DRM2,最终完成从头甲基化修饰过程。
不同类型的甲基化形式由不同的甲基转移酶催化(图1)。CG甲基化由DNA甲基转移酶1(DNA methyltransferase 1, MET1)维持。MET1是哺乳动物DNA胞嘧啶5-甲基转移酶1(DNA cytosine-5-methyltransferase 1, DNMT1)的直系同源物,它通过识别DNA复制后半甲基化的CG二核苷酸,并将子链中未修饰的胞嘧啶甲基化。CHG甲基化由染色质甲基化酶2(Chromomethylase 2, CMT2)或者染色质甲基化酶3(Chromomethylase 3, CMT3)维持。CHH甲基化由结构域重排甲基转移酶2(Domains rearranged methyltransferase 2, DRM2)或CMT2维持。
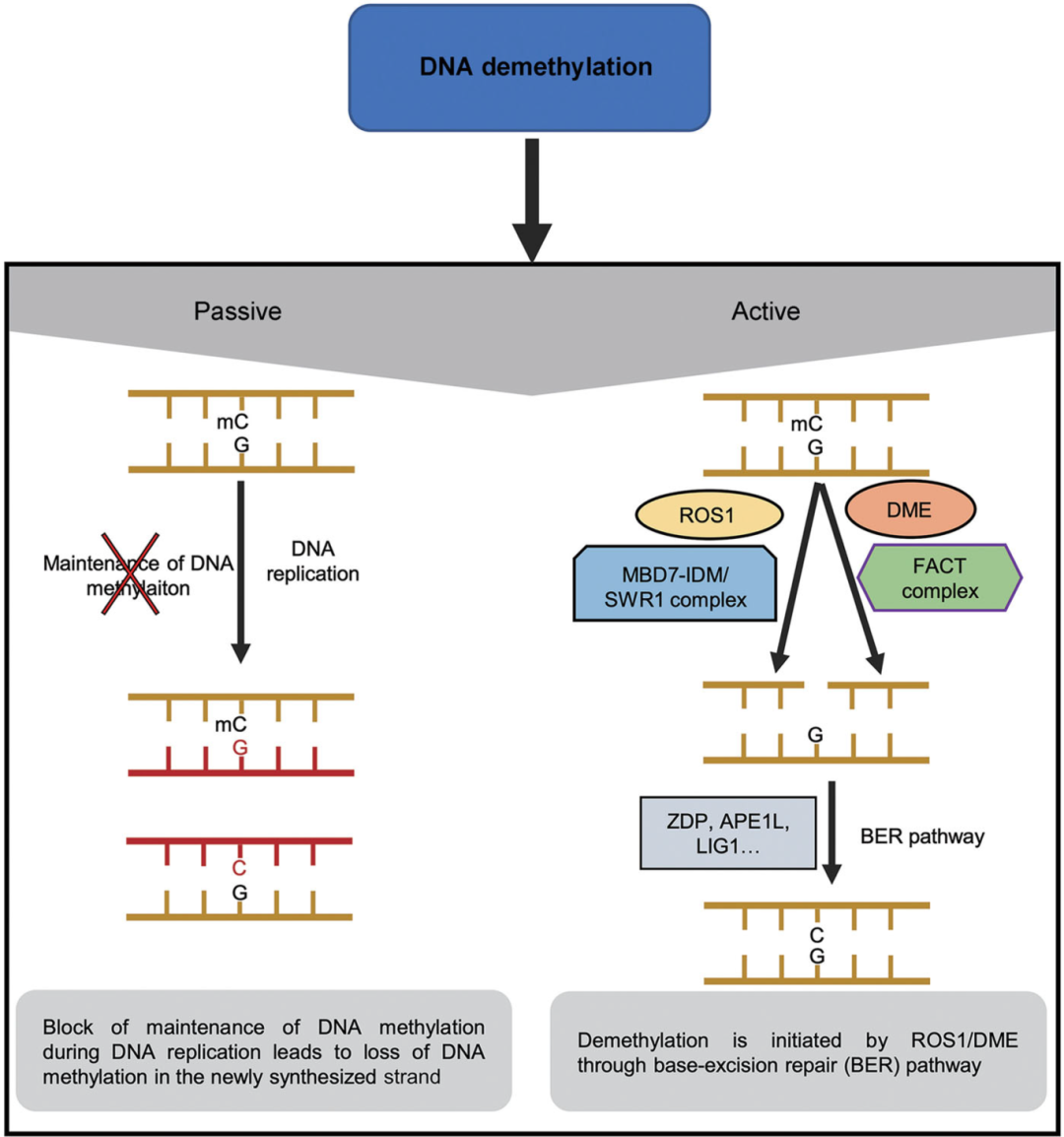
图3 DNA去甲基化机制(Liu et al., 2020)。被动DNA去甲基化可在DNA复制过程中发生,新合成DNA中的DNA甲基化水平会被降低。主动DNA去甲基化是由植物特有的ROS1/DME酶家族介导的,ROS1和DME分别通过MBD7-IDM或SWR1复合体以及促进染色质交易(FACT)复合体被招募到目标位点。甲基化的胞嘧啶经ROS1或DME识别后,从氮端的糖苷键处断裂形成了无碱基位点,然后被一个未甲基化的胞嘧啶修复,从而完成了这个位点的去甲基化修饰过程。
DNA甲基化参与调控植物花色。海棠花青素生物合成相关基因MhMYB10的启动子区域高度甲基化,使MhMYB10基因表达减少,最终导致花青素积累减少(Han et al., 2020)。牡丹PrANS和PrF3H启动子的DNA甲基化使其在花瓣非色斑区域的表达量降低,进而影响了牡丹花瓣颜色(Zhu et al., 2023)。
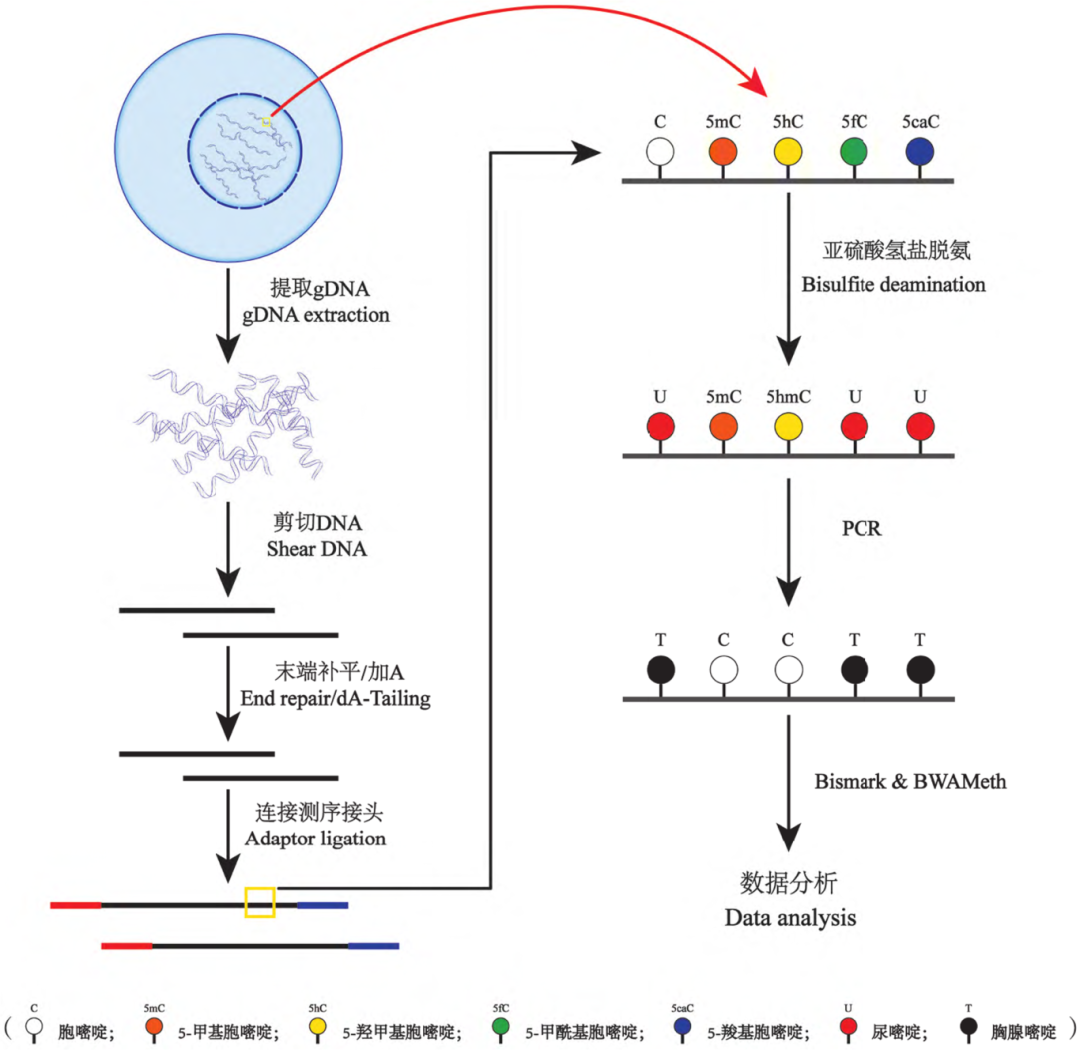
图4 BS-seq技术原理(袁明波等,2024)。
案例1——不同发育阶段的DNA甲基化
2024年2月,青岛农业大学张玉刚课题组在Horticulture Research杂志上发表了一篇题为“Hypermethylation in the promoter regions of flavonoid pathway genes is associated with skin color fading during ‘Daihong’ apple fruit development”的研究论文。作者以R1R6型红肉苹果品种“黛红”(DH)为材料,结合遗传背景分析、代谢组、转录组、甲基化组及分子生物学手段解析了DNA甲基化参与苹果果皮褪色的机理。
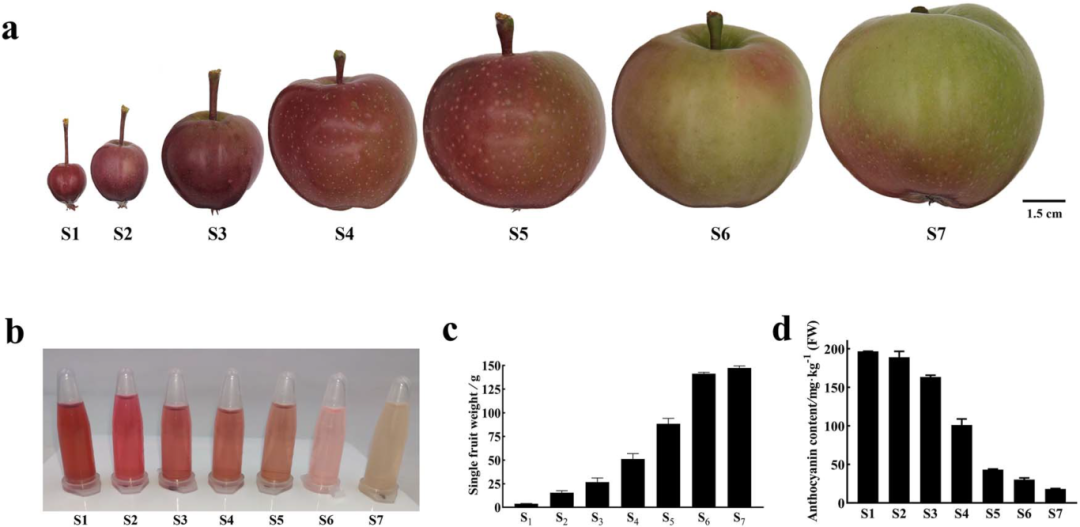
作者观察了七个发育阶段DH果实表型性状和果皮花青素含量,发现其幼果期果皮色泽为深红色,花青素含量丰富,随着果实发育果皮色泽变浅,成熟期的DH苹果果皮以绿色为主(图5)。
图5 七个发育阶段的DH果实表型性状和果皮花青素含量(Xu et al., 2024)。(a)S1-S7阶段的果皮颜色。S1-S7表示开花后30d、45d、55d、71d、90d、110d和131d的果实;(b)从DH果皮中提取的花青素;(c)单果重量;(d)通过pH示差法测定DH果皮花青素含量。
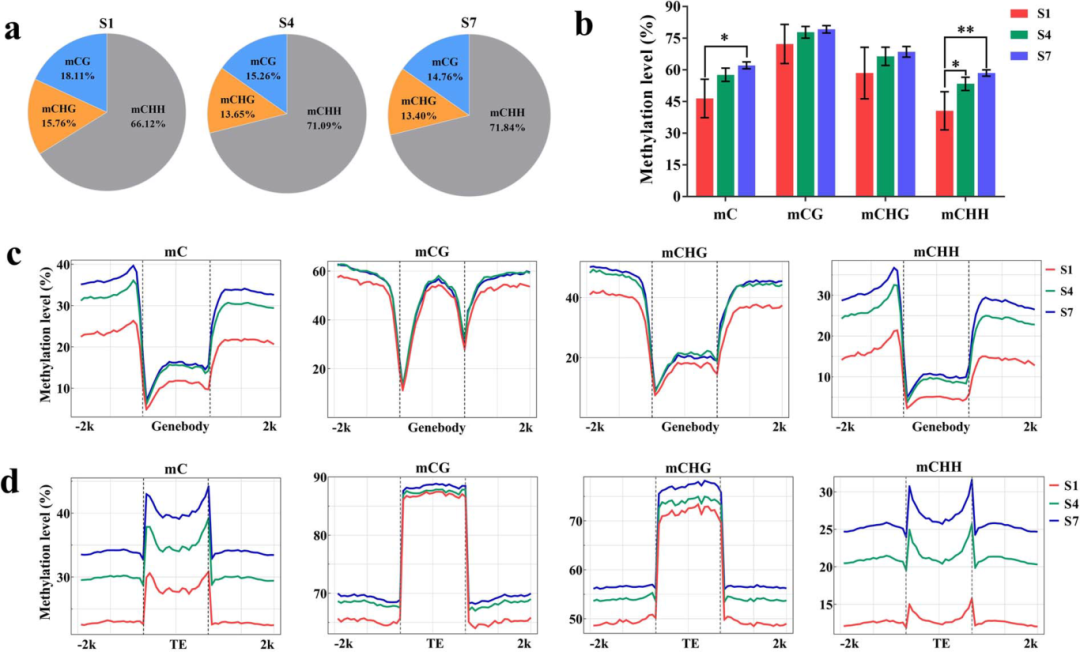
为了研究DH果实发育过程中的DNA甲基化图谱,作者对S1、S4和S7三个发育阶段的果实果皮进行了BS-seq分析,发现随着果实的发育,CG和CHG类型的相对甲基化水平下降,而CHH类型的相对甲基化水平上升(图6a)。总体而言,全基因组范围内甲基化水平随着果实发育而增加(图6b)。此外,在CG、CHG和CHH三种甲基化类型中,基因体和转座子元件(TE)上游或下游2k区域的DNA甲基化水平从S1到S7均逐渐增加(图6c、d)。
图6 DH果皮的DNA甲基化分析(Xu et al., 2024)。(a)mC、mCG、mCHG和mCHH在发育阶段S1、S4和S7的相对比例;(b)CG、CHG和CHH在S1、S4和S7处的DNA甲基化水平;(c)在S1、S4和S7区域,基因上游和下游2k区域的DNA甲基化水平;(d)在S1、S4和S7区域,TE上游和下游2k区域的DNA甲基化水平。
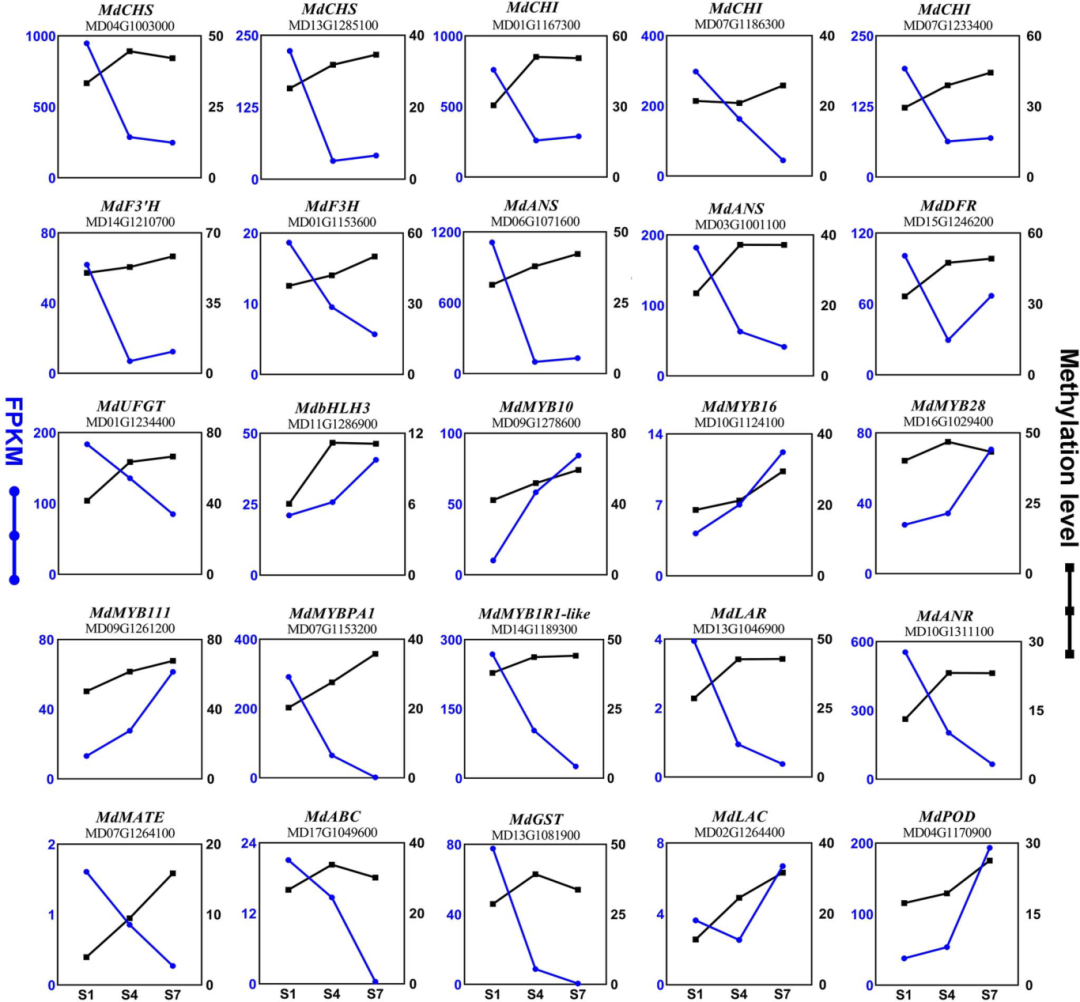
由于启动子区域的DNA甲基化会影响基因的表达,因此作者将花青素生物合成和降解过程中关键基因的甲基化水平和转录水平进行了关联分析,发现结构基因MdCHS、MdCHI、MdF3H、MdANS、MdUFGT、MdLAR和MdANR在其启动子区域内的DNA甲基化水平从S1到S7阶段呈上升趋势,而其转录水平呈下降趋势(图7)。调控基因MdbHLH3、MdMYB10、MdMYB16、MdMYB28和MdMYB111启动子区域内的DNA甲基化水平和转录水平均从S1到S7呈上升趋势(图7)。
案例2——胁迫处理下的DNA甲基化
2022年10月,云南大学乔琴课题组与云南中医药大学张体操课题组联合在BMC plant biology杂志上发表了一篇题为“Integrated transcriptome and methylome analyses reveal the molecular regulation of drought stress in wild strawberry (Fragaria nilgerrensis)”的研究论文,作者通过对干旱胁迫处理四个不同时间点的草莓的生理性状、基因表达谱、全基因组DNA甲基化图谱分析,综合解析了草莓的干旱响应调控网络。
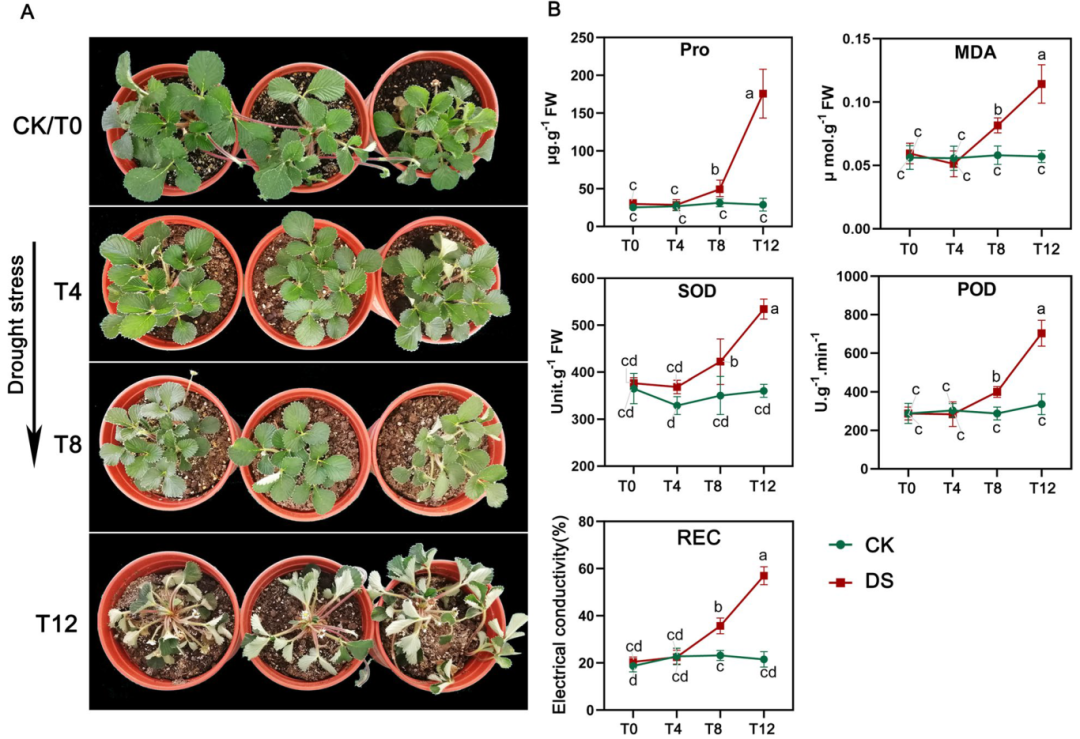
作者观察了干旱胁迫不同时间点的草莓形态,并测定了脯氨酸(Pro)、丙二醛(MDA)、超氧化物歧化酶(SOD)、过氧化物酶(POD)的含量和相对电导率(REC),发现在T8时间点草莓形态以及生理指标的变化较大(图8)。
图8 干旱胁迫下草莓的生理性状分析(Cao et al., 2022)。(A)草莓在干旱胁迫下的形态变化;(B)在干旱处理的四个时间点检测Pro、MDA、SOD、POD和REC。
为了研究干旱胁迫下DNA甲基化的调控机制,作者进行了BS-seq分析,发现CG、CHG和CHH三种类型的DNA甲基化水平分别为47.69%、30.87%和10.56%(图9A)。mCG、mCHG和mCHH在总DNA甲基化位点中的百分比在不同干旱胁迫时间点表现出动态变化,其中mCHH不仅占比最高,而且表现出最大变化(图9B)。在干旱胁迫期间,草莓基因启动子和重复序列的DNA甲基化水平显示出CHH甲基化的显著变化(图9C)。由于启动子和基因体的甲基化对基因表达有不同的影响,将差异甲基化基因(DMG)分为启动子DMG和基因体DMG,结果表明在干旱胁迫的所有时间点草莓启动子DMG均高于基因体DMG(图9D)。在启动子DMG中,所有时间点有317个低甲基化(hypo)共有基因和21个高甲基化(hyper)共有基因(图9E),表明这些基因的甲基化水平动态变化,可能直接或间接参与调控表达基因对干旱胁迫的调控。
图9 干旱胁迫下四个时间点DNA甲基化水平变化(Cao et al., 2022)。(A)T8时间点CG、CHG和CHH类型的DNA甲基化水平变化;(B)干旱胁迫不同时间点CG、CHG和CHH类型的DNA甲基化相对比例;(C)DNA甲基化的动态变化,包括基因组分和重复序列;(D)不同干旱胁迫时间点的DMR及DMG数量;(E)启动子DMG的Upset-Venn图。
案例3——突变体与野生型材料的DNA甲基化
2023年1月,华中农业大学康春颖课题组在Plant Physiology杂志上发表了一篇题为“Factor of DNA methylation 1 affects woodland strawberry plant stature and organ size via DNA methylation”的研究论文,作者通过BS-seq和转录组分析揭示了林地草莓(Fragaria vesca)DNA甲基化因子1(FDM1)通过调控DNA甲基化,从而影响林地草莓叶片、花朵和果实大小,表明了RdDM在草莓生长发育过程中的关键调控作用。
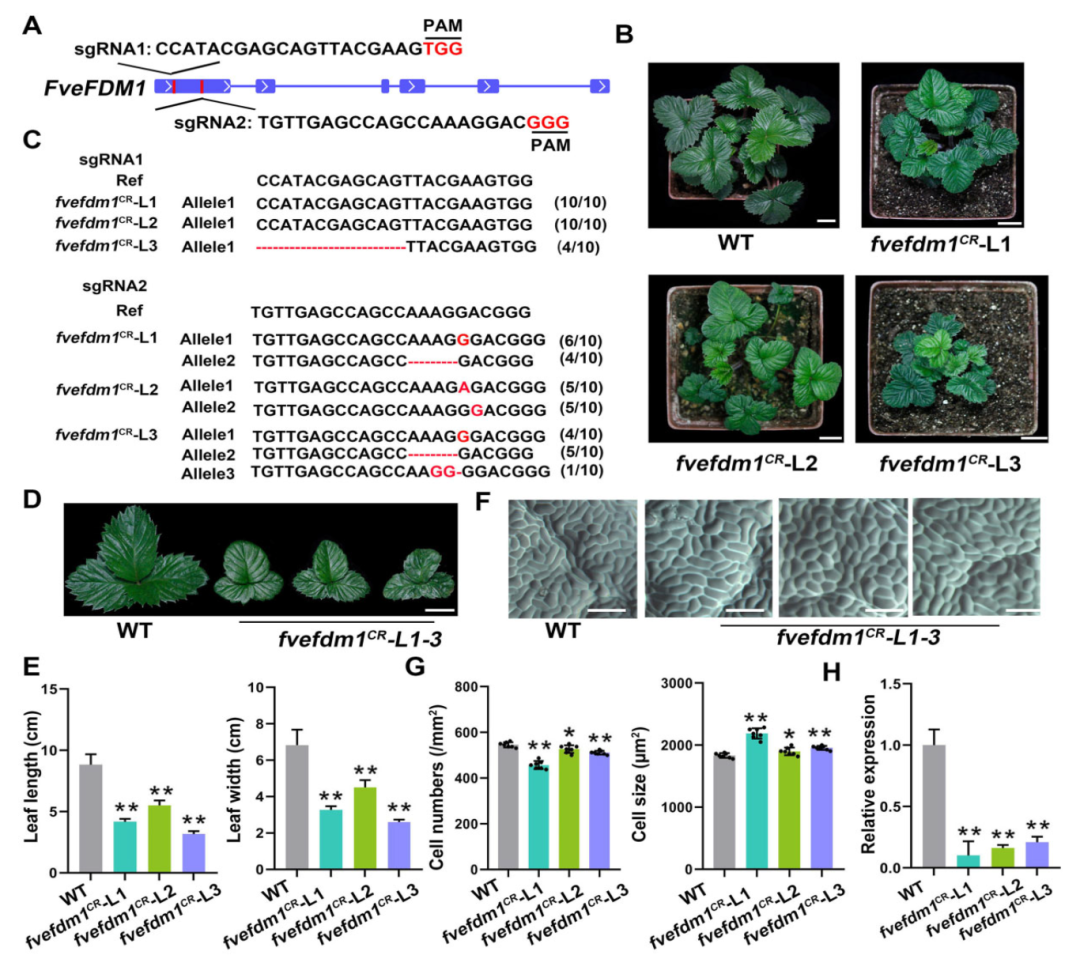
作者利用CRISPR/Cas9技术敲除FveFDM1获得三个敲除株系fvefdm1(L1-L3),发现与WT相比,fvefdm1发育出较小的叶片(图10A-E),fvefdm1的近轴叶表皮细胞数量显著减少,细胞大小增加(图10F、G),fvefdm1幼叶中FveFDM1的表达水平显著降低(图10H)。
图10 利用CRISPR/Cas9技术获得fvefdm1突变体及表型观察(Zheng et al., 2023)。(A)FveFDM1中sgRNA靶位点示意图;(B)T0代中的三个fvefdm1突变体(L1-L3)和WT的植株表型;(C)T0代中的三个fvefdm1突变体(L1-L3)的突变情况;(D)fvefdm1(L1-L3)和WT成熟叶片;(E)fvefdm1(L1-L3)和WT叶片大小;(F)fvefdm1(L1-L3)和WT叶片表皮近轴侧的细胞;(G)fvefdm1(L1-L3)和WT叶片表皮近轴侧的细胞数量和细胞大小;(H)FveFDM1在fvefdm1(L1-L3)和WT幼叶中的表达水平。
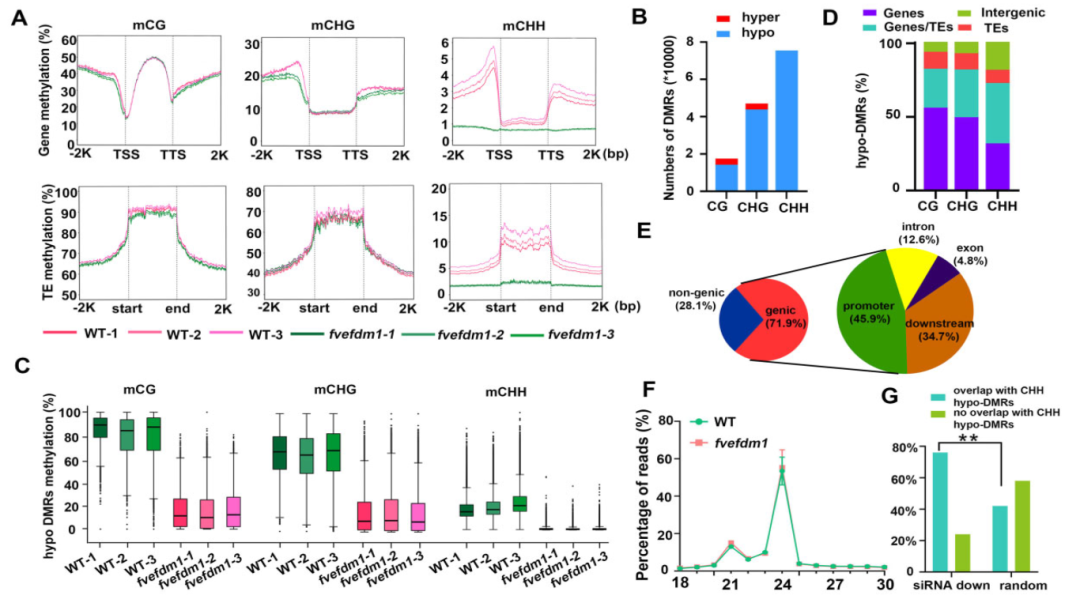
为了研究FveFDM1的分子功能,作者使用BS-seq检测了WT和fvefdm1中DNA甲基化水平,发现与WT相比,fvefdm1所有基因体和TE中CG和CHG类型的平均甲基化水平基本保持不变,CHH类型的平均甲基化水平显著减少(图11A)。接着作者在fvefdm1和WT之间鉴定了差异甲基化区域(DMRs),共识别出16831个CG DMR、46264个CHG DMR和74749个CHH DMR,其中大多数为hypo DMR(图11B),并且在fvefdm1中hypo DMR的DNA甲基化水平显著降低(图11C)。三种甲基化类型的hypo DMR主要富集于仅基因体区域或同时覆盖基因体和TE区域(图11D)。更具体地说,三种情况下的hypo DMR在基因的启动子中相对更丰富(图11E)。结果表明,FveFDM1在最有可能影响基因表达的位置调节DNA甲基化。
图11 fvefdm1中的DNA甲基化水平和siRNA丰度(Zheng et al., 2023)。(A)WT和fvefdm1中所有基因和TE中CG、CHG和CHH甲基化的平均水平,包括转录起始位点(TSS)上游2kb和转录终止位点(TTS)下游2kb;(B)fvefdm1中CG、CHG和CHH中相对于WT的hyper-DMR和hypo-DMR数量;(C)WT和fvefdm1中hypo-DMR中的CG、CHG和CHH甲基化水平方框图;(D)CG、CHG和CHH中hypo-DMR的基因组分布;(E)CHH hypo-DMR在不同基因组区域分布。Promoter表示TSS上游2kb,Downstream表示TTS的下游2kb;(F)WT和fvefdm1中与基因组比对的siRNA的大小分布;(G)下调(downregulated)(n=23395)或随机(random)(n=23395)siRNA簇与CHH-hypo-DMR重叠或不重叠的百分比。
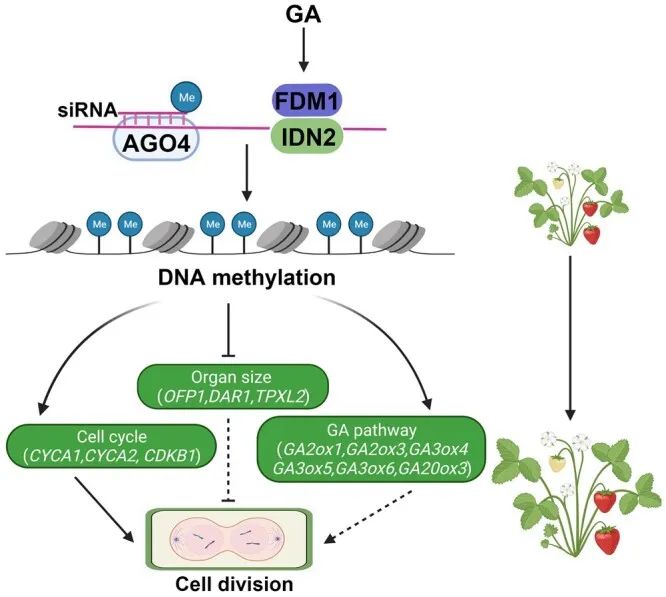
此外,作者还发现FveFDM1和FveIDN2蛋白的表达可由GA诱导,FveFDM2和FveIDN2的蛋白复合物直接结合AGO4 siRNA,以影响靶位点的DNA甲基化水平,下游调控基因受到DNA甲基化的促进或抑制,通过干扰细胞分裂最终导致器官大小改变。
References:
袁明波,叶国华,杨丹等. DNA甲基化测序技术研究进展[J/OL].生物技术通报:1-9.
Arora H, Singh R K, Sharma S, et al. DNA methylation dynamics in response to abiotic and pathogen stress in plants[J]. Plant Cell Reports, 2022, 41(10): 1931-1944.
Cao Q, Huang L, Li J, et al. Integrated transcriptome and methylome analyses reveal the molecular regulation of drought stress in wild strawberry (Fragaria nilgerrensis)[J]. BMC plant biology, 2022, 22(1): 613.
Chen X, Chen G, Guo S, et al. SlSAMS1 enhances salt tolerance through regulation DNA methylation of SlGI in tomato[J]. Plant Science, 2023, 335: 111808.
Cheng J, Niu Q, Zhang B, et al. Downregulation of RdDM during strawberry fruit ripening[J]. Genome biology, 2018, 19: 1-14.
Cokus S J, Feng S, Zhang X, et al. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning[J]. Nature, 2008, 452(7184): 215-219.
Han M L, Yin J, Zhao Y H, et al. How the color fades from Malus halliana flowers: transcriptome sequencing and DNA methylation analysis[J]. Frontiers in Plant Science, 2020, 11: 576054.
Huang H, Liu R, Niu Q, et al. Global increase in DNA methylation during orange fruit development and ripening[J]. Proceedings of the National Academy of Sciences, 2019, 116(4): 1430-1436.
Law J A, Jacobsen S E. Establishing, maintaining and modifying DNA methylation patterns in plants and animals[J]. Nature Reviews Genetics, 2010, 11(3): 204-220.
Lister R, O’Malley R C, Tonti-Filippini J, et al. Highly integrated single-base resolution maps of the epigenome in Arabidopsis[J]. Cell, 2008, 133(3): 523-536.
Liu R, How-Kit A, Stammitti L, et al. A DEMETER-like DNA demethylase governs tomato fruit ripening[J]. Proceedings of the National Academy of Sciences, 2015, 112(34): 10804-10809.
Liu R, Lang Z. The mechanism and function of active DNA demethylation in plants[J]. Journal of integrative plant biology, 2020, 62(1): 148-159.
Manning K, Tör M, Poole M, et al. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening[J]. Nature genetics, 2006, 38(8): 948-952.
Matzke M A, Mosher R A. RNA-directed DNA methylation: an epigenetic pathway of increasing complexity[J]. Nature Reviews Genetics, 2014, 15(6): 394-408.
Sun S, Zhu J, Guo R, et al. DNA methylation is involved in acclimation to iron‐deficiency in rice (Oryza sativa)[J]. The Plant Journal, 2021, 107(3): 727-739.
Xu J, Xiong L, Yao J L, et al. Hypermethylation in the promoter regions of flavonoid pathway genes is associated with skin color fading during ‘Daihong’apple fruit development[J]. Horticulture Research, 2024: uhae031.
Zheng G, Hu S, Cheng S, et al. Factor of DNA methylation 1 affects woodland strawberry plant stature and organ size via DNA methylation[J]. Plant Physiology, 2023, 191(1): 335-351.
Zhu J, Wang Y, Wang Q, et al. The combination of DNA methylation and positive regulation of anthocyanin biosynthesis by MYB and bHLH transcription factors contributes to the petal blotch formation in Xibei tree peony[J]. Horticulture Research, 2023, 10(7): uhad100.
文章来源:伯远生物公众号