本文内容速览:

水稻是世界上重要的粮食作物之一。水稻生产在农业中占据着极其重要的地位。稻田杂草与水稻争夺养分、水分和阳光等资源,限制了水稻的生长空间和养分吸收,导致水稻生长受到严重阻碍,甚至引发生长畸形或减产。此外,稻田杂草还为多种病虫害提供了理想的栖息地,进一步加剧了病虫害对水稻的危害程度。因此,有效地控制稻田杂草对于维护水稻生产的稳定性和提升产量至关重要。
目前,控制稻田杂草的方法多种多样,包括物理防治、生物防治和化学除草剂等。在这些方法中,化学除草剂作为一种高效、易用、成本相对较低的除草工具,在现代农业中得到了广泛的应用。根据作用机理的不同,化学除草剂可以分为草甘膦、草铵膦、对羟基苯基丙酮酸双氧化酶(HPPD)抑制剂、乙酰乳酸合成酶(ALS)抑制剂、乙酰辅酶A羧化酶(ACCase)抑制剂、二硝基苯胺除草剂等(图1)。然而,随着除草剂的大量应用,抗除草剂杂草的增多以及土壤中除草剂残留量的升高等问题被暴露出来,这些问题限制了作物的轮作和产量的提高。因此,迫切需要培育抗除草剂的水稻品种。在本篇推文中,伯小远向大家介绍了常见的化学除草剂,并结合文献案例针对这些除草剂的作用机理进行了详细说明。
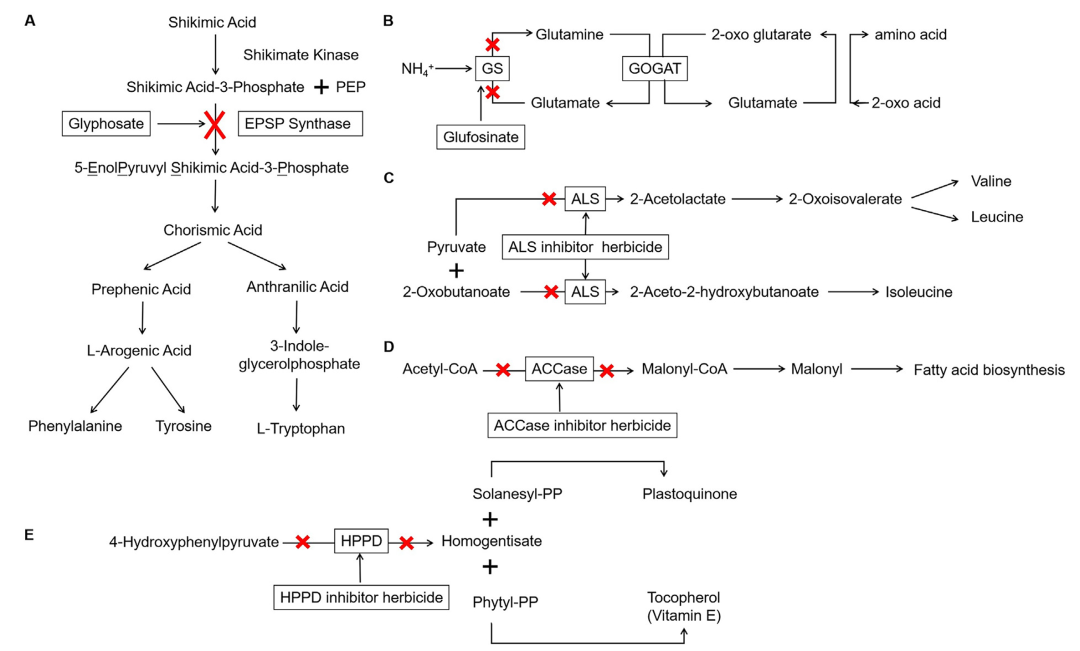
图1 除草剂的作用机理(Jin et al., 2021)。
草甘膦是一种有机磷除草剂,主要通过抑制植物体内的烯醇丙酮基莽草素磷酸合成酶(EPSPS),从而抑制莽草素向苯丙氨酸、酪氨酸及色氨酸的转化,使植物蛋白质合成受到干扰,从而导致植物死亡。目前,大多数对草甘膦具有抗性的杂草被鉴定出是由于EPSPS发生突变引起的。2016年9月,中国科学院遗传与发育生物学研究所高彩霞课题组和李家洋课题组合作在Nature Plants杂志上发表了一篇题为“Gene replacements and insertions in rice by intron targeting using CRISPR–Cas9”的研究论文。作者利用非同源末端连接(NHEJ)修复方式在水稻中建立了基于CRISPR/Cas9技术的基因替换以及基因定点插入体系。该研究成功实现了水稻内源OsEPSPS基因保守区两个氨基酸的定点替换(T102I和P106S, TIPS),在T0代获得了TIPS定点替换的杂合体(T0-RP4、T0-RP6、T0-RP8、T0-RP10、T0-IP8和T0-IP11),该杂合植株对草甘膦具有抗性,并且能够稳定遗传到下一代,不过该突变形式会导致纯合致死。

图2 EPSPS基因TIPS突变赋予植物草甘膦抗性(Li et al., 2016)。(a)通过RT-PCR检测TIPS突变株系是否存在EPSPS的剪接;(b)通过qRT-PCR检测叶片中EPSPS的相对表达水平;(c)T0代TIPS突变株系草甘膦抗性试验结果;(d)T1代TIPS突变株系草甘膦抗性试验结果,(1)第二个外显子缺失的突变株系,(2)野生型,(3)TIPS突变株系T0-RP4,(4)TIPS突变株系T0-IP8。
此外,有研究发现阿根廷野生杂草绿穗苋(Amaranthus hybridus)普遍具有高草甘膦抗性,进一步解析发现是由于TAP-IVS突变(T102I、A103V和P106S)所引起的(Perotti et al., 2019)。2022年9月,中国农业大学生物学院陈其军课题组在Molecular Plant杂志上发表了一篇题为“Optimized prime editing efficiently generates glyphosate-resistant rice plants carrying homozygous TAP-IVS mutation in EPSPS”的研究论文。在该研究中,作者通过在水稻原生质体中对EPSPS基因TAP位点进行引导编辑测试,利用优化后的引导编辑器成功获得能够稳定遗传的纯合和杂合的TAP-IVS突变体。草甘膦抗性测试结果表明,相比于野生型,纯合的TAP-IVS突变体能够明显增强草甘膦的抗性。综上所述,该研究利用优化的引导编辑技术高效诱导了EPSPS基因的纯合和杂合TAP-IVS突变,为水稻的非转基因草甘膦抗性育种奠定了坚实的基础。
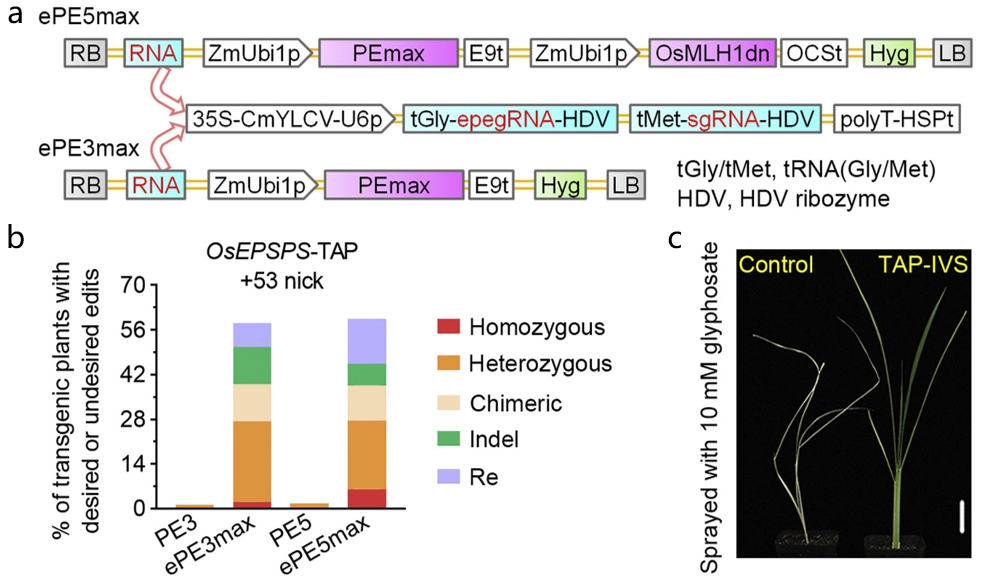
图3 优化后的编辑器提高了编辑效率,并成功产生了可遗传的TAP-IVS突变,使水稻具备了草甘膦抗性(Jiang et al., 2022)。(a)两种优化后的编辑器结构;(b)两种优化后的编辑器诱导产生的突变形式二代测序分析;(c)EPSPS中的TAP-IVS突变赋予了水稻草甘膦抗性。
草铵膦是一种非选择性、广谱的除草剂,其特点在于具有高效的除草活性、广泛的除草范围以及良好的环境适应性。该除草剂通过作用于植物的谷氨酰胺合成酶(GS)来抑制其活性,进而导致植物体内氨基酸积累和活性氧的释放。这些效应进一步抑制了植物光合作用的进行,引起脂质过氧化,最终导致植物死亡。2023年8月,中国科学院合肥物质院智能所离子束研究中心吴跃进课题组在Plant Biotechnology Journal杂志上发表了一篇题为“Precision editing of GLR1 confers glufosinate resistance without yield penalty in rice”的研究论文。作者利用重离子束诱变技术成功在大田中筛选出具有草铵膦抗性的突变体glr1和glr2。在该研究中,作者选择glr1突变体进行研究,草铵膦抗性测试结果表明,glr1突变体相比于野生型具有较低的H2O2、丙二醛(MDA)含量以及较高的GS、超氧化物歧化酶(SOD)和过氧化氢酶(CAT)活性。
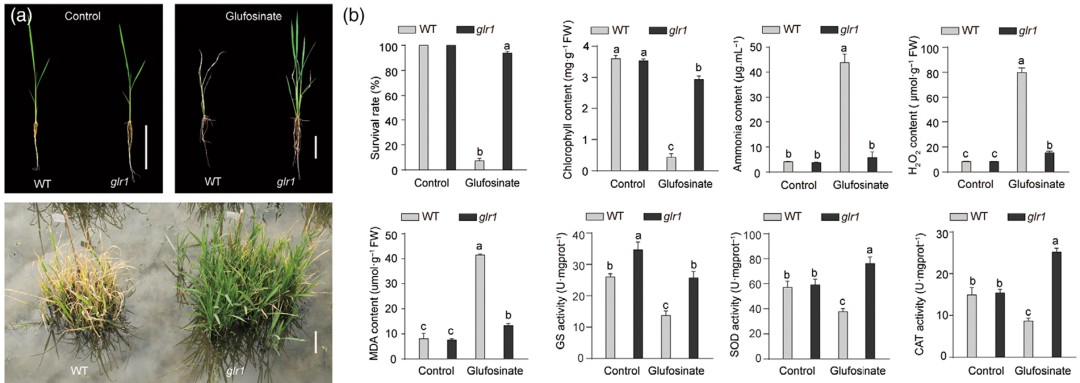 图4 GLR1调节水稻中的草铵膦抗性(Ren et al., 2022)。(a)草铵膦喷施前后WT和glr1的表型分析;(b)幼苗成活率、叶绿素含量、氨含量、H2O2含量、MDA含量、GS活性、SOD活性和CAT活性分析。
图4 GLR1调节水稻中的草铵膦抗性(Ren et al., 2022)。(a)草铵膦喷施前后WT和glr1的表型分析;(b)幼苗成活率、叶绿素含量、氨含量、H2O2含量、MDA含量、GS活性、SOD活性和CAT活性分析。
作者进一步通过图位克隆确定,glr1突变体是由GLR1基因的第一个外显子中G的缺失所导致的。接着,利用CRISPR-Cas9介导的基因编辑技术成功获得glr1-ko1和glr1-ko2株系,这两个基因编辑株系表现出与glr1突变体相似的抗草铵膦表型。RNA-seq分析显示,glr突变体中参与氨和活性氧清除相关基因(OsGS1、OsCYP51G3和OsCATA)表达上调。尽管这些glr1突变体表现出草铵膦抗性,但是产量受到了一定的影响。于是作者寻找是否存在GLR1的理想等位基因变异形式,对GLR1的不同区域进行编辑,获得glr1-ko4株系。研究发现GLR1的DNA结合域和中间/阻遏结构域的保留可以维持草铵膦抗性的同时保证产量。综上所述,考虑到GLR1在植物中的进化和功能保守性,将GLR1的应用扩展到其他重要作物可能会提高它们对除草剂的耐受性,而不会对产量造成显著影响。
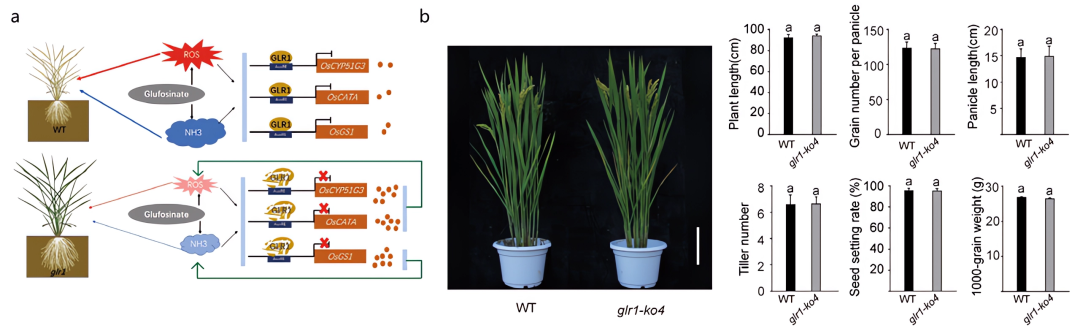
图5 GLR1调节水稻中的草铵膦抗性(Ren et al., 2022)。(a)GLR1在调控草铵膦耐药性方面的工作模型;(b)WT和glr1-ko4植株的农艺性状分析。
HPPD抑制剂类除草剂主要通过抑制HPPD的活性,使对羟基苯基丙酮酸转化为尿黑酸的过程受阻,导致生育酚及质体醌无法正常合成,进而影响类胡萝卜素的生物合成,促使植物分生、新生组织产生白化症状,最终导致植株死亡。2023年4月,江苏里下河地区农业科学研究所李爱宏课题组在Plant Communications杂志上发表了一篇题为“CRISPR-Cas9-mediated editing of OsHPPD 3′ UTR confers enhanced resistance to HPPD-inhibiting herbicide in rice”的研究论文。作者在OsHPPD基因的3′ UTR上选择了10个靶位点,利用CRISPR-Cas9介导的基因编辑技术成功获得了基因编辑的水稻植株。研究中选择了适宜浓度的硝磺草酮(MST),对T2代的基因编辑株系进行喷施,结果显示仅有两个株系(TS8-2#和TS8-8#)能够存活,而其他133个T2株系和野生型材料均已死亡。进一步对TS8-2#和TS8-8#的sgRNA位点进行测序,发现这两个抗性株系是在同一个位点发生了突变。具体而言,TS8-2#是纯合子,而TS8-8#是杂合子。随后,研究人员从其后代中筛选出两个无转基因成分的纯合子编辑系(TS8-2#-10和TS8-8#-6),用于后续分析。研究结果显示,这两个株系对HPPD抑制剂的抗性分别是野生型抗性的4.8倍和3.7倍,并且在产量和株高等农艺性状上与野生型之间没有明显区别。综上所述,该研究通过对水稻OsHPPD基因3′ UTR进行靶位点的基因编辑,创制了水稻对HPPD抑制性除草剂具有抗性的新种质材料,并且不影响其他农艺性状。

图6 通过CRISPR-Cas9介导的OsHPPD 3′ UTR编辑,生成抗HPPD抑制除草剂的新水稻种质(Wu et al., 2023)。(A)OsHPPD位点示意图。深灰色,外显子;浅灰色,UTR;(B)OsHPPD 3′ UTR中目标位点(TS1–TS10)的位置;(C)T0代株系Sanger测序结果;(D)10个籼稻品种和10个粳稻品种在不同浓度MST处理下存活率分析;(E)用适宜浓度的MST处理OsHPPD编辑株系;(F)TS8-2#和TS8-8#株系序列分析;(G)T2代株系转基因检测;(H)TS8-2#-10和TS8-8#-6纯合T3代株系的序列分析;(I)MST处理的YG3012(WT)、TS8-2#-10和TS8-8#-6植株的表型;(J)YG3012(WT)、TS8-2#-10和TS8-8#-6植株在温室处理14 d后对不同MST剂量的响应曲线;(K)YG3012(WT)、TS8-2#-10和TS8-8#-6的主要农艺性状。
除了对3′ UTR区进行编辑外,还可以通过片段倒位和片段重复的方法来替换目的基因附近的高表达基因启动子,从而实现基因敲高,最终提高水稻对除草剂的抗性。2021年11月,中国农业大学姜临建课题组在Nature Plants杂志上发表了一篇题为“A Donor DNA-free CRISPR/Cas-based Approach to Gene Knock-up in Rice”的研究论文。作者构建了双靶点CRISPR载体,将Ubiquitin2启动子进行重复,从而实现Ubiquitin2基因的启动子驱动HPPD基因的表达,大幅提高了水稻HPPD基因的内源表达水平。这使得水稻植株表现出了预期的抗除草剂性状。详情可参阅“基因敲高——通过基因编辑改善作物农艺性状”的相关内容。
ALS抑制剂类除草剂是一类常见的广谱除草剂,其作用机制是抑制植物体内的ALS酶活性,阻断了植物对于氨基酸苯丙氨酸的合成途径,导致植物无法正常生长并最终死亡。2020年11月,华南师范大学唐晓艳课题组和深圳市作物分子设计育种研究院邓兴旺课题组合作在The Crop Journal杂志上发表了一篇题为“Trp548Met mutation of acetolactate synthase in rice confers resistance to a broad spectrum of ALS-inhibiting herbicides”的研究论文。作者通过EMS化学诱变,筛选获得了一个对咪唑啉酮(IMI)除草剂具有抗性的突变体植株,鉴定分析发现突变体抗除草剂的表型是由于OsALS基因中的Trp548(TGG)被Met(ATG)取代所导致的。为了研究W548M突变是否会对水稻生长发育造成影响,作者进一步通过田间实验来观察突变株系JTD-001和野生型株系HHZ在相同条件下的形态生理特征,结果发现JTD-001和HHZ在植株形态、叶片形态和产量等方面没有显著差异。综上,结果表明W548M突变不影响水稻生长发育和产量。
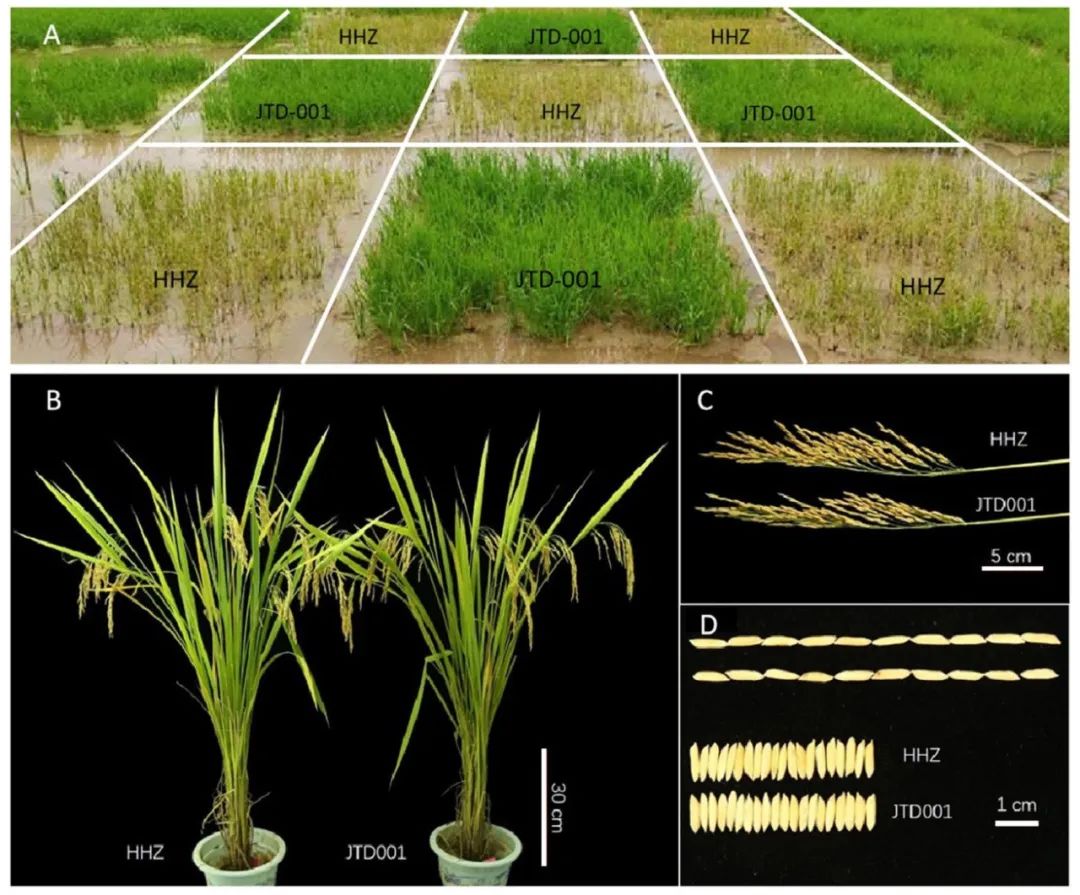
图7 JTD-001与HHZ的表型观察(Chen et al., 2021)。(A)喷施甲咪唑烟酸后JTD-001与HHZ的幼苗;(B-D)成熟期阶段JTD-001和HHZ的植物结构(B)、穗(C)和种子(D)的比较。
S627N突变已被报道具有除草剂抗性。在本研究中,作者为了比较W548M和S627N在除草剂抗性水平的差异,用不同类型和不同浓度的ALS抑制剂类除草剂喷施HHZ、W548M突变株系(JTD-001)和S627N突变株系(KS-2)。结果显示,相比于S627N,W548M对广谱ALS抑制剂具有较高的抗性。

图8 JTD-001、HHZ和KS-2在各种ALS抑制除草剂的抗性谱和抗性水平方面的比较(Chen et al., 2021)。
随后作者为了检验W548M突变在不同水稻背景中是否稳定,通过回交的方法将JTD-001上的W548M基因座转移到其它72个水稻品种中,结果显示无论水稻背景如何改变,W548M的除草剂抗性均稳定。综上所述,作者推断W548M水稻具有巨大的应用前景,W548M水稻突变体有望为杂草控制提供强有力的工具。

图9 W548M基因座转移到其他水稻品种后仍然具有除草剂抗性(Chen et al., 2021)。(A)喷施水后的不同品种突变株系表型;(B)喷施甲咪唑烟酸后的不同品种突变株系表型。
ACCase抑制剂类除草剂的作用原理是通过抑制植物体内的ACCase活性来实现的。ACCase参与植物脂肪酸的生物合成,负责将乙酰辅酶A转化为丙酸辅酶A,是脂肪酸合成的关键酶之一。当ACCase受到抑制时,植物无法正常合成脂肪酸,导致细胞膜的组成受到影响,进而破坏了细胞结构和功能,最终导致植物的生长停止和死亡。2020年1月,安徽农业大学吴德祥课题组在Plant Biotechnology Journal杂志上发表了一篇题为“A CRISPR-Cas9-mediated domain-specific base-editing screen enables functional assessment of ACCase variants in rice”的研究论文。为了寻找水稻OsACC抗性位点并创制抗性材料,作者在OsACC的CT结构域上共设计了141条sgRNA,并结合课题组前期开发的基因编辑工具,构建了三个碱基编辑文库。然后,这些文库被导入到肥粳2020中,经过抗生素以及ACCase抑制剂的筛选,最终从三个文库中共鉴定出I1879V、W2125S和C2186R这三种突变形式的抗性事件。进一步研究发现,与野生型相比,W2125S和纯合C2186R植株出现严重的生长迟缓和完全不育的表型,而纯合I1879V和杂合C2186R植株不仅具有除草剂抗性,而且在株高、旗叶长度和结实率等性状方面与野生型相比没有显著差异。综上所述,作者推断OsACC基因的I1879V突变可能是具有ACCase抑制剂类除草剂抗性的理想OsACC等位基因。
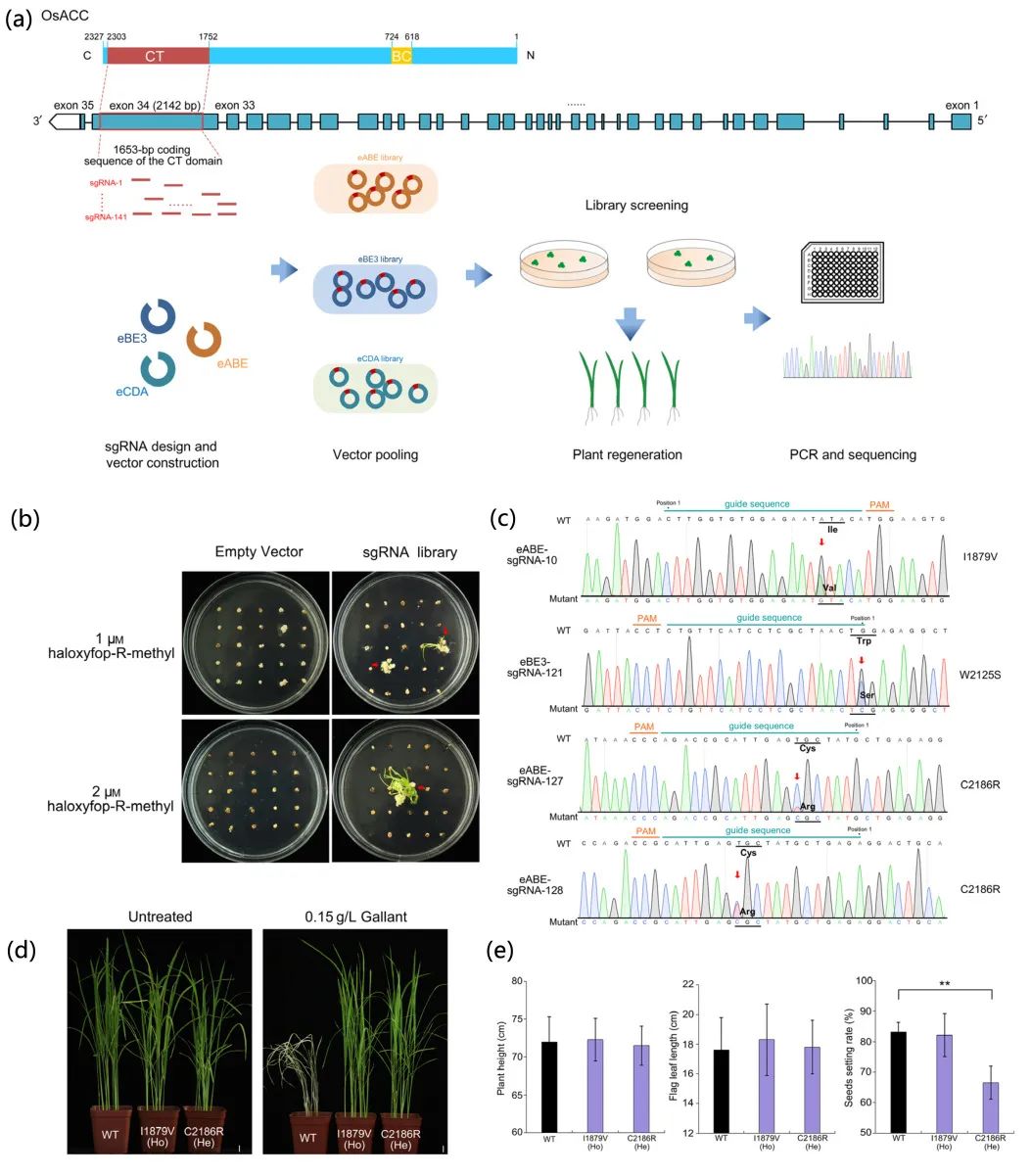
图10 筛选赋予水稻除草剂抗性的ACCase氨基酸(Liu et al., 2022)。(a)水稻OsACC碱基编辑筛选示意图。OsACC的蛋白质和基因结构如上图所示。CT,羧基转移酶结构域;BC,生物素羧化酶结构域。在CT结构域的编码序列中,选择20 bp序列后跟5′-NGG PAM作为引导RNA序列。将引导序列的退火寡核苷酸的等摩尔混合物连接到pHUC411衍生的植物BE载体中,以汇集碱基编辑文库。收集具有显著抗除草剂性的枝条和愈伤组织进行基因分型;(b)通过筛选OsACC的碱基编辑文库,选择具有代表性的抗性事件;(c)从碱基编辑文库中筛选出的突变体的基因型;(d)适宜浓度的盖草能喷施3-5叶期OsACC突变体的表型;(e)OsACC突变体在成熟阶段的农艺性状。
二硝基苯胺除草剂的作用原理是二硝基苯胺与微管蛋白结合,从而干扰微管的结构和功能,导致微管的异常聚集和破坏。这种干扰会影响细胞的正常生理过程,如细胞分裂、细胞形态维持等,最终导致植物的生长受到阻碍甚至死亡。2020年4月,中国农科院植保所周焕斌课题组在Plant Biotechnology Journal杂志上发表了一篇题为“Developing a novel artificial rice germplasm for dinitroaniline herbicide resistance by base editing of OsTubA2”的研究论文。作者利用课题组前期开发的基因编辑工具rBE14,对水稻OsTubA2基因进行了定点编辑,成功获得63个独立株系,其中有8个株系存在M268T突变形式。进一步对T1代株系进行检测,结果发现T1代存在无转基因株系、纯合株系和杂合株系。选择合适浓度的除草剂(二甲戊灵和三氟灵)分别对野生型和M268T突变株系进行喷施,结果显示,与野生型相比,纯合株系表现出较高的除草剂抗性,并且这种突变形式能够在后代中稳定遗传。此外,作者还对野生型植株和M268T突变株的农艺性状进行观察,发现两者在植株形态以及产量等农艺性状上并无明显差异。综上所述,OsTubA2基因中的M268T突变赋予了水稻对二硝基苯胺除草剂抗性,而不会诱导适应性成本。
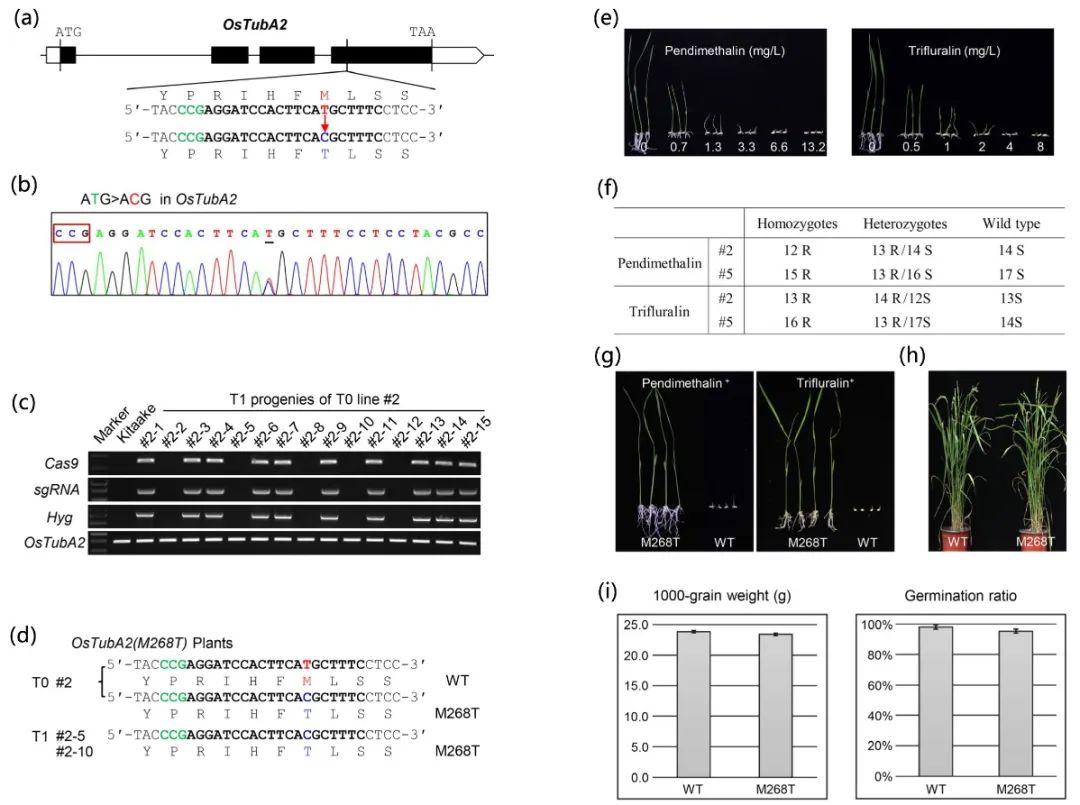
图11 通过精准编辑OsTubA2基因,生成抗二硝基苯胺除草剂的新型人工水稻种质(Liu et al., 2021)。(a)水稻OsTubA2基因中的靶位点。外显子由黑框表示;(b)T0转基因系中rBE14编辑的OsTubA2等位基因的代表性Sanger测序色谱图。核苷酸变化带有下划线,PAM序列标记在框中;(c)分离出无T-DNA插入的T1植物。通过使用基因特异性引物(Cas9、Hyg、sgRNA)进行PCR扩增来检测单个基因的存在和缺失;(d)杂合子T0代#2及其纯合子T1代#2-5和#2-10的基因型;(e)野生型Kitaake种子的除草剂耐受性测定。野生型Kitaake种子分别辅以0、0.7、1.3、3.3、6.6、13.2 mg/L二甲戊灵和0、0.5、1.0、2.0、4.0、8.0 mg/L三氟灵;(f)抗二硝基苯胺类除草剂水稻T1代的基因型-表型关联分析。R = 抗病;S = 易感;(g)二甲戊灵和三氟灵处理下纯合OsTubA2(M268T)幼苗的表型;(h)抽穗期野生型和OsTubA2(M268T)植物的植物形态;(i)纯合OsTubA2(M268T)T2种子的1000粒重和发芽率统计。
稻田杂草对水稻的危害不可小觑,化学除草一直是主要控制手段之一。然而,由于一些杂草与水稻在分类学和生理学上相似,因此对稻田杂草的防治一直是一大挑战。在本篇推文中,伯小远详细介绍了几种当前主流的除草剂,分别从它们的作用机理和对应靶基因的抗性突变位点两个方面进行了分析。对于优化除草剂的使用策略和培育更耐除草剂的水稻品种都具有重要的指导意义。
References:
Chen L, Gu G, Wang C, et al. Trp548Met mutation of acetolactate synthase in rice confers resistance to a broad spectrum of ALS-inhibiting herbicides[J]. The Crop Journal, 2021, 9(4): 750-758.
Jiang Y, Chai Y, Qiao D, et al. Optimized prime editing efficiently generates glyphosate-resistant rice plants carrying homozygous TAP-IVS mutation in EPSPS[J]. Molecular Plant, 2022, 15(11): 1646-1649.
Jin M, Chen L, Deng X W, et al. Development of herbicide resistance genes and their application in rice[J]. The Crop Journal, 2022, 10(1): 26-35.
Li J, Meng X, Zong Y, et al. Gene replacements and insertions in rice by intron targeting using CRISPR–Cas9[J]. Nature plants, 2016, 2(10): 1-6.
Liu L, Kuang Y, Yan F, et al. Developing a novel artificial rice germplasm for dinitroaniline herbicide resistance by base editing of OsTubA2[J]. Plant biotechnology journal, 2021, 19(1): 5.
Liu X, Qin R, Li J, et al. A CRISPR‐Cas9‐mediated domain‐specific base‐editing screen enables functional assessment of ACCase variants in rice[J]. Plant Biotechnology Journal, 2020, 18(9): 1845.
Lu Y, Wang J, Chen B, et al. A donor-DNA-free CRISPR/Cas-based approach to gene knock-up in rice[J]. Nature plants, 2021, 7(11): 1445-1452.
Perotti V E, Larran A S, Palmieri V E, et al. A novel triple amino acid substitution in the EPSPS found in a high‐level glyphosate‐resistant Amaranthus hybridus population from Argentina[J]. Pest management science, 2019, 75(5): 1242-1251.
Ren Y, Liu B, Jiang H, et al. Precision editing of GLR1 confers glufosinate resistance without yield penalty in rice[J]. Plant Biotechnology Journal, 2023, 21(12): 2417.
Wu Y, Xiao N, Cai Y, et al. CRISPR-Cas9-mediated editing of the OsHPPD 3′ UTR confers enhanced resistance to HPPD-inhibiting herbicides in rice[J]. Plant communications, 2023, 4(5).













