本文内容速览:
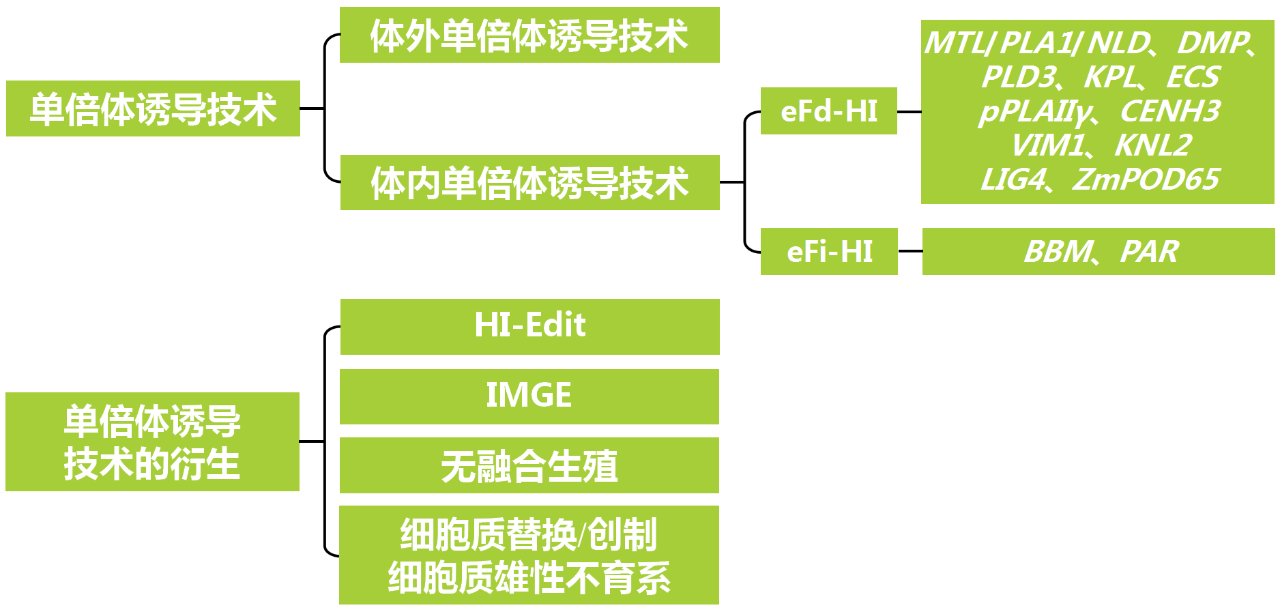
根据诱导方式,可将单倍体诱导技术分为体外单倍体诱导技术和体内单倍体诱导技术。体外单倍体诱导技术依赖于特定的组织培养条件,使配子体重编程为单倍体胚或幼苗,这种方法成本高、劳动强度大、对基因型的依赖程度高,且组培过程中易发生突变,因此应用范围较狭窄。相比而言,目前应用更为广泛的是体内单倍体诱导技术,按照诱导方式不同可将其分为远缘杂交诱导、花粉诱导、着丝粒介导、诱导系诱导和基因编辑诱导等,按照诱导过程是否进行受精则可将其分为卵细胞受精依赖性单倍体诱导(egg cell fertilization-dependent HI, eFd-HI)和卵细胞受精非依赖性单倍体诱导(egg cell fertilization-independent HI, eFi-HI)(Quiroz et al., 2024),下面伯小远以第二种分类方式带大家回顾一下单倍体诱导相关的关键基因。
eFd-HI
基于破坏配子间的通讯或融合的eFd-HI
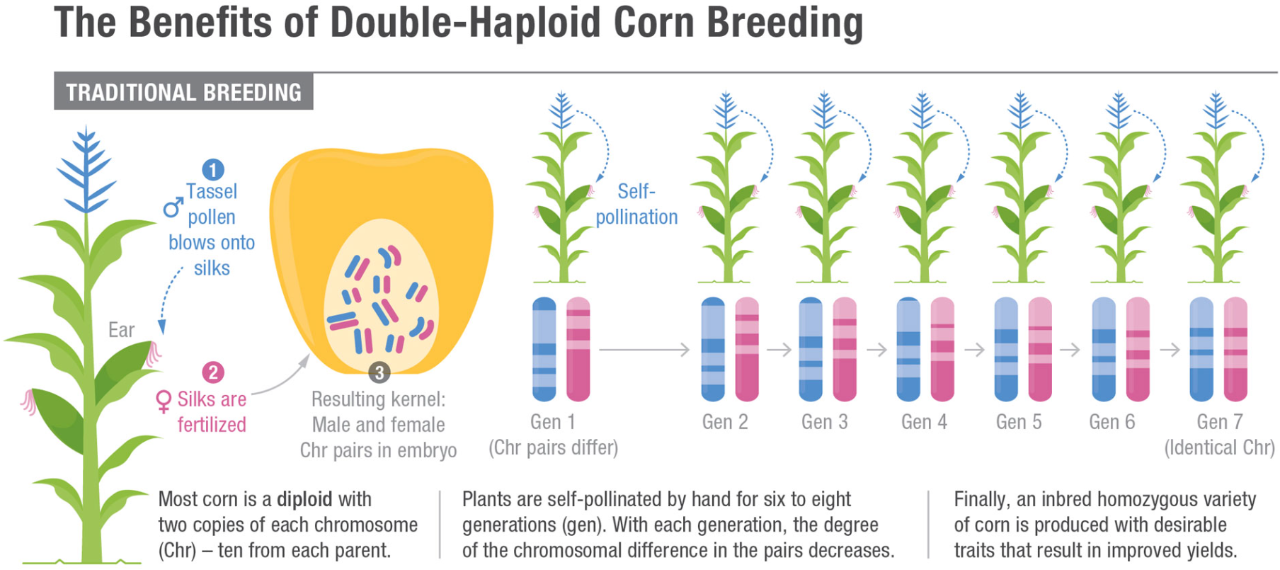
单倍体诱导系已在多个物种中应用,最出名的例子是玉米突变体Stock6作为单倍体诱导系与不同的母本杂交时可产生2-3%的单倍体(Coe, 1959)。在玉米中,传统创制纯系的方法需要6-7代甚至更多,而通过单倍体诱导技术创制纯系则只需要2代。目前,这项技术已经成为国内外玉米育种单位普遍应用的核心技术。然而,玉米中的单倍体诱导机制已困扰研究者们60余年。
图1 在玉米中通过单倍体诱导技术创制纯系能大大缩短育种周期。图片来源:Lucy reading。
除了MTL/ZmPLA1/NLD基因,另一个关键基因ZmDMP被发现与玉米非Stock6材料的单倍体诱导有关,其等位基因的突变导致蛋白在第一个跨膜结构域中有一个氨基酸的替换(Zhong et al., 2019)。ZmDMP基因在其它物种中功能保守,目前在双子叶植物如拟南芥(Zhong et al., 2020)、蒺藜苜蓿(Wang et al., 2022)、番茄(Zhong et al., 2022)、烟草(Zhang et al., 2022)、西瓜(Tian et al., 2023; Chen et al., 2023)和大豆(Yu et al., 2024)中通过敲除ZmDMP的同源基因,也建立了这些物种的单倍体诱导体系。
另外,花粉特异性基因ZmPLD3的敲除也被发现可诱导玉米产生单倍体后代。pld3突变体的花粉管生长发生改变,破坏了与雌配子体的相互作用,导致父本染色体在受精过程中无法整合到雌配子中(Li et al., 2021)。
KPL(Jacquier et al., 2023)、ECS(Mao et al., 2023; Zhang et al., 2023)和pPLAIIγ(Jang et al., 2023)是另外一些潜在的参与单倍体诱导的候选基因,研究者们推测这些基因的作用机制与上述基因MTL/ZmPLA1/NLD、ZmDMP和ZmPLD3类似,均涉及破坏配子间的通讯或融合。
基于着丝粒功能改变的eFd-HI
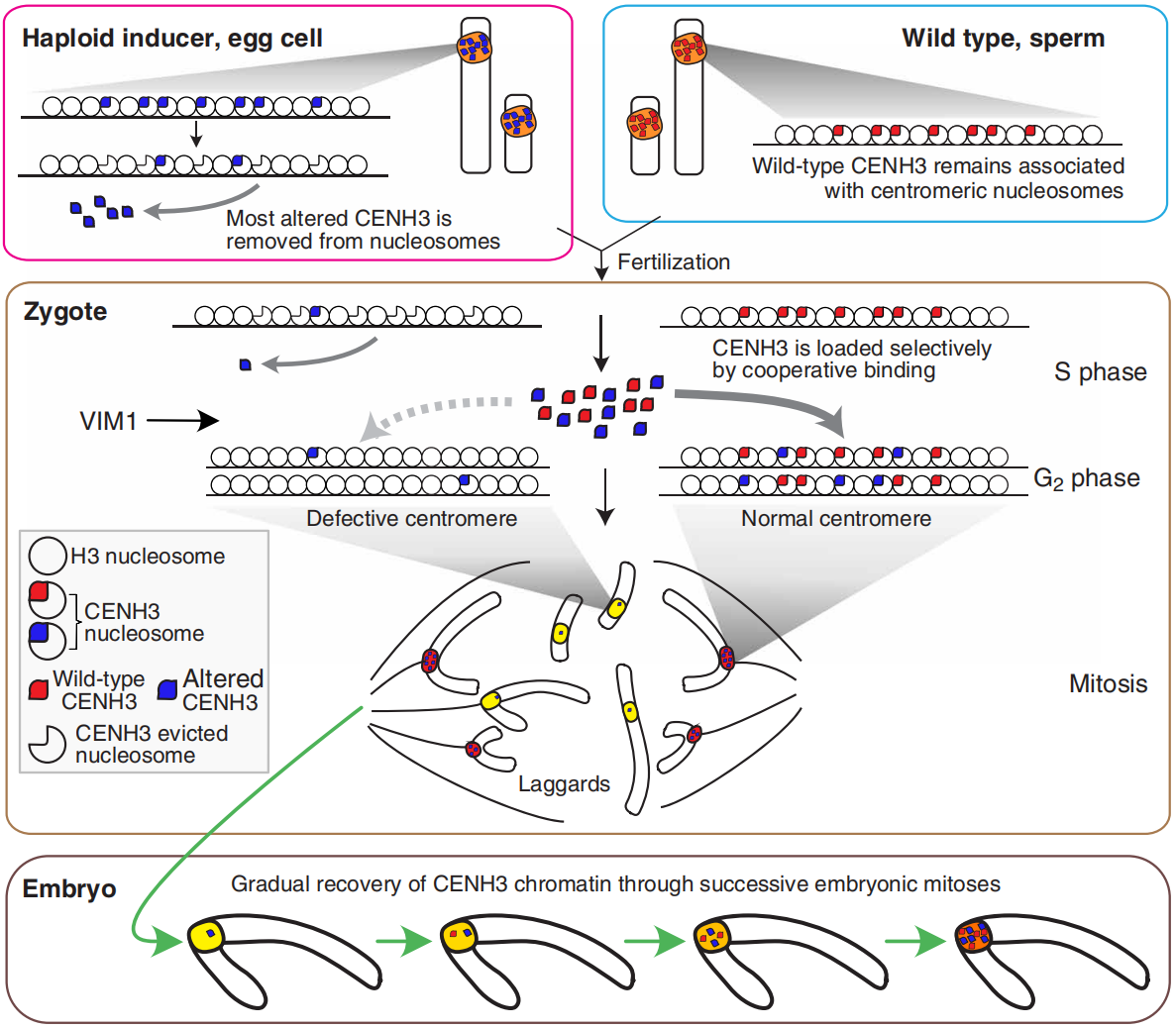
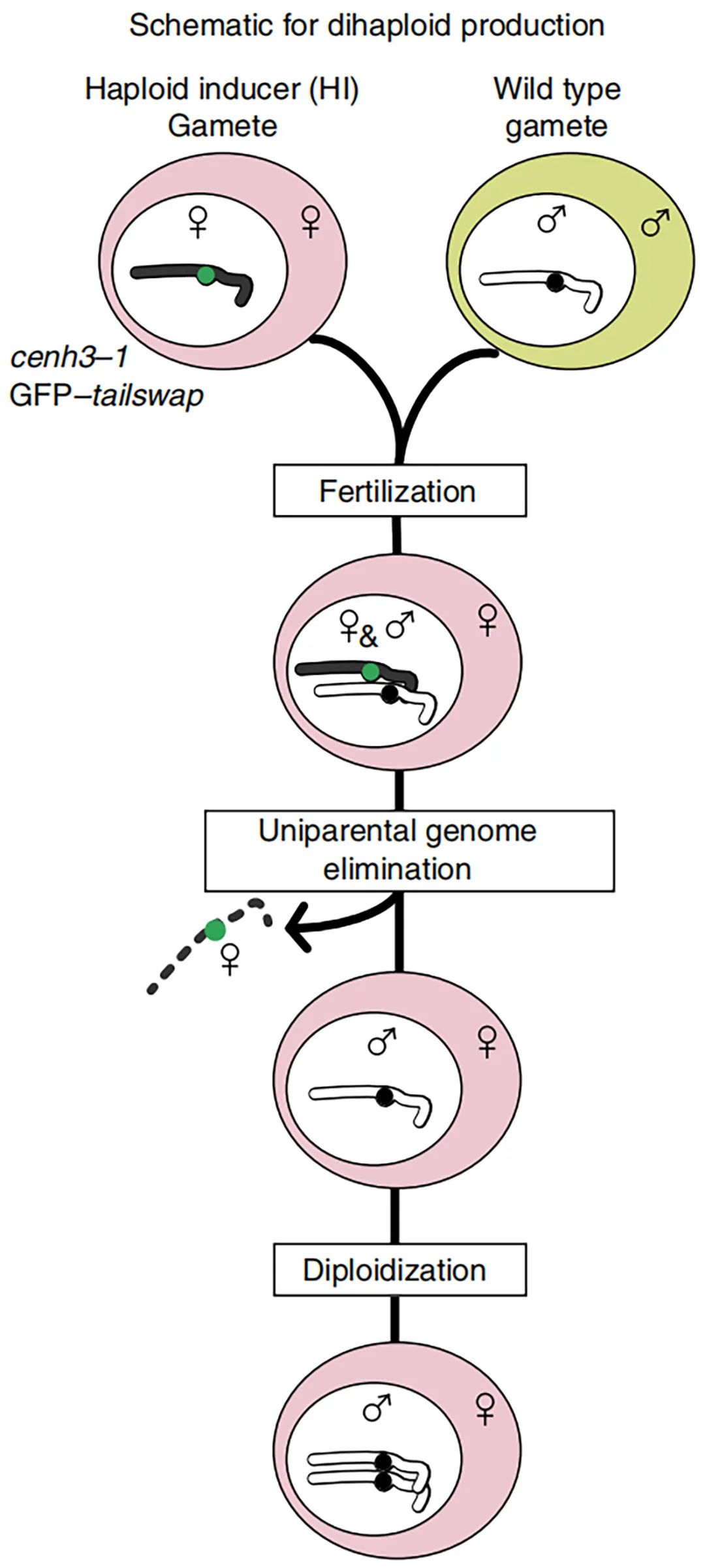
中心型组蛋白3(CENH3)基因是另一种创制单倍体诱导系的关键基因。CENH3为核小体组蛋白H3的变体,存在于着丝粒中,通过在有丝分裂和减数分裂期间确保染色体的准确分离来使植物保持二倍体状态。如果以CENH3表达发生改变的植株为亲本与野生型杂交,可使其基因组在合子中消失,产生只保留野生型基因组的单倍体植株(图2)。该基因的功能在各个物种中保守存在,在拟南芥(Ravi and Chan, 2010)、玉米(Kelliher et al., 2016)和小麦(Lv et al., 2020)中均有相关报道。除了CENH3基因,与着丝粒功能有关的VIM1基因(Marimuthu et al., 2021)、KNL2基因(Ahmadli et al., 2023)突变也可增加单倍体诱导率。
图2 拟南芥中CENH3介导的单倍体诱导模型(Marimuthu et al., 2021)。在成熟的母细胞中,CENH3变体会被选择性地从染色质中移除,但野生型中的CENH3不受影响;杂交后,CENH3弱化的母本着丝粒会与父本野生型着丝粒竞争加载CENH3,由于协同结合效应,CENH3会优先加载在野生型亲本的着丝粒上;在随后的有丝分裂过程中,单倍体诱导系的染色体由于其弱着丝粒而不能正常分离;其中,VIM1介导的泛素化或甲基化可以影响CENH3的稳定性,并且VIM1有利于CENH3的加载。
基于DNA/染色体损伤的eFd-HI
LIG4是真核生物中非同源末端连接(NHEJ)系统的组成部分,突变LIG4基因被证明可提高其他诱导系统的单倍体诱导率(Tan et al., 2015)。
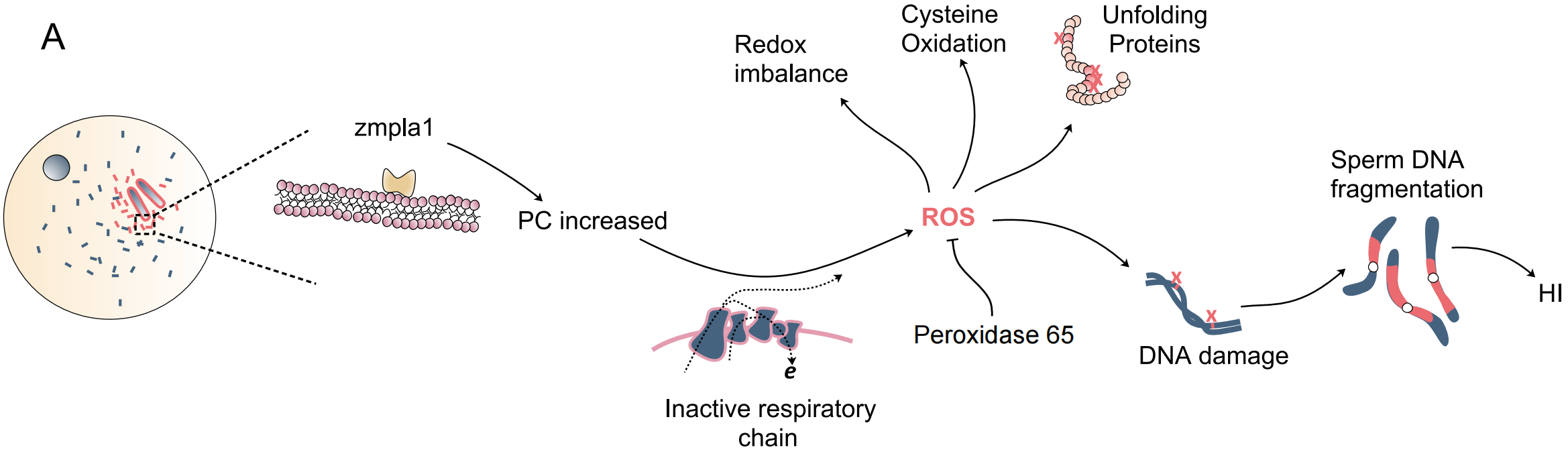
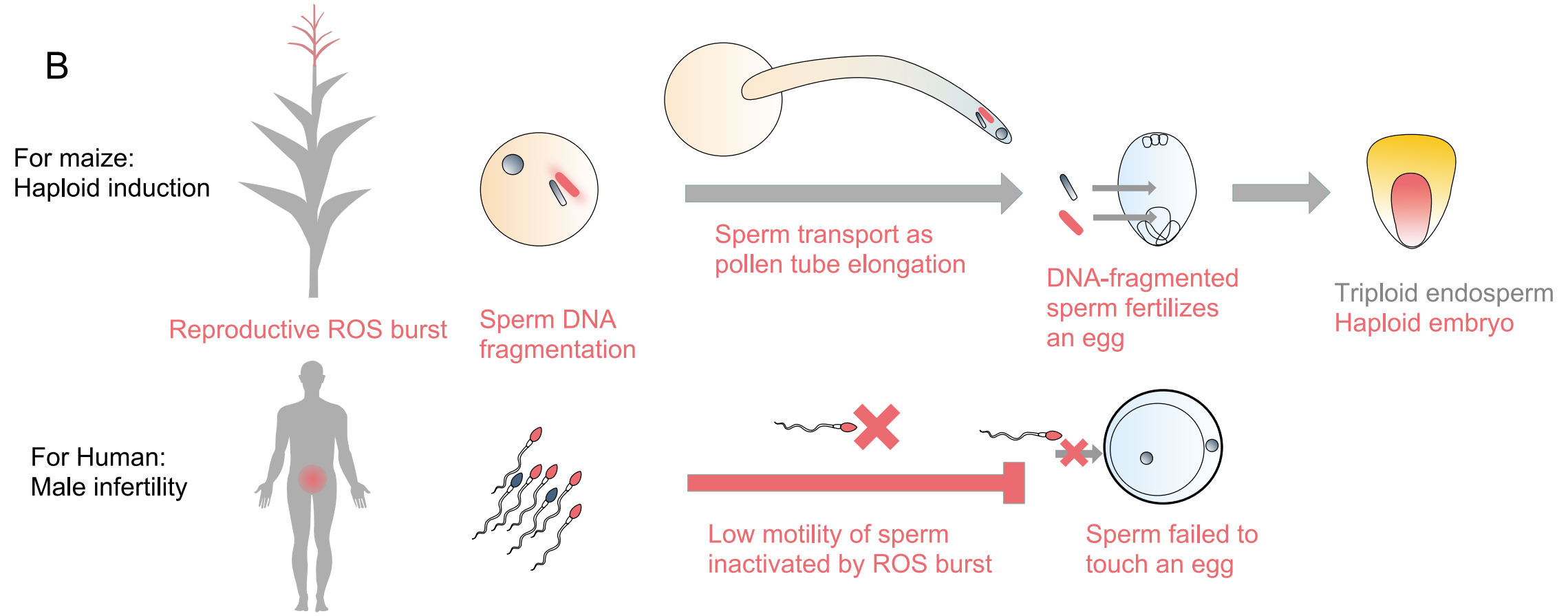
在玉米磷脂酶介导的单倍体诱导系统中,活性氧(ROS)的爆发会破坏染色体的稳定性,导致精细胞染色体片段化,进而引起单倍体诱导的发生。基于这一原理,可以推测与活性氧产生相关的基因可以诱导单倍体的发生。已有研究证实通过编辑过氧化物酶ZmPOD65基因的植株可作为单倍体诱导系(Jiang et al., 2022)。利用此原理,使用甲硫咪唑(过氧化物酶抑制剂)或磷脂酰胆碱(ROS诱导剂)处理花粉会导致ROS的爆发和DNA的断裂,从而引发单倍体诱导现象(Jiang et al., 2022)。
图3 ROS导致染色体被破坏进而诱导单倍体的产生(Jiang et al., 2022)。(A)精细胞的ZmPLA1失活,导致周边卵磷脂积累,使线粒体发生紊乱导致ROS爆发,使精细胞的染色体发生片段化,最终形成单倍体;(B)ROS影响动植物生殖过程的机制:植物精子通过花粉管伸长以完成受精,但受精后精细胞来源的DNA会持续降解,最终形成单倍体的胚;在人类中也发现精细胞ROS爆发会导致男性不育。
eFi-HI
BBM基因在卵细胞中异位表达,无需依赖受精即可诱导拟南芥、油菜及番茄的单倍体胚胎发生(Chen et al., 2022),另一个候选基因是来自蒲公英的PAR基因,其在莴苣(Underwood et al., 2022)和谷子(Huang et al., 2024)卵细胞中异位表达同样能够在未受精的情况下使单倍体胚胎发育即引发孤雌生殖。
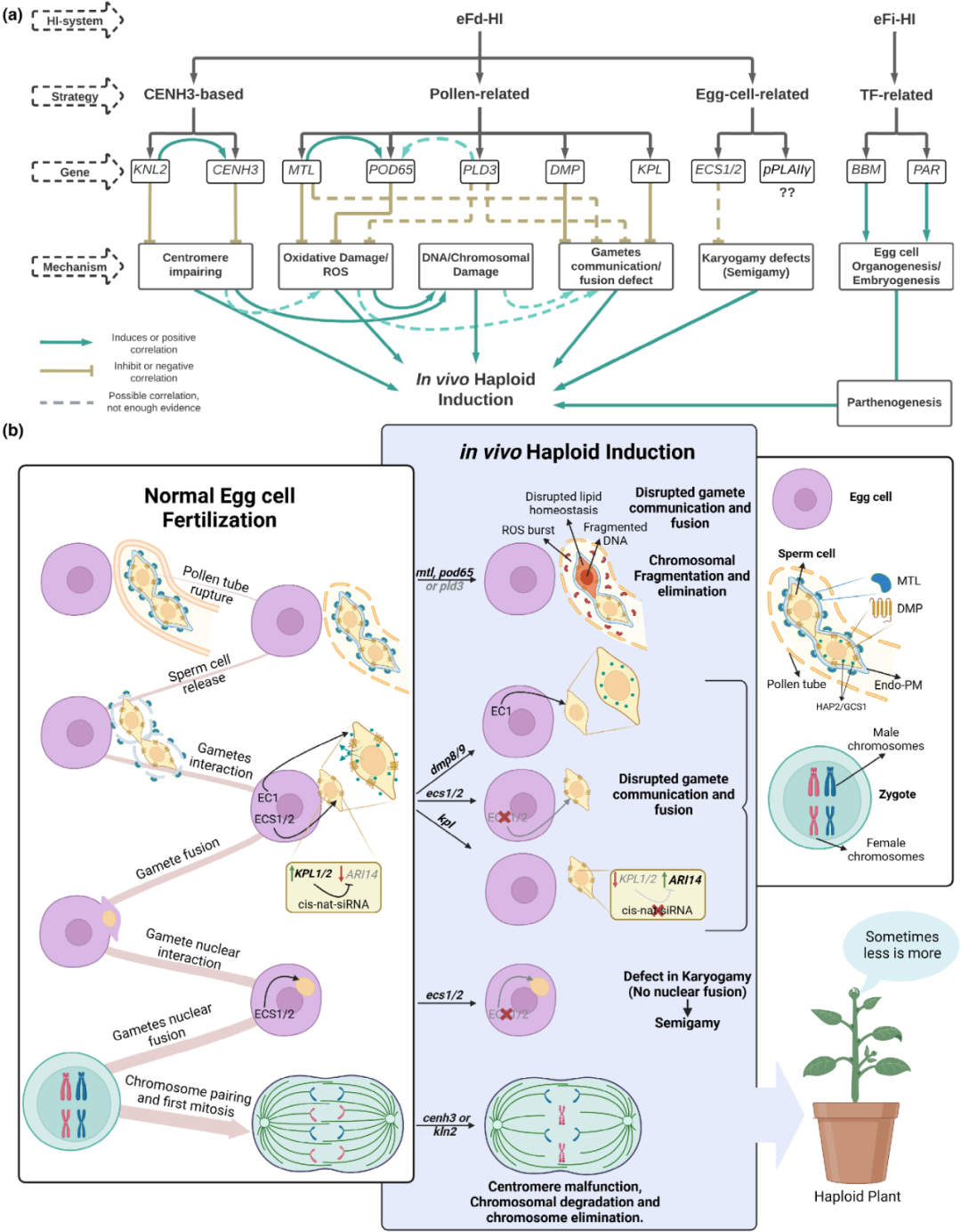
综上所述,体内单倍体诱导技术中的eFd-HI和eFi-HI的分子机制有所不同(图4)。eFd-HI依赖受精,因其中一个亲本产生缺陷型配子导致该亲本的基因组无法融入合子或以其他方式将某个亲本的基因组从合子的基因组中消除,最终产生单倍体后代。eFi-HI不依赖受精,通过在卵细胞中异位表达相关的转录因子,激活器官发生或胚胎发生途径,可直接诱导单倍体胚胎的发育。
图4 植物体内单倍体诱导机制(Quiroz et al., 2024)。(a)单倍体诱导分为eFd-HI和eFi-HI;(b)敲除参与卵细胞受精和早期受精卵发育的基因可诱导单倍体的发生。
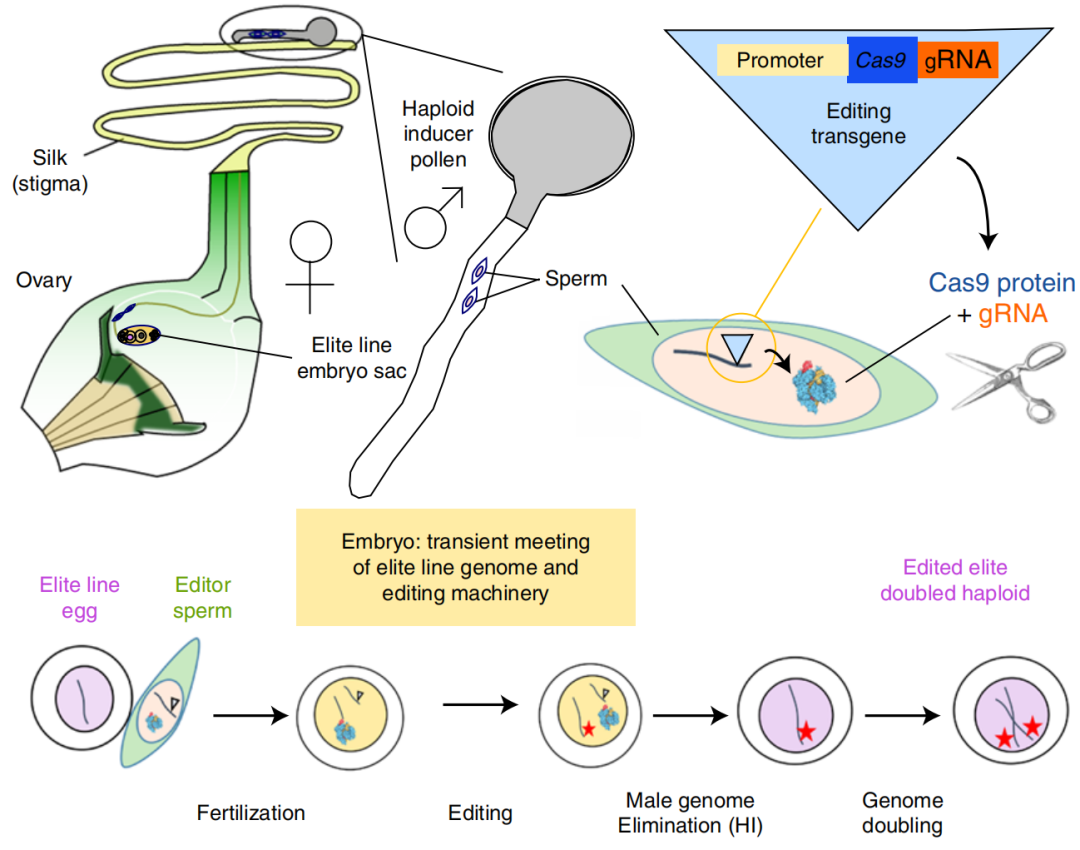
2019年3月,先正达公司Timothy Kelliher和Qiudeng Que团队在Nature biotechnology杂志上发表了一篇题为“One-step genome editing of elite crop germplasm during haploid induction”的研究论文,作者提出了HI-Edit策略,将单倍体诱导育种与基因编辑技术结合,可在短时间内对任意品种进行改良,使基因编辑技术的应用不再受遗传转化技术及政策的限制。
图5 HI-Edit创制单倍体诱导系(Kelliher et al., 2019)。
应用二,将利用CRISPR技术对玉米自交系NP2222的VLPH1基因进行编辑获得的纯合且含有Cas9+的植株,与自然单倍体诱导系RWKS(含有纯合mtl基因)杂交,从F2后代中筛选含有Cas9+且mtl基因纯合的个体与自交系GP650杂交,筛选出不含Cas9+且VLPH1基因被敲除的单倍体,加倍后可获得VLPH1纯合敲除植株,且植株背景为自交系GP650。此方法表明HI-Edit可用于对任意品种创制相应的基因编辑植株。备注:原文中试了多个基因,伯小远在这里只用了其中一个基因举例。
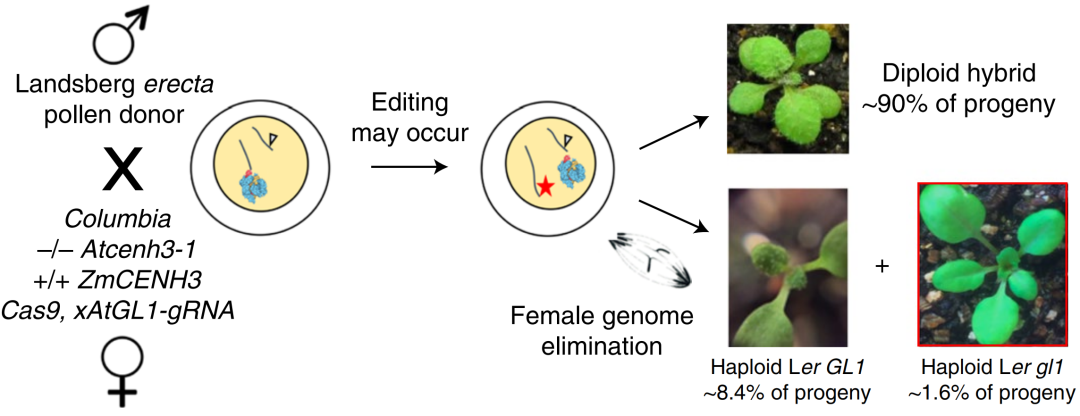
应用三,作者针对双子叶植株,开发了CENH3 HI-Edit系统。用ZmCENH3替换Col生态型拟南芥原有的AtCENH3,再向此基因型的植株中转入Cas9-AtGL1载体,之后与Ler生态型拟南芥杂交,后代中可筛选出AtGL1纯合敲除株系,且株系背景为Ler生态型。此方法表明CENH3 HI-Edit可用于对双子叶植物的任意品种创制相应的基因编辑植株。
图6 拟南芥CENH3 HI-Edit过程(Kelliher et al., 2019)。
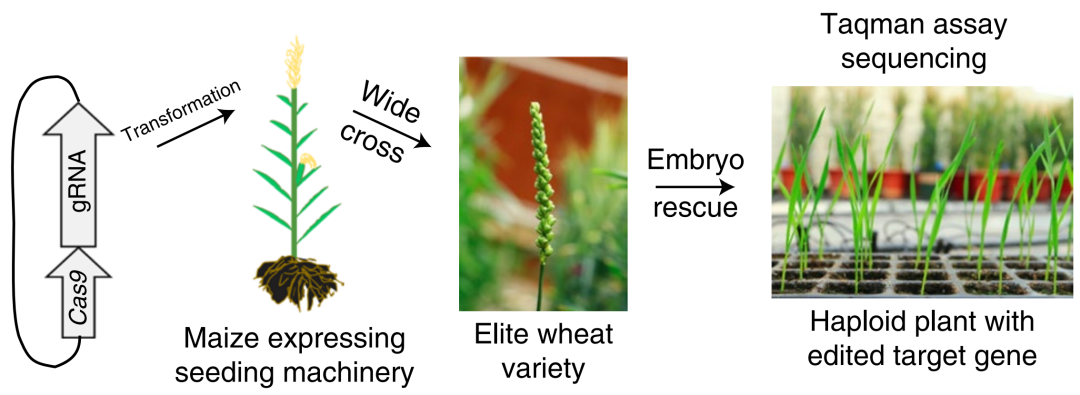
应用四,将携带Cas9-TaGT1载体的玉米花粉授粉至小麦细胞质雄性不育系(CMS)的柱头上,通过胚胎拯救可获得具有CMS背景且TaGL1被编辑的单倍体植株。此方法表明HI-Edit可用于创制属间杂交基因编辑植株。
图7 HI-Edit创制属间杂交基因编辑植株(Kelliher et al., 2019)。备注:远缘杂交诱导包括属间杂交和种间杂交,以小麦做母本与玉米杂交属于属间杂交,在其合子发育过程中由于玉米染色体与小麦染色体有丝分裂进程不同步,玉米染色体被消除,因而可诱导出小麦单倍体幼胚。
IMGE
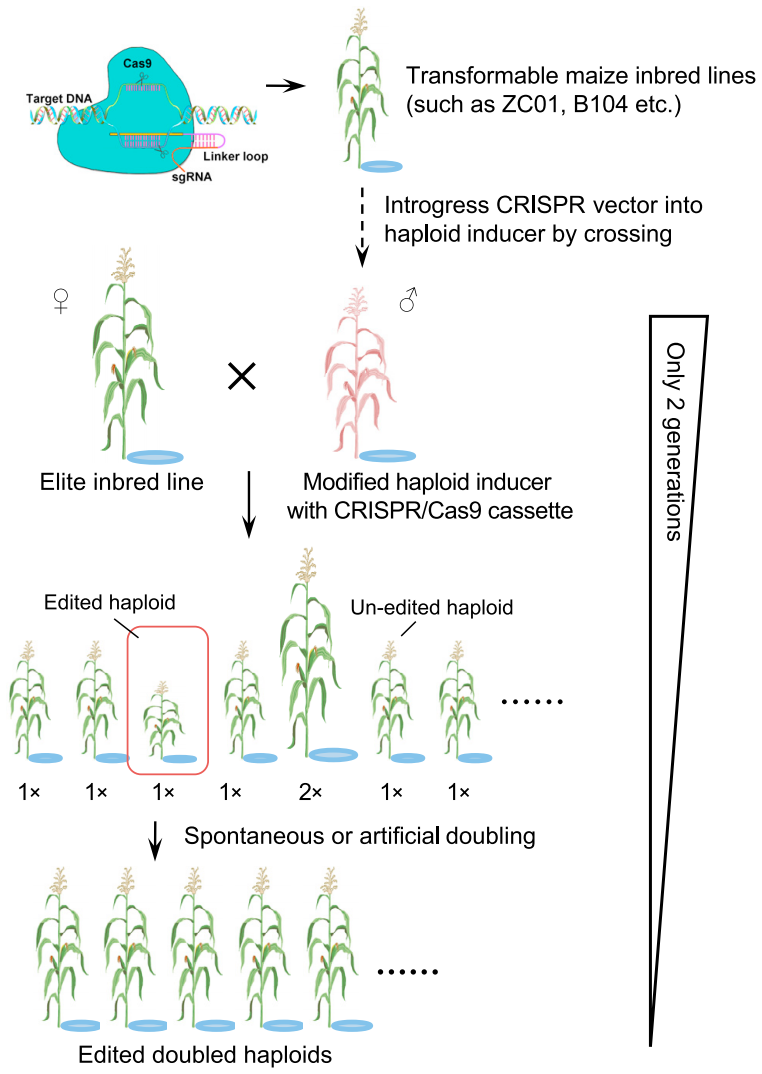
2019年4月,中国农业科学院生物技术研究所王宝宝团队和华南农业大学王海洋团队在Molecular Plant杂志上发表了一篇题为“Development of a Haploid-Inducer Mediated Genome Editing System for Accelerating Maize Breeding”的研究论文,作者提出了一种基于单倍体诱导介导的基因组编辑技术(Haploid-Inducer Mediated Genome Editing,IMGE),巧妙地将单倍体诱导与CRISPR基因编辑技术结合起来,成功地在两代内创造出经基因编辑改良的DH纯系。该方法打破了基因编辑育种对遗传转化体系的依赖,并可快速创制出不含CRISPR载体的DH系,在学术界被誉为作物育种的“马良神笔”。
IMGE的技术路线是将改良某个农艺性状的CRISPR载体转化至有稳定转化体系的玉米品种如ZC01或B104中,然后与单倍体诱导系(含有纯合mtl基因)杂交,选择含有Cas9+载体且含有纯合mtl基因的后代,以此新创制的单倍体诱导系为父本,与任意自交系如B73杂交,选择后代中目标农艺性状变化的单倍体,通过加倍可获得具有改良农艺性状的纯合基因编辑DH系,且其背景为B73。读到这里,大家会发现,这个技术路线与上述HI-Edit方法的应用二其实是一样的,这两个独立的研究为该技术路线的可靠性提供了进一步的佐证。
图8 IMGE技术路线图(Wang et al., 2019)。
无融合生殖
2019年1月,中国农科院王克剑团队和Raphaël Mercier团队在Nature Biotechnology杂志上发表了题为“Clonal seeds from hybrid rice by simultaneous genome engineering of meiosis and fertilization genes”的文章,该研究将杂交水稻中三个减数分裂关键基因REC8 、PAIR1 及OSD1 和单倍体诱导基因MTL共四个内源基因进行了基因编辑,得到了Fix 基因型植株,自交后其后代中存在二倍体子代,实现了杂种一代杂种优势的固定,无需额外的杂交就可以直接获得克隆种子(Wang et al., 2019b)。或者,也可以敲除REC8 、PAIR1 和OSD1 三个基因并异位表达BBM1 或BBM4 基因得到克隆种子(Khanday et al., 2019; Vernet et al., 2022; Wei et al., 2023)。关于无融合生殖的详细内容可参考文章“杂交作物如何通过自交获得克隆种子?”、“袁隆平爷爷心系的‘一系法’,能否实现?”。
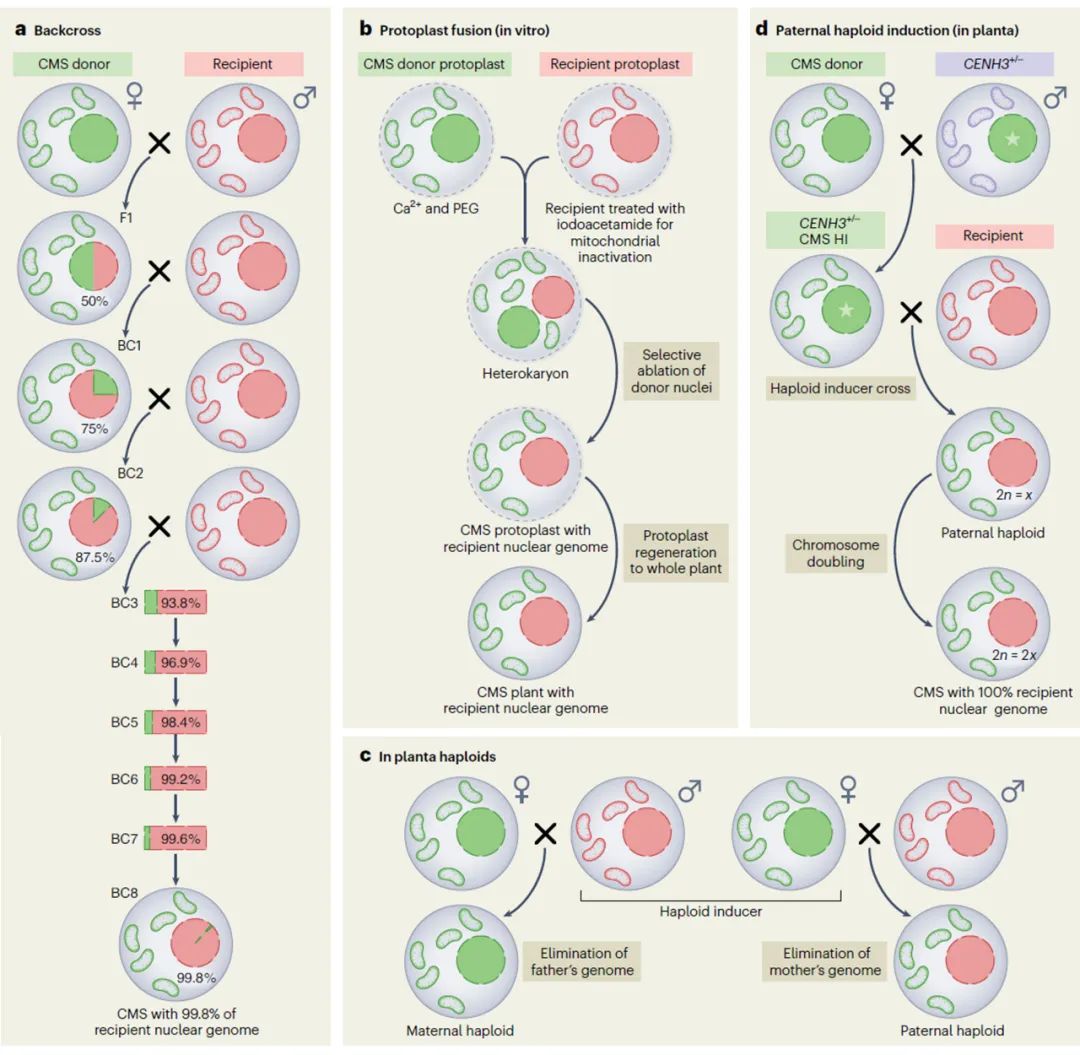
细胞质替换/创制细胞质雄性不育系
利用细胞质雄性不育系(CMS)生产杂交种子在实际生产中应用较多,然而,在传统方法中,将CMS从供体导入商业化亲本需要多次回交(图9a),这一过程既费时又耗力。如今,随着单倍体诱导技术的发展,CMS的快速转移成为可能(图9d)。

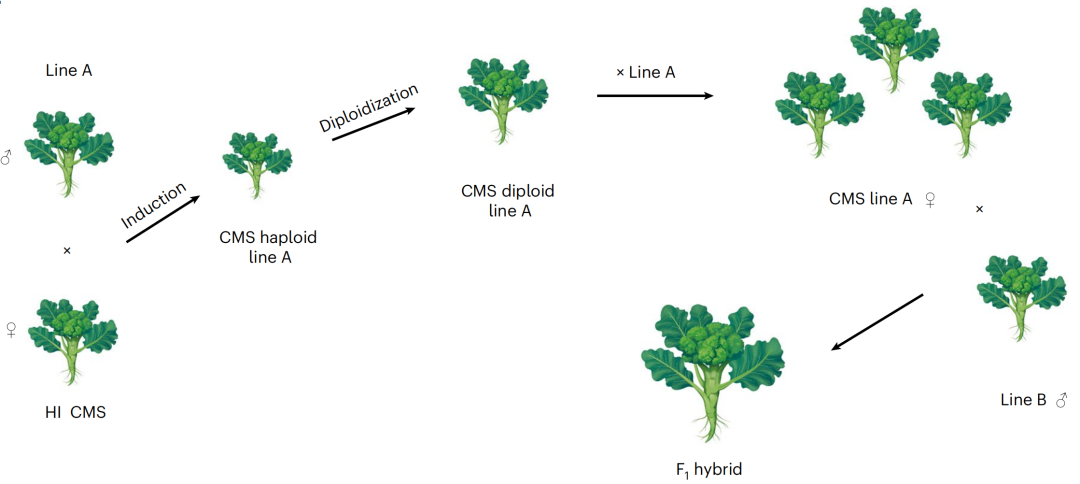
作者利用CRISPR技术创制了以青花菜自交系C33为背景的基因敲除材料,发现T1代中得到的两种纯合突变体材料BoCENH3Δ3和BoCENH3RFS33只具有父系单倍体诱导能力。以BoCENH3RFS33作为父本,与CMS品种CMS219(该品种含有Ogura CMS型细胞质)杂交、回交,在BC1F1代中得到含有Ogura CMS细胞质及核基因为BoCENH3RFS33的纯合个体,创制了Ogura CMS细胞质的父系单倍体诱导系,随后将其命名为HI-CMS系(即图9中提到的CMS-单倍体诱导系)。再利用HI-CMS系作为母本与青花菜自交系B54杂交,在F1代中可得到细胞质基因型为Ogura CMS、细胞核基因型为B54的单倍体,将该单倍体命名为CMS B54。以秋水仙素加倍处理后的倍性恢复材料作为母本,以B54作为恢复系父本,发现其后代可以正常结实,且保持基因型为CMS B54。此时即可利用已经完成创制的雄性不育系,将其作为母本进行杂交制种。利用此方法,可创制任意青花菜自交系的雄性不育系。
图12 青花菜中基于CMS的杂种优势利用模型(Han et al.,2024)。
小远叨叨
单倍体育种已成为继分子标记辅助育种、转基因育种之外的核心育种技术。在本篇文章中,伯小远首先带大家回顾了单倍体诱导有关的技术,着重回顾了其中体内单倍体诱导技术有关的基因,其中属于卵细胞受精依赖性单倍体诱导的相关基因有MTL/PLA1/NLD、DMP、PLD3、KPL、ECS、pPLAIIγ、CENH3、LIG4和ZmPOD65等,属于卵细胞受精非依赖性单倍体诱导的相关基因有BBM和PAR等。接着,伯小远重点介绍了几种由单倍体诱导衍生出的新技术,这些新技术大多基于MTL突变基因创制的母系单倍体诱导技术和CENH3突变基因创制的父系单倍体诱导技术,包括HI-Edit、IMGE、无融合生殖和细胞质替换/创制细胞质雄性不育系等,它们为未来的育种带来了巨大的想象空间,十分振奋人心!由单倍体诱导衍生出的更多技术,欢迎大家继续补充喔!
References:
Ahmadli U, Kalidass M, Khaitova LC, Fuchs J, Cuacos M, Demidov D, Zuo S, Pecinkova J, Mascher M, Ingouff M, Heckmann S, Houben A, Riha K, Lermontova I (2023) High temperature increases centromere-mediated genome elimination frequency and enhances haploid induction in Arabidopsis. PLANT COMMUN 4: 100507
Bortiri E, Selby R, Egger R, Tolhurst L, Dong S, Beam K, Meier K, Fabish J, Delaney D, Dunn M, Mcnamara D, Setliff K, Castro MLR, Gergen S, Dawe RK, Kelliher T (2024) Cyto-swapping in maize by haploid induction with a cenh3 mutant. NAT PLANTS 10: 567-571
Chen B, Maas L, Figueiredo D, Zhong Y, Reis R, Li M, Horstman A, Riksen T, Weemen M, Liu H, Siemons C, Chen S, Angenent GC, Boutilier K (2022) BABY BOOM regulates early embryo and endosperm development. P NATL ACAD SCI USA 119: e2093206177
Chen X, Li Y, Ai G, Chen J, Guo D, Zhu Z, Zhu X, Tian S, Wang J, Liu M, Yuan L (2023) Creation of a watermelon haploid inducer line via ClDMP3-mediated single fertilization of the central cell. HORTIC RES-ENGLAND 10: d81
Cheng Z, Sun Y, Yang S, Zhi H, Yin T, Ma X, Zhang H, Diao X, Guo Y, Li X, Wu C, Sui Y (2021) Establishing in planta haploid inducer line by edited SiMTL in foxtail millet (Setaria italica). PLANT BIOTECHNOL J 19: 1089-1091
Gilles LM, Khaled A, Laffaire JB, Chaignon S, Gendrot G, Laplaige J, Berges H, Beydon G, Bayle V, Barret P, Comadran J, Martinant JP, Rogowsky PM, Widiez T (2017) Loss of pollen-specific phospholipase NOT LIKE DAD triggers gynogenesis in maize. EMBO J 36: 707-717
Han F, Zhang X, Liu Y, Liu Y, Zhao H, Li Z (2024) One-step creation of CMS lines using a BoCENH3-based haploid induction system in Brassica crop. NAT PLANTS 10: 581-586
Huang Y, Liang Y, Xie Y, Rao Y, Xiong J, Liu C, Wang C, Wang X, Qian Q, Wang K (2024) Efficient haploid induction via egg cell expression of dandelion PARTHENOGENESIS in foxtail millet (Setaria italica). PLANT BIOTECHNOL J
Han F, Zhang X, Liu Y, Liu Y, Zhao H, Li Z (2024) One-step creation of CMS lines using a BoCENH3-based haploid induction system in Brassica crop. NAT PLANTS 10: 581–586
Jacquier N, Calhau A, Fierlej Y, Martinant JP, Rogowsky PM, Gilles LM, Widiez T (2023) In planta haploid induction by kokopelli mutants. PLANT PHYSIOL 193: 182-185
Jang JH, Seo HS, Widiez T, Lee OR (2023) Loss-of-function of gynoecium-expressed phospholipase pPLAIIgamma triggers maternal haploid induction in Arabidopsis. NEW PHYTOL 238: 1813-1824
Jiang C, Sun J, Li R, Yan S, Chen W, Guo L, Qin G, Wang P, Luo C, Huang W, Zhang Q, Fernie AR, Jackson D, Li X, Yan J (2022) A reactive oxygen species burst causes haploid induction in maize. MOL PLANT 15: 943-955
Kelliher T, Starr D, Richbourg L, Chintamanani S, Delzer B, Nuccio ML, Green J, Chen Z, McCuiston J, Wang W, Liebler T, Bullock P, Martin B (2017) MATRILINEAL, a sperm-specific phospholipase, triggers maize haploid induction. NATURE 542: 105-109
Kelliher T, Starr D, Su X, Tang G, Chen Z, Carter J, Wittich PE, Dong S, Green J, Burch E, McCuiston J, Gu W, Sun Y, Strebe T, Roberts J, Bate NJ, Que Q (2019) One-step genome editing of elite crop germplasm during haploid induction. NAT BIOTECHNOL 37: 287-292
Kelliher T, Starr D, Wang W, McCuiston J, Zhong H, Nuccio ML, Martin B (2016) Maternal Haploids Are Preferentially Induced by CENH3-tailswap Transgenic Complementation in Maize. FRONT PLANT SCI 7: 414
Khanday I, Skinner D, Yang B, Mercier R, Sundaresan V (2019) A male-expressed rice embryogenic trigger redirected for asexual propagation through seeds. NATURE 565: 91-95
Li Y, Lin Z, Yue Y, Zhao H, Fei X, E L, Liu C, Chen S, Lai J, Song W (2021) Loss-of-function alleles of ZmPLD3 cause haploid induction in maize. NAT PLANTS 7: 1579-1588
Liu C, Li X, Meng D, Zhong Y, Chen C, Dong X, Xu X, Chen B, Li W, Li L, Tian X, Zhao H, Song W, Luo H, Zhang Q, Lai J, Jin W, Yan J, Chen S (2017) A 4-bp Insertion at ZmPLA1 Encoding a Putative Phospholipase A Generates Haploid Induction in Maize. MOL PLANT 10: 520-522
Liu C, Zhong Y, Qi X, Chen M, Liu Z, Chen C, Tian X, Li J, Jiao Y, Wang D, Wang Y, Li M, Xin M, Liu W, Jin W, Chen S (2020a) Extension of the in vivo haploid induction system from diploid maize to hexaploid wheat. PLANT BIOTECHNOL J 18: 316-318
Liu H, Wang K, Jia Z, Gong Q, Lin Z, Du L, Pei X, Ye X (2020b) Efficient induction of haploid plants in wheat by editing of TaMTL using an optimized Agrobacterium-mediated CRISPR system. J EXP BOT 71: 1337-1349
Lv J, Yu K, Wei J, Gui H, Liu C, Liang D, Wang Y, Zhou H, Carlin R, Rich R, Lu T, Que Q, Wang WC, Zhang X, Kelliher T (2020) Generation of paternal haploids in wheat by genome editing of the centromeric histone CENH3. NAT BIOTECHNOL 38: 1397-1401
Mao Y, Nakel T, Erbasol SI, Joshi S, Tekleyohans DG, Baum T, Gross-Hardt R (2023) ECS1 and ECS2 suppress polyspermy and the formation of haploid plants by promoting double fertilization. ELIFE 12
Marimuthu M, Maruthachalam R, Bondada R, Kuppu S, Tan EH, Britt A, Chan S, Comai L (2021) Epigenetically mismatched parental centromeres trigger genome elimination in hybrids. SCI ADV 7: k1151
Maruthachalam R (2024) Haploids fast-track hybrid plant breeding. NAT PLANTS 10: 530-532
Quiroz LF, Gondalia N, Brychkova G, McKeown PC, Spillane C (2024) Haploid rhapsody: the molecular and cellular orchestra of in vivo haploid induction in plants. NEW PHYTOL 241: 1936-1949
Ravi M, Chan SW (2010) Haploid plants produced by centromere-mediated genome elimination. NATURE 464: 615-618
Ravi M, Marimuthu MP, Tan EH, Maheshwari S, Henry IM, Marin-Rodriguez B, Urtecho G, Tan J, Thornhill K, Zhu F, Panoli A, Sundaresan V, Britt AB, Comai L, Chan SW (2014) A haploid genetics toolbox for Arabidopsis thaliana. NAT COMMUN 5: 5334
Tan EH, Henry IM, Ravi M, Bradnam KR, Mandakova T, Marimuthu MP, Korf I, Lysak MA, Comai L, Chan SW (2015) Catastrophic chromosomal restructuring during genome elimination in plants. ELIFE 4
Tang H, Qiu Y, Wang W, Yu M, Chang Y, Han Z, Du L, Lin Z, Wang K, Ye X (2023) Development of a haploid inducer by editing HvMTL in barley. J GENET GENOMICS 50: 366-369
Tian S, Zhang J, Zhao H, Zong M, Li M, Gong G, Wang J, Zhang J, Ren Y, Zhang H, Li S, Wen C, Xu Y (2023) Production of double haploid watermelon via maternal haploid induction. PLANT BIOTECHNOL J 21: 1308-1310
Underwood CJ, Vijverberg K, Rigola D, Okamoto S, Oplaat C, Camp R, Radoeva T, Schauer SE, Fierens J, Jansen K, Mansveld S, Busscher M, Xiong W, Datema E, Nijbroek K, Blom EJ, Bicknell R, Catanach A, Erasmuson S, Winefield C, van Tunen AJ, Prins M, Schranz ME, van Dijk PJ (2022) A PARTHENOGENESIS allele from apomictic dandelion can induce egg cell division without fertilization in lettuce. NAT GENET 54: 84-93
Vernet A, Meynard D, Lian Q, Mieulet D, Gibert O, Bissah M, Rivallan R, Autran D, Leblanc O, Meunier AC, Frouin J, Taillebois J, Shankle K, Khanday I, Mercier R, Sundaresan V, Guiderdoni E (2022) High-frequency synthetic apomixis in hybrid rice. NAT COMMUN 13: 7963
Wang B, Zhu L, Zhao B, Zhao Y, Xie Y, Zheng Z, Li Y, Sun J, Wang H (2019) Development of a Haploid-Inducer Mediated Genome Editing System for Accelerating Maize Breeding. MOL PLANT 12: 597-602
Wang C, Liu Q, Shen Y, Hua Y, Wang J, Lin J, Wu M, Sun T, Cheng Z, Mercier R, Wang K (2019a) Clonal seeds from hybrid rice by simultaneous genome engineering of meiosis and fertilization genes. NAT BIOTECHNOL 37: 283-286
Wang C, Liu Q, Shen Y, Hua Y, Wang J, Lin J, Wu M, Sun T, Cheng Z, Mercier R, Wang K (2019b) Clonal seeds from hybrid rice by simultaneous genome engineering of meiosis and fertilization genes. NAT BIOTECHNOL 37: 283-286
Wang N, Xia X, Jiang T, Li L, Zhang P, Niu L, Cheng H, Wang K, Lin H (2022) In planta haploid induction by genome editing of DMP in the model legume Medicago truncatula. PLANT BIOTECHNOL J 20: 22-24
Wei X, Liu C, Chen X, Lu H, Wang J, Yang S, Wang K (2023) Synthetic apomixis with normal hybrid rice seed production. MOL PLANT 16: 489-492
Yu Zhong, Mingliang Yang, Dehe Cheng, Jinchu Liu, Qi Han, Chunyan Liu, Xiaolong Qi, Tongzheng Yan, Lei Teng, Chang Xv, Jingjing Hou, Lianjun Sun, Chenxu Liu, Qingshan Chen, Shaojiang Chen (2024) Mutation of GmDMP genes triggers haploid induction in soybean. bioRxiv. 585499
Zhang X, Shi C, Li S, Zhang B, Luo P, Peng X, Zhao P, Dresselhaus T, Sun MX (2023) A female in vivo haploid-induction system via mutagenesis of egg cell-specific peptidases. MOL PLANT 16: 471-480
Zhang X, Zhang L, Zhang J, Jia M, Cao L, Yu J, Zhao D (2022) Haploid induction in allotetraploid tobacco using DMPs mutation. PLANTA 255: 98
Zhong Y, Chen B, Li M, Wang D, Jiao Y, Qi X, Wang M, Liu Z, Chen C, Wang Y, Chen M, Li J, Xiao Z, Cheng D, Liu W, Boutilier K, Liu C, Chen S (2020) A DMP-triggered in vivo maternal haploid induction system in the dicotyledonous Arabidopsis. NAT PLANTS 6: 466-472
Zhong Y, Chen B, Wang D, Zhu X, Li M, Zhang J, Chen M, Wang M, Riksen T, Liu J, Qi X, Wang Y, Cheng D, Liu Z, Li J, Chen C, Jiao Y, Liu W, Huang S, Liu C, Boutilier K, Chen S (2022) In vivo maternal haploid induction in tomato. PLANT BIOTECHNOL J 20: 250-252
Zhong Y, Liu C, Qi X, Jiao Y, Wang D, Wang Y, Liu Z, Chen C, Chen B, Tian X, Li J, Chen M, Dong X, Xu X, Li L, Li W, Liu W, Jin W, Lai J, Chen S (2019) Mutation of ZmDMP enhances haploid induction in maize. NAT PLANTS 5: 575-580