基因表达的调节依赖于从DNA到RNA的蛋白质和核酸的复杂相互作用,基因调控的关键机制之一是順式调控元件(CRE)对转录的控制,CRE是基因近端基因组区域内的短DNA序列基序,可被转录因子(TF)识别。在转录水平上,RNA生物过程也起到了关键作用。这些生物学调控过程是相互依存的,也被称为基因调控网络(GRN)。目前,分子生物学实验技术只能揭示其调控过程中的部分,由此,植物基因调控密码的探索还有待于通过深度学习的方法进行深入整体的研究。
近日,来自德国莱布尼茨植物遗传与作物研究所、生物与地球科学研究所、德国杜塞尔大学的Jędrzej Szymański团队,在《Nature Communications》杂志发表了题为“Deep learning the cis-regulatory code for gene expression in selected model plants”的文章。该研究通过深度学习模型对拟南芥、番茄、玉米等植物基因的调控元件和基因表达谱进行预测,有效识别了物种特异性调控序列特征及其对基因表达变化之间的关系,通过该模型也可以预测关键功能基因群的基因型特异性表达。
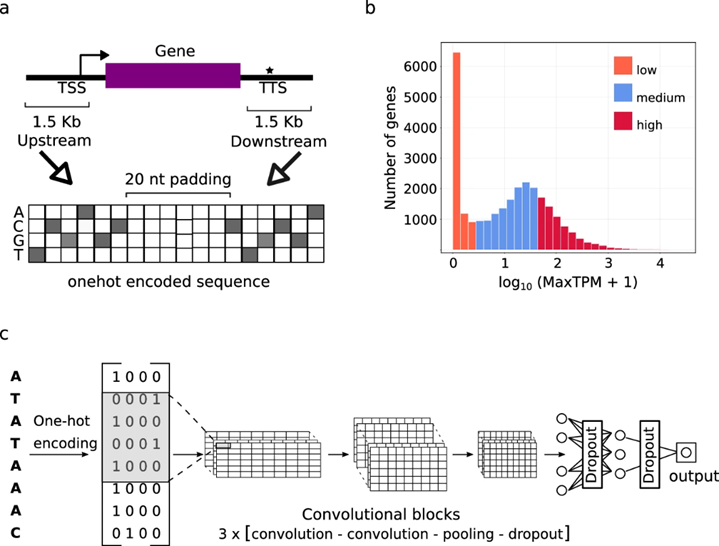
图1 基因表达预测模型的数据来源和模型架构。
首先,作者生成了一个数据资源,作为训练模型的数据来源。选取拟南芥、番茄、高粱、玉米的基因组组装并注释,提取转录起始位点(TSS)上游500-3000nt和下游100-700nt,转录终止位点(TTS)上游100-700nt和下游500-3000nt。研究的方法建立在CNN模型架构的基础上。(图1)
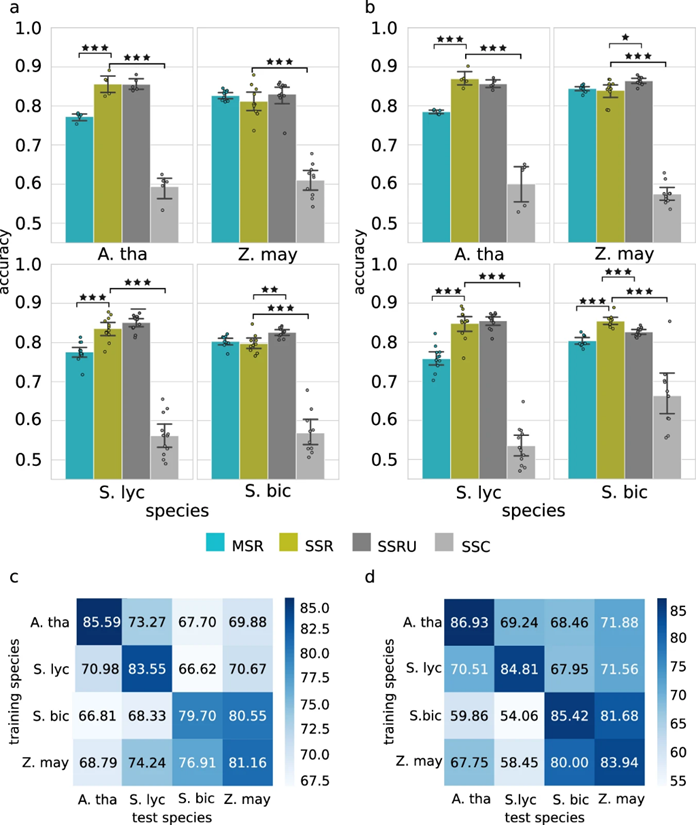
图2 不同训练数据组合下深度学习CNN基因表达预测模型对农作物的预测性能比较。
作者针对不同训练数据组合下深度学习CNN基因表达预测模型对农作物的预测性能进行比较。通过计算预测叶片模型的性能,以不同的作物为研究对象,基于训练数据的不同组合和变异,生成了四种不同的基因表达预测模型,同时也通过计算来预测估计根系模型的性能(图2a、b)。作者通过在三个物种上进行训练模型,并在第四个物种中评估,发现预测序列特征是保守的(图2a、2b)。同时,作者也测试了SSR模型在其他物种上的预测准确性,即跨物种预测。例如,利用玉米的序列数据进行预测,高粱SSR模型的根和叶的跨物种预测准确率最高,分别为80.55%和81.68%(图2c、2d)。跨物种预测的精度分析表明,该模型的性能可能取决于物种的进化关系,亲缘关系越近的物种的交叉性能越高。与SSR和MSR模型相比,跨物种预测性能显著下降,也表明了每个物种的基因表达由两类序列特征决定:物种特异性和跨物种保守性。
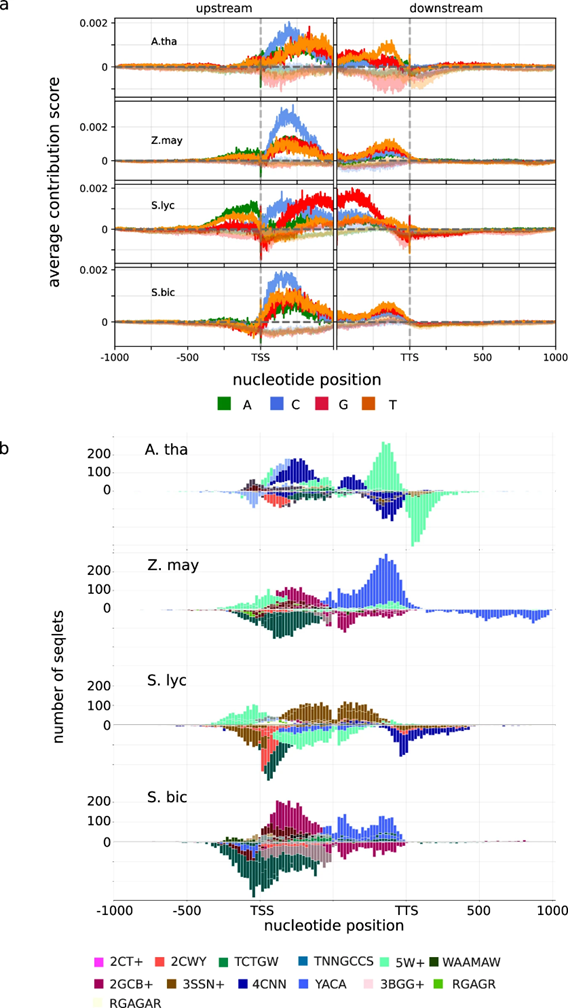
图3 预测序列特征的识别与表征。
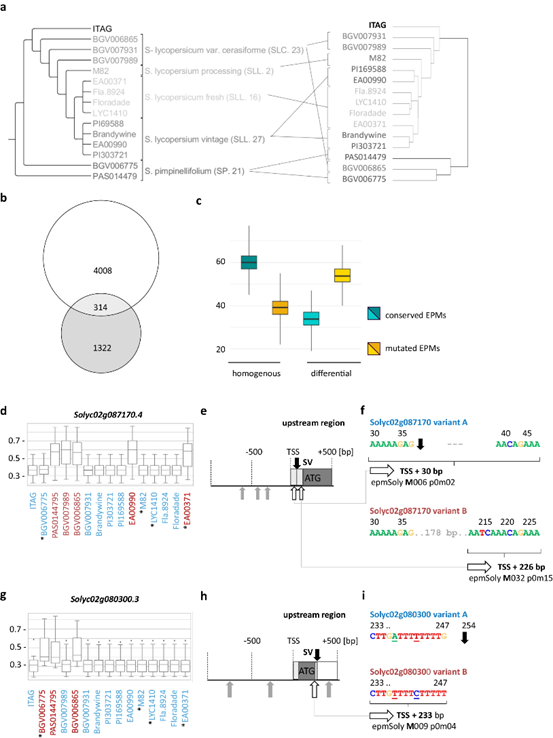
图4 15种不同茄属基因型的表达预测基序(EPMs)与转录起始位点(TSS)和转录终止位点(TTS)结构变异的可预测性比较。
作者对15种茄属基因型的表达预测基序(EPMs)与转录起始位点(TSS)和转录终止位点(TTS)结构变异的可预测性进行比较,结果表明,EPM在不同基因型中的发生可以进一步解释基因表达水平预测的差异,且CNN的预测依赖于EPM在其首选范围内的识别,并且这些权重是不同的(图4)。同时,作者也检索了可用于预测基因表达率和表征启动子或终止子区域的EPM。
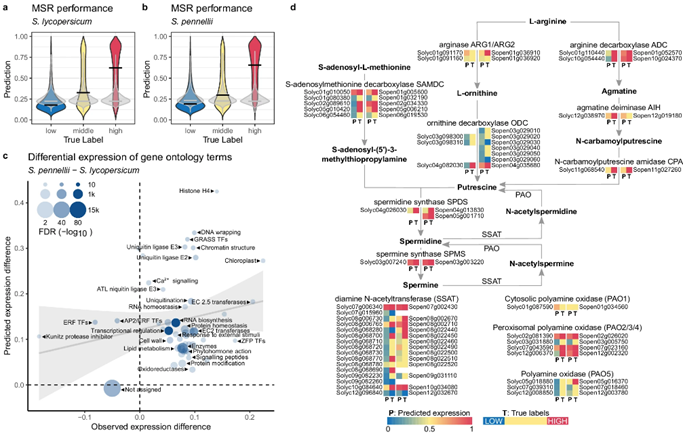
图5 利用MSR模型预测S. lycopersicum和 S. pennellii基因的表达。
作者也进一步评估了MSR模型是否突出了未用于训练的植物物种之间的可解释性差异,结果表明MSR模型正确地表明了多胺生物合成途径在S. lycopersicum和 S. pennellii物种中的高表达,高精度地鉴定了每个生物反应中最可能高表达的同工酶,并强调了S. lycopersicum 和S. pennellii在酶及其同系物表达方面的一些差异(图5)。
该研究作者通过深度学习模型对不同物种进行了探索和预测,验证了例如UTR区域在决定基因表达水平中的重要作用,显示了显著的跨物种性能,并且可以有效的预测关键功能基因组和基因型特异性表达,为植物基因调控的探索提供了新的学习预测方法和重要的理论基础。
原文链接:
https://www.nature.com/articles/s41467-024-47744-0
文章来源:植物生物技术Pb