近日,中国农业大学玉米生物育种全国重点实验室、国家玉米改良中心宋伟彬团队在Nature Genetics杂志上发表题为“Genomic variation in weedy and cultivated broomcorn millet accessions uncovers the genetic architecture of agronomic traits”的研究成果。
玉米,作为我国的第一大粮食作物,对于保障国家粮食安全具有至关重要的意义。长期的人工选择造成了优良等位基因的缺失,导致种质基础趋于狭窄,严重限制了玉米育种创新。因此,开展玉米近缘物种组学研究,通过比较基因组学技术手段挖掘玉米近缘种特有、特优基因资源并应用于玉米育种,对玉米种质基础的拓展具有重要的理论和实践指导意义。玉米近缘种黍稷起源于中国而且是最为古老的农作物之一,具有生育期短、耐旱耐瘠薄、适应盐碱胁迫等特点,暗示着黍稷基因组中蕴藏着丰富的优异基因资源。宋伟彬前期与合作者组装了黍稷(陇糜4号)基因组,共注释出63,671个编码基因,进一步与玉米自交系Mo17(39,756个编码基因)的比较基因组学分析发现,63,671个编码基因中11521个是黍稷特有(Nature Communications, 2019);为了从11,521个黍稷特有基因中发掘出与农艺性状和育种选择相关的基因,团队开展了育种过程中黍稷基因组变异图谱构建工作。
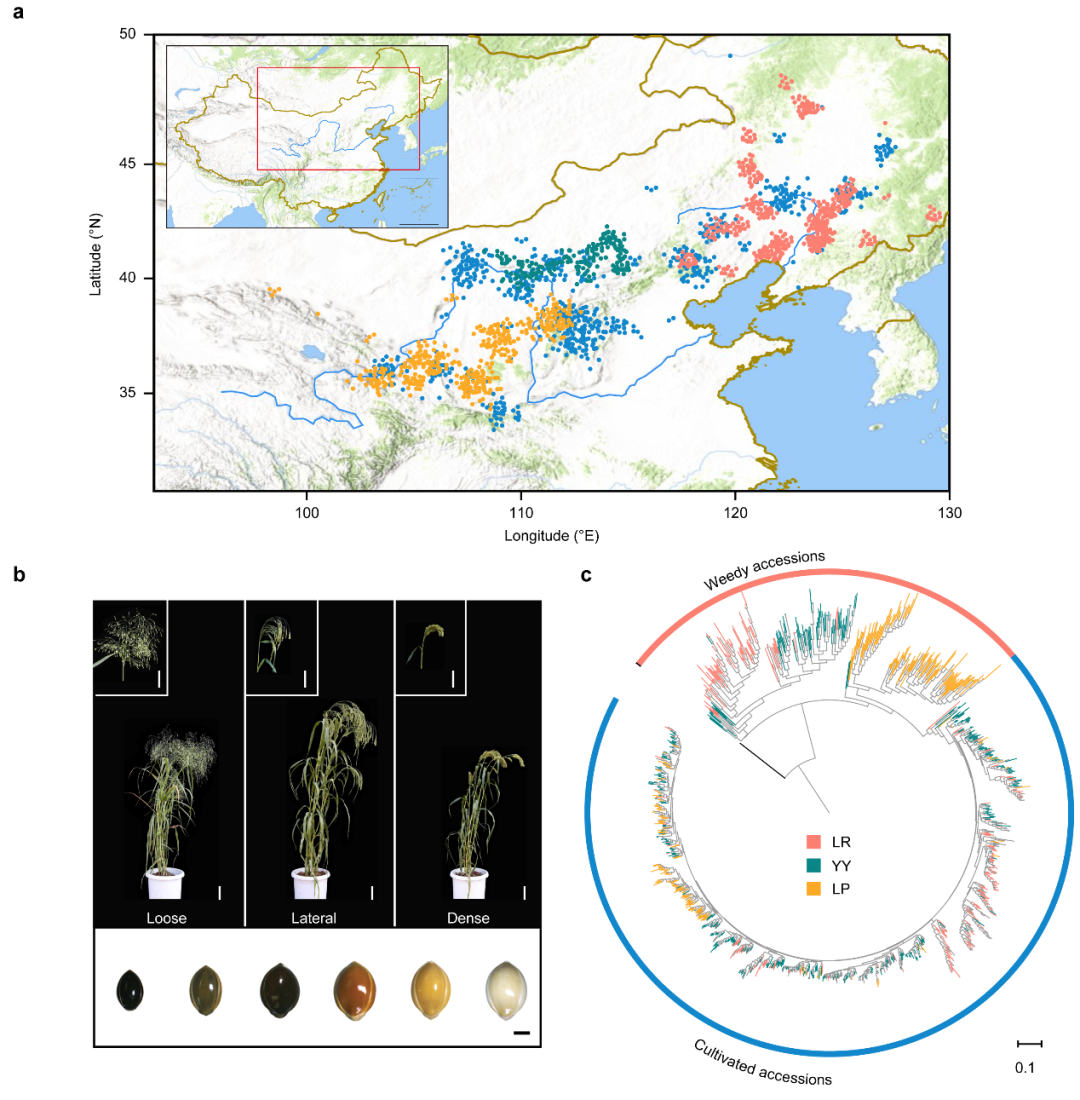
该研究团队系统地完成了中国野生黍稷种质资源的收集和基因资源挖掘工作。团队从内蒙古、陕西、甘肃、辽宁、吉林、河北、山西、宁夏、黑龙江的等10个省份的路边、丘陵、山地、农田等地收集了967份野生黍稷,结合937份栽培黍稷组成了一个1,904份的黍稷大群体。利用双末端150 bp测序获得黍稷群体的全基因组重测序数据,产生了65.1 Tb的数据,平均基因组覆盖度为40.3倍。根据基因组覆盖度和IBS,对野生黍稷和栽培黍稷进行筛选,过滤低覆盖度和遗传背景相似的品种。最后保留了1,094份黍稷进行后续分析,包含316份野生黍稷和778份栽培黍稷(图1)。
图1 黍稷群体的地理分布及进化树
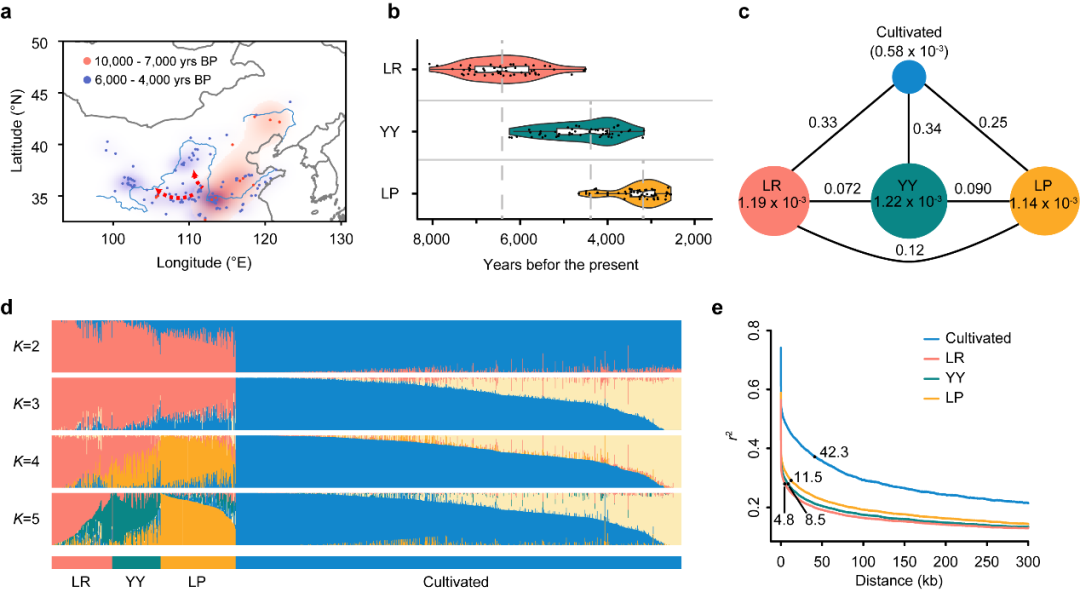
研究人员利用鉴定的5,603,840个SNPs解析了黍稷的群体结构,将黍稷群体分为四个亚群:LR(Liao River,辽河野生黍稷亚群)、YY(Yin Mountain and Yan Mountain,阴山-燕山野生黍稷亚群)、LP(Loess Plateau,黄土高原野生黍稷亚群)、Cultivated(栽培黍稷亚群),发现野生黍稷和栽培黍稷具有明显的遗传差异。进一步分析发现,黄土高原野生黍稷亚群与栽培黍稷分化时间最为接近(4,600年前),这与考古学的时间相吻合,也证实了现代栽培黍稷起源于黄土高原地区的单一起源理论,为黍稷的起源中心提供了理论支持。这些结果填补了中国黍稷遗传多样性研究的空白,并明确了现代栽培黍稷在中国的精确起源地里位置,有助于我们更加全面地了解中国黍稷的遗传特征、起源及其与栽培黍稷的关系(图2)。
图2 黍稷群体的群体结构分析
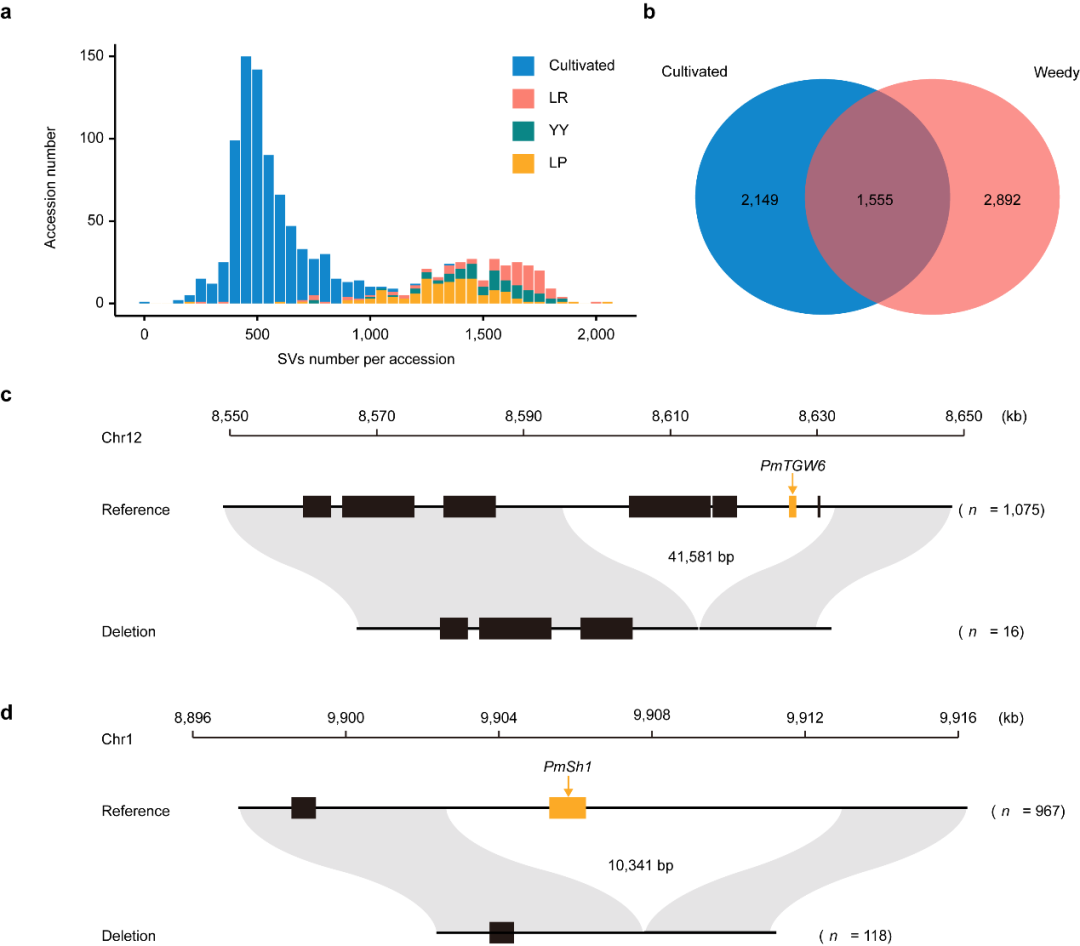
分析发现,在驯化过程中黍稷基因组不仅发生了单核苷酸变异和小片段的序列变异,也存在大片段的结构变异。每个野生黍稷材料平均含有1,431个SVs,每个栽培黍稷平均含有538个SVs,每个野生黍稷的SVs数量接近是栽培黍稷的3倍。为了分析SVs在驯化过程中的动态变化,该研究分析了SVs和TE的关系。发现SVs主要发生在Gypsy类型的TE上,另外Gypsy类型的SVs在黍稷驯化过程中受到选择,可能在驯化过程中发挥重要作用。为了探究SVs对基因的影响,分析了SVs和基因的位置关系,结果显示结构变异使基因组10%的基因为PAV。其中,栽培黍稷特有2,149个PAV基因,野生黍稷特有2,892个PAV基因,这些基因更多表现在影响植株的抗性和驯化性状。比如,栽培黍稷群体中的一个长度为41 kb的deletion引起影响籽粒大小基因PmTGW6的缺失、另外一个长度为10.3 kb的deletion引起影响落粒性基因PmSh1的缺失(图3)。
图3 野生黍稷和栽培黍稷的结构变异
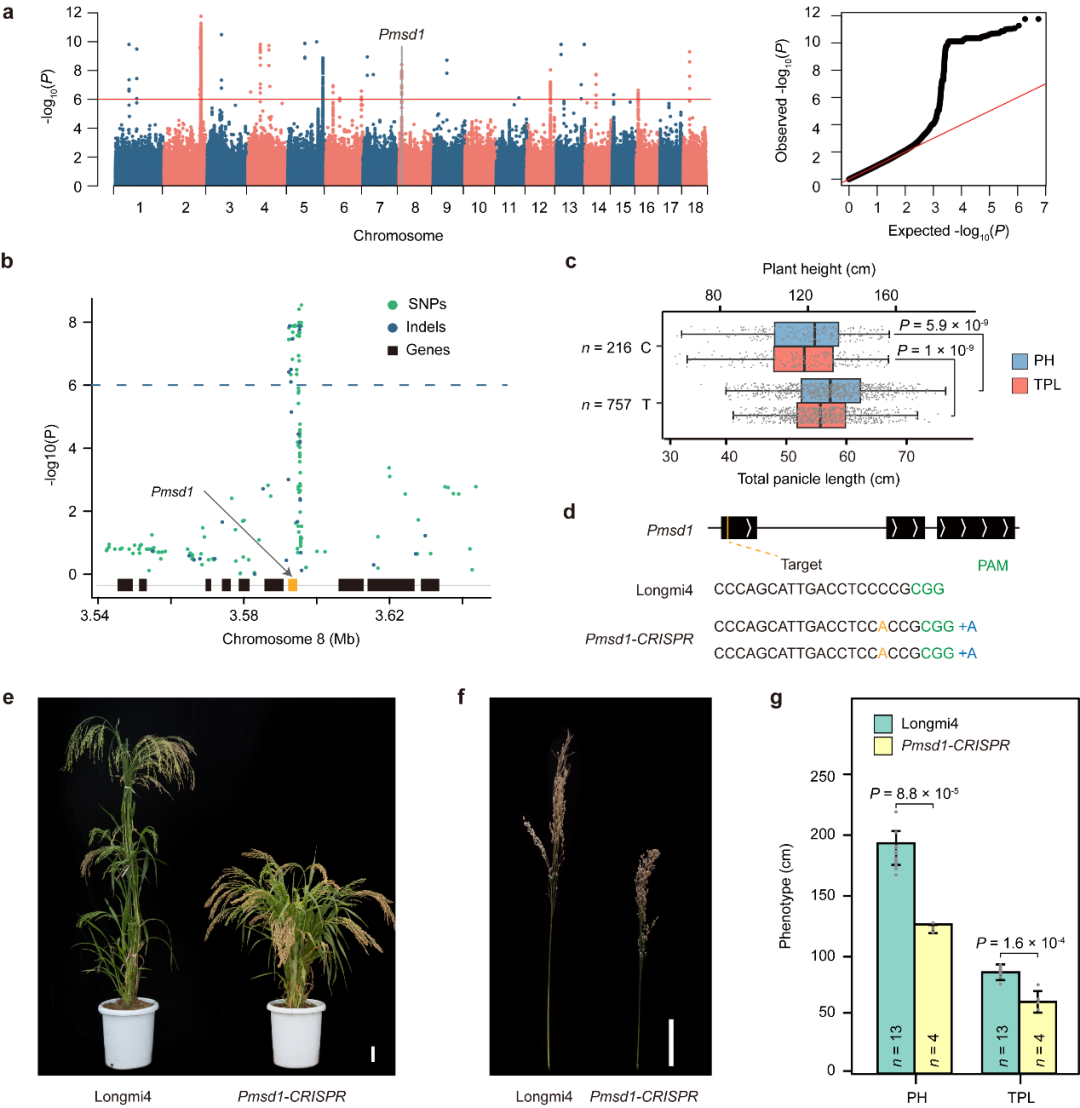
为了鉴定与产量相关的QTL位点,研究人员利用鉴定到的SNPs、Indels、SVs对12个农艺性状进行全基因组关联分析,并鉴定到186个QTL。其中与籽粒大小显著关联的PmGW8外显子上的T碱基缺失导致该基因发生移码突变,并且该基因在黍稷驯化过程中受到了强烈的选择;鉴定到两个与抽穗期显著关联的基因PmFT和PmPRR37,通过在拟南芥中过表达验证了PmFT能够正向调控花期,可以显著缩短开花时间;鉴定到了与株高显著关联的水稻sd1-1的同源基因Pmsd1基因,并且通过在黍稷中敲除Pmsd1,证实了Pmsd1参与了调控黍稷的株高和总穗长。综合分析发现,野生黍稷能鉴定更多籽粒相关的QTL,暗示着野生黍稷资源在黍稷籽粒性状的改良育种过程中可发挥重要作用(图4)。
图4 黍稷株高GWAS候选基因Pmsd1的鉴定与验证
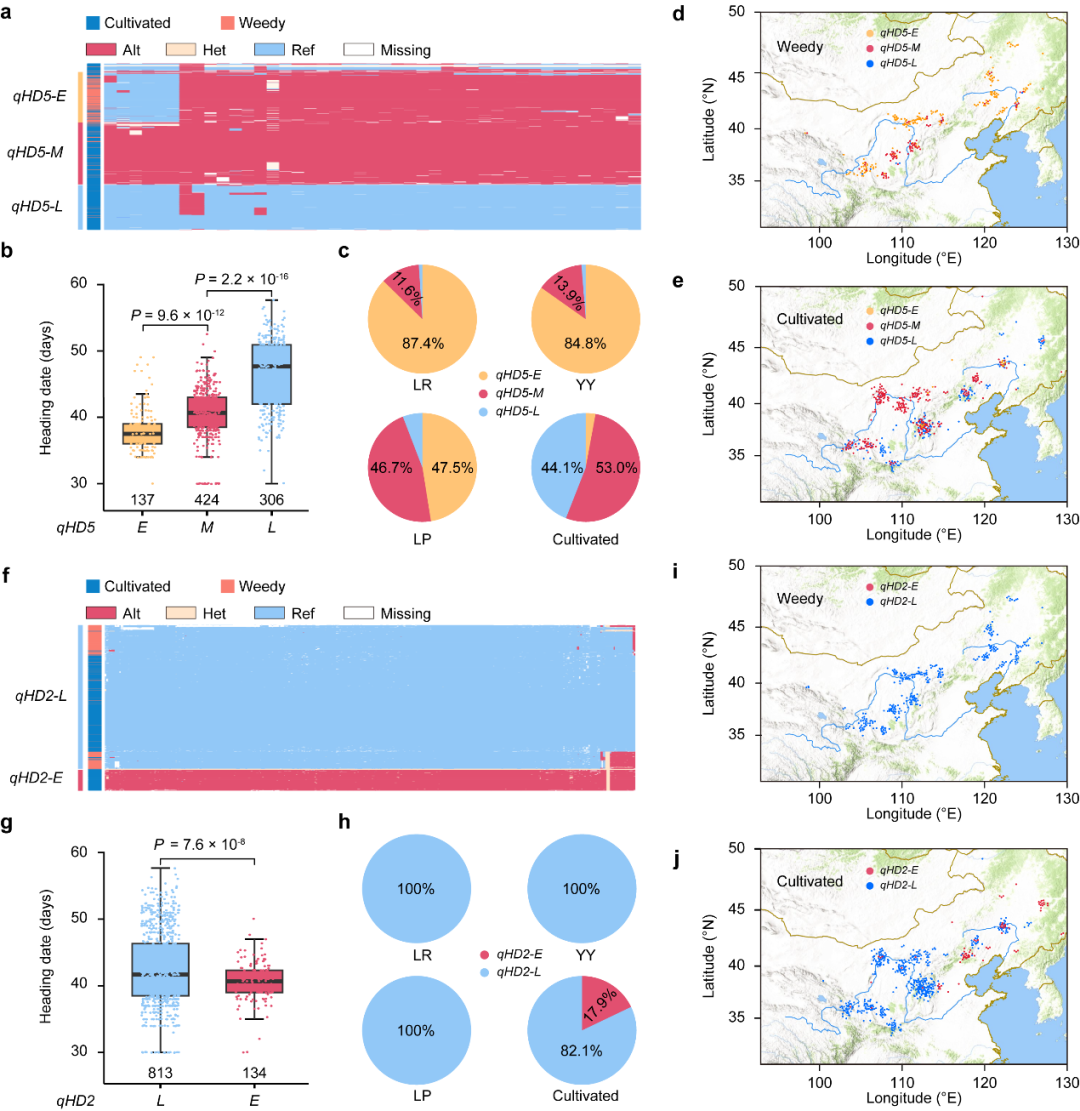
野生黍稷和栽培黍稷在我国北方地区具有广泛的分布,因而需要适应不同的生态和地理环境。开花期是植物适应当地环境的重要性状,解析花期的分子进化机制是了解黍稷适应性的关键。研究人员利用鉴定到的两个抽穗期QTL,qHD2和qHD5来探究野生黍稷和栽培黍稷的适应机制。结果表明野生黍稷通过选择qHD5的不同单倍型调节生长周期来适应不同纬度的环境,栽培黍稷通过选择qHD2的早花单倍型来适应高纬度的光周期条件(图5)。
图5 野生黍稷和栽培黍稷的平行适应
中国农业大学玉米生物育种全国重点实验室、国家玉米改良中心、农学院宋伟彬教授为该论文通讯作者。中国农业大学博士生陆琼、中国农业大学农学院、分子设计育种前沿科学中心赵海楠研究员、博士生张政权、博士生白雨禾、中国农业大学赵海铭副教授、河北省农科院刘国庆研究员为该论文共同第一作者。