本文内容预览:
真核生物中多肽和蛋白质分子经过核糖体合成后大多需要经过翻译后修饰才能正常的行使相应的生物学功能,蛋白质糖基化是真核生物常见的蛋白质翻译后修饰方式。蛋白质糖基化是在糖基转移酶的作用下,通过糖苷键将蛋白质分子与糖链分子进行连接,形成糖蛋白的过程。蛋白质糖基化修饰不仅参与细胞间相互作用、细胞外基质组织的形成以及细胞信号传导等重要的细胞过程,还可以改变蛋白质的结构、蛋白质的定位和蛋白质的运输方式,影响蛋白质的稳定性、生物学活性和功能,在生物体内发挥着重要的生理功能。
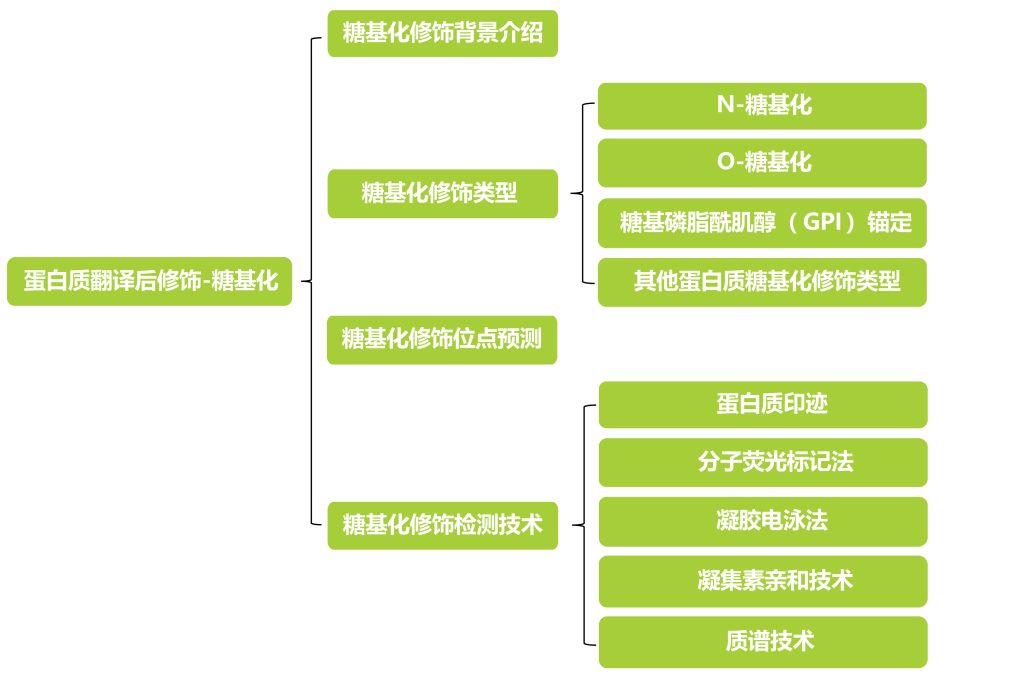
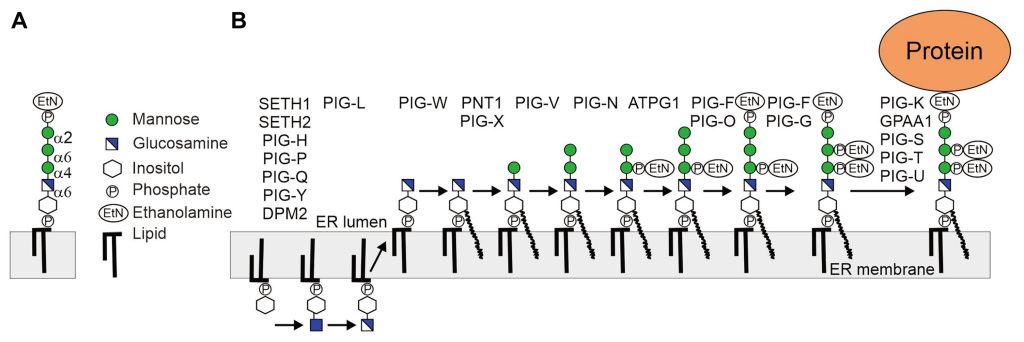
根据糖肽链的不同,蛋白质糖基化修饰主要包括N-糖基化、O-糖基化、糖基磷脂酰肌醇(GPI)锚定以及其他修饰类型等。
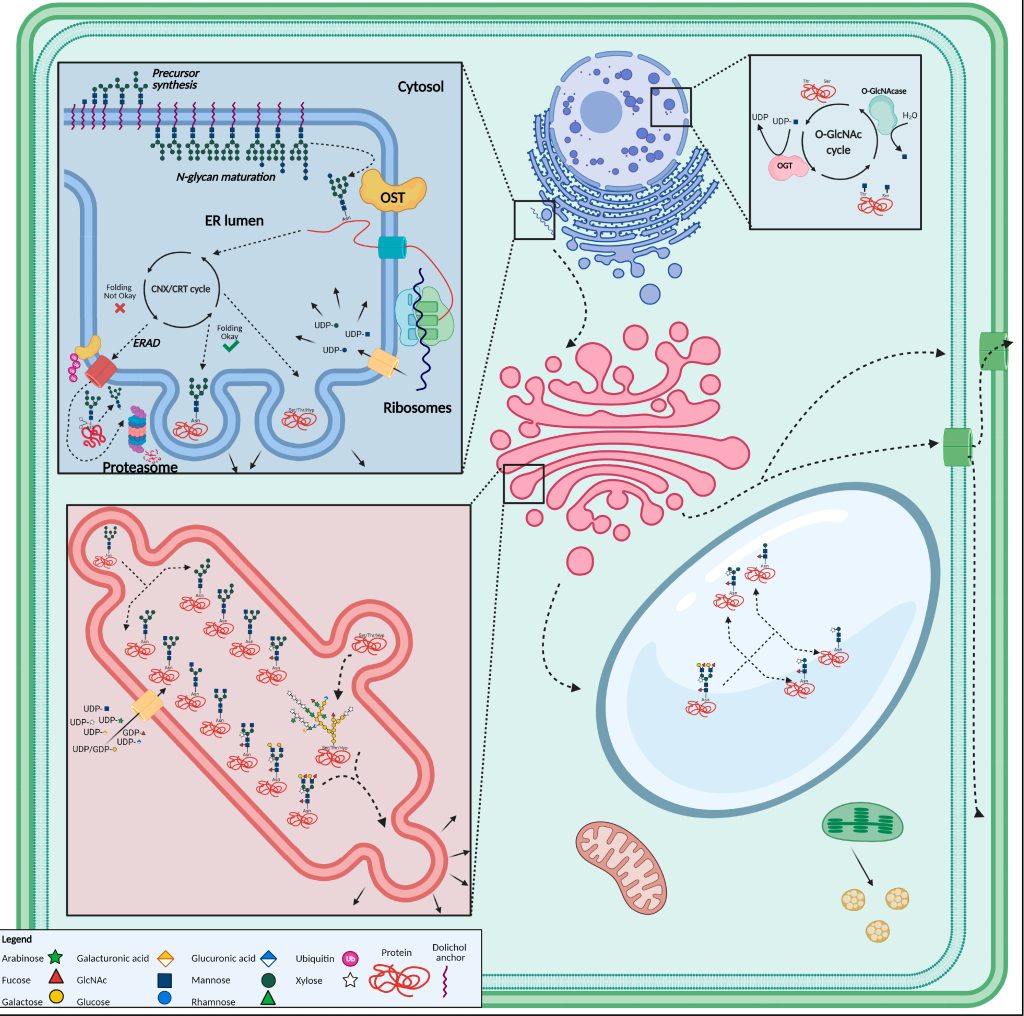
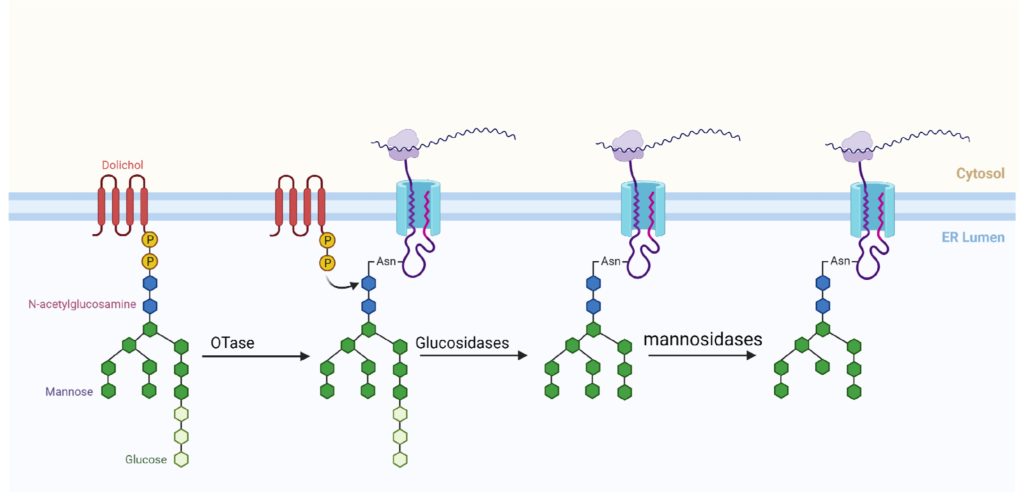
图1 植物细胞中N-糖基化结构示意图(De Coninck et al., 2021)。N-聚糖的合成起始于内质网,在糖基转移酶的作用下聚糖分子链与蛋白质中的氨基残基连接形成糖蛋白,糖蛋白最终在高尔基体的囊膜中进行转运,经过进一步的修饰加工,会带上不同的糖链结构。
N-糖基化过程中,除了氨基酸序列具有一定的特征之外,聚糖也具有一定的特征。植物中常见的能够发生N-糖基化的聚糖形式主要包括3个葡萄糖(Glucose, Glc/Glu,本文中统一使用Glc),9个甘露糖(Mannose, Man)和2个N-乙酰氨基葡萄糖(N-acetylglucosamine, GlcNAc),即Glc3Man9GlcNAc2。
聚糖形成的主要场所是内质网,主要包括两个阶段:第一阶段主要发生在内质网的基质中,将两个GlcNAc连接到多萜醇(Dolichol)上,然后将五个Man与GlcNAc相连,生成Man5-GlcNAc2-Dolichol。第二阶段主要发生在内质网的管腔内侧,将四个Man和三个Glc分别连接到聚糖分子链中,得到成熟的聚糖(Glc3-Man9-GlcNAc2-Dolicol)。紧接着,在内质网的管腔中,特定的寡糖转移酶(Oligosaccharyl transferases, OTase)将成熟的聚糖转运到蛋白的天冬氨酸的氨基上,形成糖蛋白。
与蛋白质翻译后修饰N-糖基化不同,O-糖基化没有固定特征的氨基酸序列,糖链分子也没有固定的核心寡糖结构,糖链分子中聚糖类型、聚糖数量呈现多样性,在分离糖蛋白的过程中缺乏从蛋白质中水解O-聚糖的通用酶,因此O-糖基化是随机的、多变的且没有规律可循的,这些原因导致发生O-糖基化的糖蛋白结构更为复杂。
关于糖基化的修饰类型就介绍到这里,大家如果感兴趣的话可以自己去查相关的文献资料进行学习哦。接下来让我们一起来聊一聊蛋白质糖基化修饰位点的预测工具和检测技术。
如果我们有感兴趣的蛋白质,想知道此蛋白质是否会发生糖基化的修饰,首先比较便捷的方法就是通过一些在线的蛋白质糖基化位点预测工具或数据库进行分析。以下是一些常用的蛋白质糖基化位点预测的网站:
(1)NetNGlyc,用于预测蛋白质序列中潜在的N-糖基化位点,网址:http://www.cbs.dtu.dk/services/NetNGlyc/;
(2)NetOGlyc,用于预测蛋白质序列中潜在的O-糖基化位点,网址:http://www.cbs.dtu.dk/services/NetOGlyc/;
(3)PredGPI,用于预测蛋白质序列中潜在的GPI锚定位点,网址:http://gpcr.biocomp.unibo.it/predgpi/;
(4)GPI-SOM,用于预测蛋白质中GPI锚定位点的神经网络工具,网址:http://gpi.unibe.ch/;
(5)UniProt,主要提供蛋白质序列的注释信息,同时也包括已知的糖基化修饰位点信息,网址:https://www.uniprot.org/。
通过一些在线的蛋白质糖基化修饰预测网站,我们可以找到可能会发生蛋白质糖基化修饰的一些蛋白,接下来可通过一些检测技术对其进行实验验证。糖基化修饰检测是鉴定蛋白质糖基化、糖基化修饰位点以及糖基化修饰水平的重要技术手段,常用的蛋白质糖基化修饰的检测技术主要包括蛋白质印迹、分子荧光标记法、凝胶电泳法、凝集素亲和技术和质谱技术等,并针对其中的凝集素亲和技术和质谱技术进行了举例。接下来让小远带大家一起了解一下吧。
2022年,东北林业大学程玉祥课题组在Forestry Research杂志上发表了一篇题为“Lectin affinity-basedglycoproteome analysis of the developing xylemin poplar”的研究论文。本研究以杨树(Populus simonii×P. nigra)为研究材料,探究杨树木质部蛋白质糖基化修饰水平对杨树发育的影响。研究人员通过凝集素亲和技术对杨树木质部中的糖蛋白进行了富集分离,并结合质谱进行鉴定。结果共鉴定出鉴定出154个糖蛋白质,功能分析显示这些糖蛋白主要包含氧化还原酶、蛋白酶和蛋白激酶。
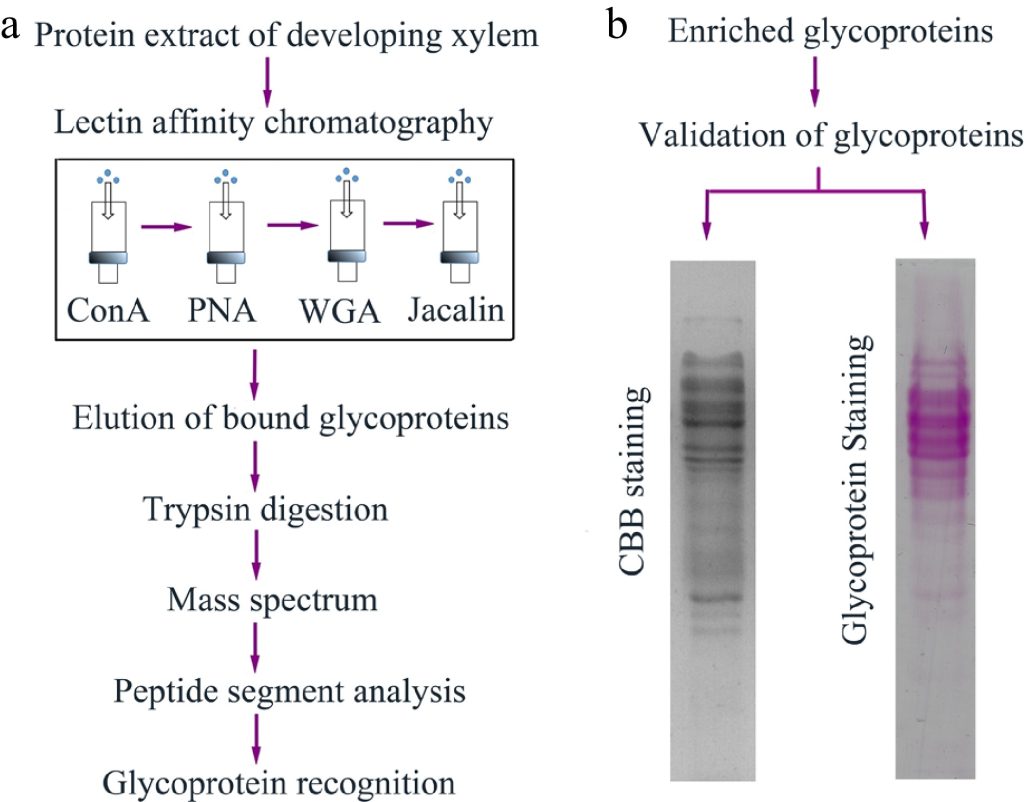
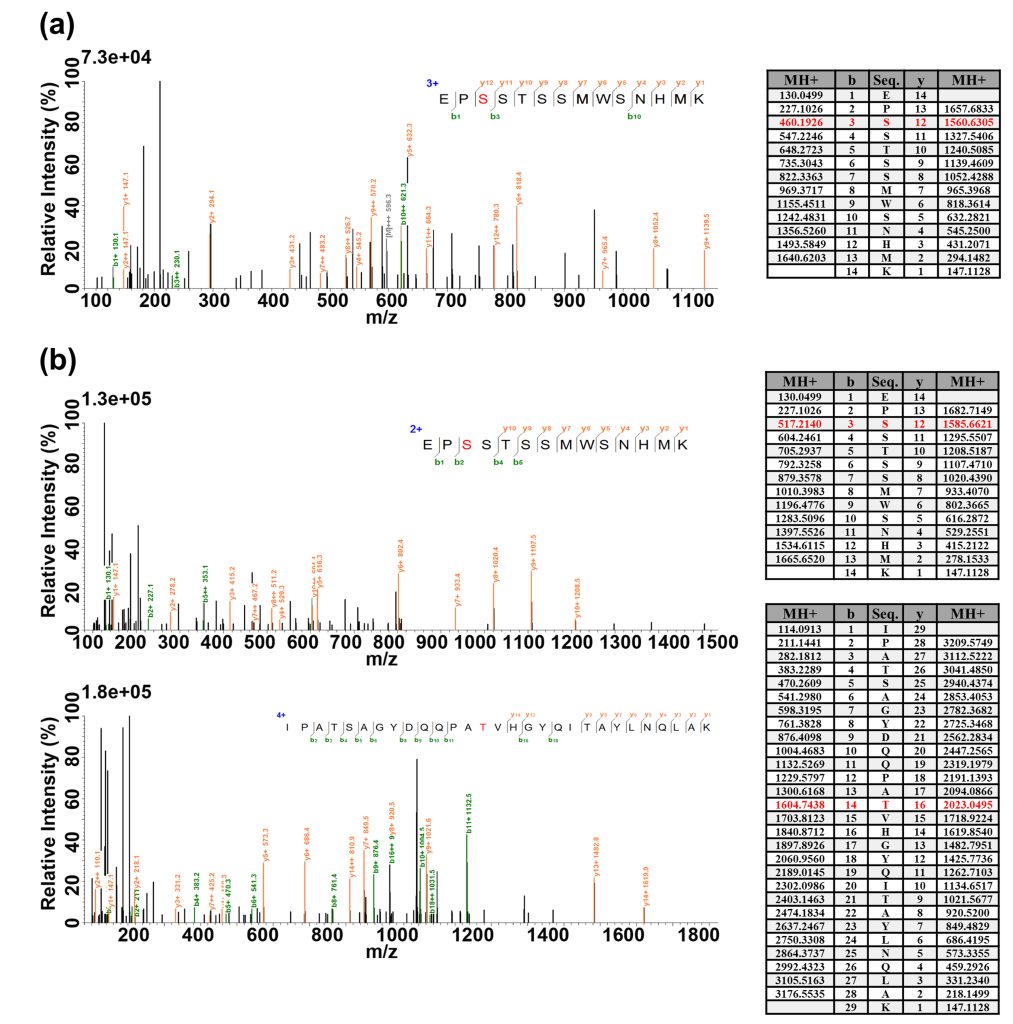
常用的质谱技术包括基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)和电喷雾电离质谱(ESI-MS)两种。MALDI-TOF-MS灵敏度高、可操作性强且对样品中的无机盐和缓冲溶液具有较好包容性,对大多数蛋白质具有较好的适用性。ESI-MS具有选择性好、质量分析范围宽和样品消耗量较少的特点,在分析时样品溶液是连续不断导入ESI源内的,可以直接与高效液相色谱联用,同时实现样品的分离与鉴定。
De Coninck T, Gistelinck K, Janse van Rensburg H C, et al. Sweet modifications modulate plant development[J]. Biomolecules, 2021, 11(5): 756.
Okagu I U, Ohanenye I C, Ezeorba T P C, et al. Phytoglycoproteins and human health: current knowledge and future applications[J]. Applied Sciences, 2021, 11(12): 5532.
Mulagapati S H R, Koppolu V, Raju T S. Decoding of O-linked glycosylation by mass spectrometry[J]. Biochemistry, 2017, 56(9): 1218-1226.
Beihammer G, Maresch D, Altmann F, et al. Glycosylphosphatidylinositol-anchor synthesis in plants: a glycobiology perspective[J]. Frontiers in Plant Science, 2020, 11: 611188.
Cheng H, Liu J, Zhou M, et al. Lectin affinity-based glycoproteome analysis of the developing xylem in poplar[J]. Forestry Research, 2022, 2(1).
Xu J, Liu S, Cai L, et al. SPINDLY interacts with EIN2 to facilitate ethylene signalling‐mediated fruit ripening in tomato[J]. Plant Biotechnology Journal, 2023, 21(1): 219-231.