通过将代谢组学高通量检测手段与正向遗传学设计相结合能够快速定位影响代谢物含量的位点并以此鉴定候选基因。根据所使用的遗传学群体类型不同,大致有mGWAS和mQTL两种研究方式,这两种方式均成功应用于小麦代谢物候选基因鉴定。然而,受限于所搜集自然材料的群体结构(GWAS),以及人工分离群体有限的变异来源(QTL),使得两种研究方式均具有其自身的局限性。因此,通过结合mGWAS和mQTL预计能够鉴定更多的候选基因,这种结合两种遗传学群体进行代谢组学研究的方式在小麦中尚未见报道。

在本项研究中,作者首先通过一个mGWAS和mQTL共定位位点鉴定到一个MYB编码基因,该基因通过调控花青素代谢物含量影响小麦籽粒颜色。接下来,作者分别从mQTL结果以及mGWAS结果独立鉴定候选基因并解析对应代谢路径。最后,通过同一个代谢物的mGWAS结果和mQTL结果分别鉴定到黄酮糖基转移酶基因(GT)和甲基转移酶基因(MT),并初步确定这两个酶对于黄酮底物的作用方式为先甲基化后糖基化修饰(图1)。以上结果表明,通过使用不同类型的群体,能够在多个层面(两个群体共定位、每一个群体定位结果、以及两个群体互补)快速鉴定候选基因,助力代谢通路解析。
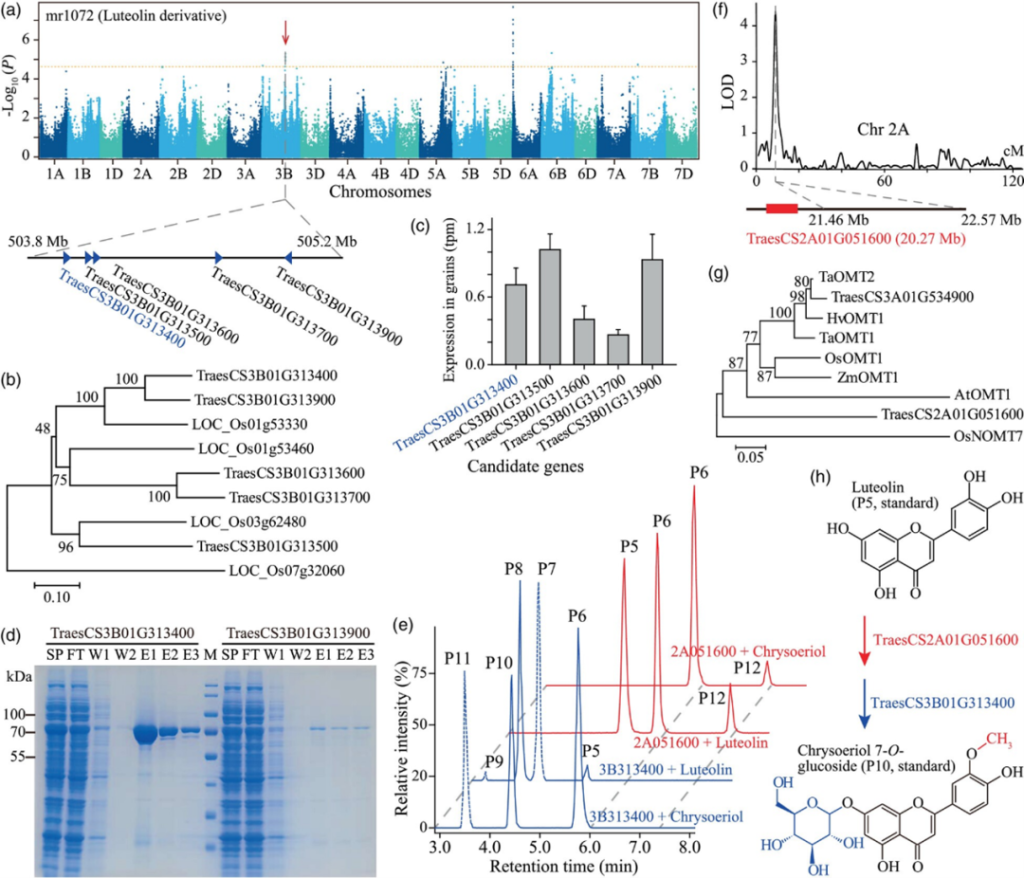
图1 结合mGWAS和mQTL定位结果进一步助力代谢路径解析
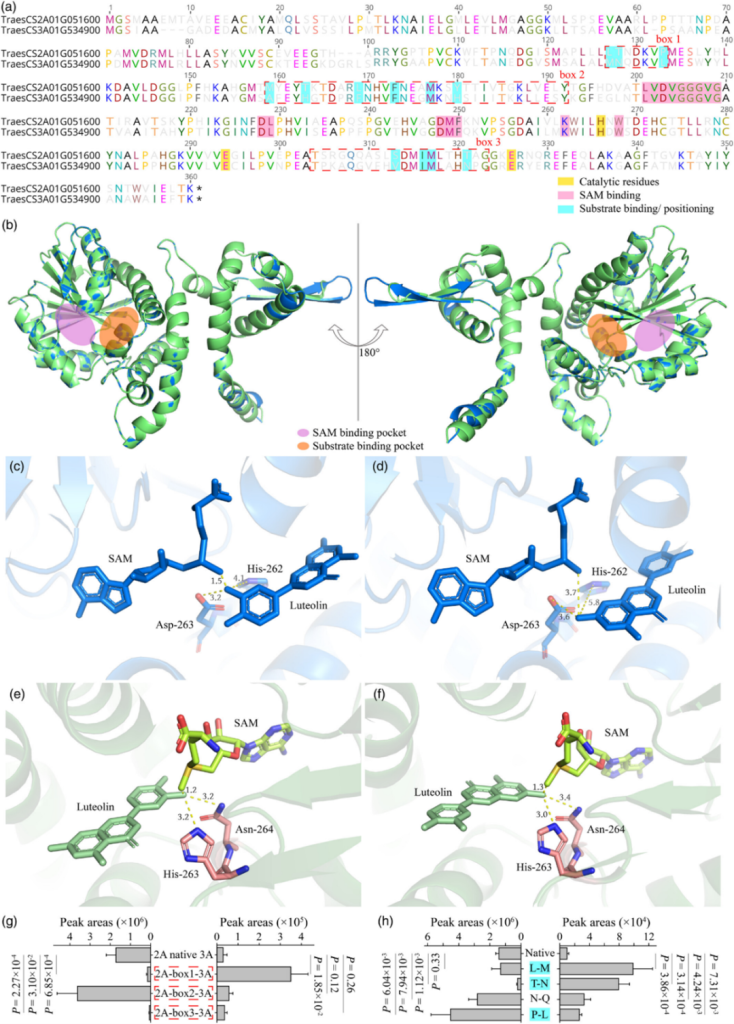
作者还注意到所鉴定到的两个MT活性特征有所差别,通过比较两个MT的氨基酸序列发现二者存在大量序列差异(图2a),该结果不能帮助有效锁定关键的差异性氨基酸。通过比较两个MT的蛋白质三维结构发现尽管二者氨基酸序列差别较大,其分子结构相似度较高,特别是对于催化活性至关重要的甲基供体口袋和底物催化口袋(图2b)。进一步使用分子对接发现,TraesCS3A01G534900不能催化7号位羟基发生甲基化修饰的原因为该位置羟基与甲基化供体以及口袋中关键氨基酸残基的空间距离过大(图2c-d),而在TraesCS2A01G051600中底物7号位羟基与甲基化供体以及口袋中关键氨基酸残基的空间距离均处于合理范围(图2e-f)。结合前人相关研究结果,对两个MT中可能影响底物位置的氨基酸片段(图2a中,红色虚线方框所示)进行调换,结果表明第一段氨基酸序列(box 1,图2g)可能为关键序列。进一步对该段氨基酸序列中四个不同的氨基酸残基分别进行调换,结果表明苏氨酸–天冬酰胺(T–N)可能为关键的氨基酸残基(图4h)。
图2 造成两个MT之间催化活性差异的关键序列解析
上述研究内容于2024年2月26日在线发表于Plant Biotechnology Journal,论文题为“Beyond pathways: accelerated flavonoids candidate identification and novel exploration of enzymatic properties using combined mapping populations of wheat”。三位共同第一作者中,陈杰为已出站博士后,目前就职于崖州湾国家实验室;张月琪为华中农业大学小麦改良创新团队在读博士生;卫佳琪为团队已毕业研究生,目前就职于武汉市农业科学院。华中农业大学作物遗传改良全国重点实验室陈伟教授为论文通讯作者。除本团队其他成员参与研究外,中国农业科学院作物科学研究所的何中虎研究员、郝元峰博士、田文斐博士,马普所的Alisdair R. Fernie教授参与了该研究的指导。该研究得到了科技创新2030-重大项目等的资助。