本文内容速览:
蛋白质翻译后修饰调节蛋白质功能,对生物学和疾病研究具有重要意义。科研工作者一直在不断深入探索蛋白质翻译后修饰的领域,其中包括磷酸化、泛素化、乙酰化等多种类型。在前几篇推文中,小远已经向大家介绍了一些常见的蛋白质翻译后修饰类型,包括磷酸化和泛素化。如果你对这些修饰类型感兴趣,不妨点击查看详细信息哦!“蛋白翻译后修饰——泛素化、蛋白翻译后修饰——泛素化(二)、蛋白翻译后修饰——磷酸化(一)、蛋白翻译后修饰——磷酸化(二)”。今天,小远将为大家带来的是另一个备受瞩目的“嘉宾”——乙酰化修饰。让我们紧跟小远的脚步,一同去探索乙酰化的奥秘吧!
蛋白N端乙酰化修饰大部分发生在真核生物中,由N端乙酰转移酶(N-acetyltransferases, NATS)催化,目前还没有发现N端去乙酰化酶,因此认为N端乙酰化是不可逆的(王旭初等, 2022);赖氨酸上的乙酰化修饰在真核生物及原核生物中均广泛存在,主要由赖氨酸乙酰转移酶(lysine acetyltransferases, KATs)和赖氨酸去乙酰化酶(lysine deacetylases, KDACs)催化,是一个可逆的过程。
2022年9月,华中农业大学郭文武课题组在Plant Physiology杂志上发表了一篇题为“Acetylome reprograming participates in the establishment of fruit metabolism during polyploidization in citrus”的研究论文。作者使用质谱检测技术,对柑橘异源四倍体杂种及其双亲果肉组织进行了分析。研究发现,大量非组蛋白类蛋白被鉴定存在乙酰化修饰,共鉴定出超过1600个蛋白的4000多个乙酰化位点。
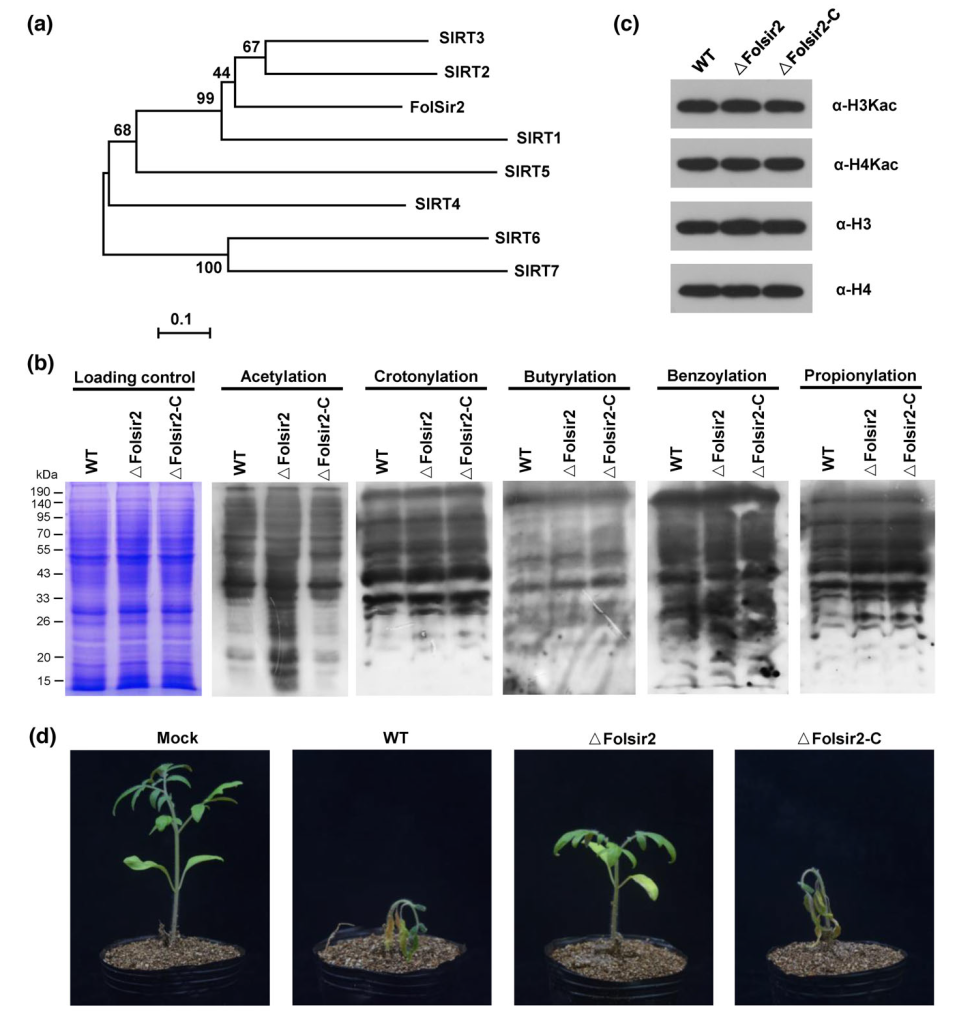
2023年11月,青岛农业大学梁文星课题组在New Phytologist杂志上发表了一篇题为“Sir2-mediated cytoplasmic deacetylation facilitates pathogenic fungi infection in host plants”的研究论文。该研究揭示了尖孢镰刀菌通过细胞质蛋白乙酰化调控病原菌致病性的新机制,作者利用亚细胞定位和乙酰化修饰免疫印迹检测,在尖孢镰刀菌番茄专化型(Fusarium oxysporum f. sp. lycopersici, Fol)中鉴定出了一个定位于细胞质的去乙酰化酶FolSir2,并发现敲除体ΔFolSir2致病力显著下降。

蛋白质乙酰化作为一种重要的蛋白质修饰方式,在细胞功能和信号转导中发挥着关键的作用。为了更全面地研究蛋白质乙酰化的功能和机制,我们可以运用多种方法进行探究。在上文的方法介绍中,小远只是重点突出了所要讲述的技术,但是统观整篇文章,作者会同时使用多种探究方法,感兴趣的小伙伴可以自行学习。
上述三种方法是目前应用较为广泛的方法,但小远还听说高通量蛋白质组学、化学生物学以及生物信息学等领域正在不断发展,有望对蛋白质乙酰化修饰的解析提供巨大的帮助,期待值已经被拉满。
已有研究表明拟南芥去乙酰化酶SOBER1能抑制效应蛋白HopZ5和AvrBsT激发的植物免疫反应。在本研究中作者发现在缺乏SOBER1的情况下,这两个病原菌效应蛋白激活的免疫反应依赖于RIN4和细胞内的抗病蛋白RPM1。作者通过选取拟南芥SOBER1功能缺失突变体sober1-3,简称为sober1,来进行乙酰转移酶效应蛋白触发超敏反应(hypersensitivity reaction, HR)的依赖性验证。携带效应蛋白HopZ5或AvrBsT的Pto DC3000病原菌在sober1 rpm1和sober1 rps2 rin4突变体中的生长与携带空载的Pto DC3000菌株相当,但与sober1 rps2突变体中的不同,这表明在拟南芥中,HopZ5或AvrBsT触发的免疫需要RPM1和RIN4,而不是RPS2。综上所述,细菌乙酰转移酶HopZ5和AvrBsT可能靶向RIN4,激活RPM1依赖性免疫。
图6 细菌乙酰转移酶效应蛋白HopZ5和AvrBsT触发拟南芥中RPM1和RIN4依赖性免疫(Choi et al., 2021)。
有其它的研究表明,另外两个效应蛋白AvrB和AvrRpm1能通过胞质类受体激酶RIPK,诱导RIN4第166位苏氨酸(T166)的磷酸化并激活RPM1依赖的免疫反应。因此,作者进一步研究了HopZ5和AvrBsT同RIN4的互作关系。
通过免疫共沉淀实验,研究人员发现HopZ5和AvrBsT与RIN4之间存在相互作用(图7A)。有趣的是,通过体外反应和质谱鉴定,研究人员发现作为乙酰化酶的HopZ5和AvrBsT能够对RIN4的保守位点T166进行乙酰化修饰(图7B、C)。这种乙酰化修饰不仅在体外反应中存在,在植物体内和病原菌侵染过程中也存在(图7D、E)。最终,RIN4 T166位点的乙酰化修饰将激活RPM1依赖的免疫反应,从而使植物能够抵御病原菌的侵袭,并展示出抗病性。
这项研究的结果表明,HopZ5和AvrBsT通过直接乙酰化RIN4蛋白激活RPM1依赖的免疫反应。这揭示了一种新的机制,即通过乙酰化修饰调控植物免疫反应。这项研究的发现对于深入理解植物与病原菌的相互作用、揭示免疫信号转导的机制以及开发抗病技术具有重要意义。
2022年1月,中国科学院分子植物科学卓越创新中心张蘅课题组在Stress Biology杂志上发表了一篇文章题为“Acetylproteomics analyses reveal critical features of lysine-ε-acetylation in Arabidopsis and a role of 14-3-3 protein acetylation in alkaline response”的研究论文,解析了拟南芥主要器官的蛋白质乙酰化,分析了植物赖氨酸乙酰化的关键特征,并揭示了14-3-3蛋白乙酰化在植物碱胁迫应答中的重要作用。
在这项研究中,作者使用高分辨率串联质谱仪对包括拟南芥的叶、根、茎、花和种子在内的五个具有代表性的植物器官进行了赖氨酸乙酰化分析,共鉴定到2887个Kac(Lysine-ε-acetylation)蛋白和5929个Kac位点。进化分析发现,Kac会优先靶向进化上保守的蛋白质和保守的赖氨酸。
作者观察到许多Kac蛋白质同时也会受到其他翻译后修饰(如磷酸化、泛素化、SUMO化)的调控,尽管乙酰化、泛素化和SUMO化都能修饰赖氨酸残基,但它们很少在相同的位点上发生修饰。被乙酰化和磷酸化共同靶向的蛋白质通常富含许多溴结构域和14-3-3蛋白结构域。结构分析显示,14-3-3蛋白结合磷酸化修饰底物的结合位点处的第56位赖氨酸乙酰化(K56ac)可能阻断其与磷酸化底物的结合(图8A)。之前的研究表明磷酸化识别蛋白14-3-3s参与质膜H+-ATPase AHA2活性的调节,同时14-3-3s与AHA2之间的相互作用会受到14-3-3s蛋白自身翻译后修饰的调控。
为了进一步探索14-3-3蛋白乙酰化的生物学功能,作者开发了一个酵母外源表达系统进行验证。在缺失内源14-3-3和H+-ATPase蛋白的酵母中,转入了拟南芥AHA2和14-3-3蛋白GRF6,构建了乙酰化(K56Q)和去乙酰化(K56R)突变体(图8C)。通过检测酵母细胞外溶液的pH变化,结果表明GRF6中Lys56的乙酰化对AHA2的活性具有负调控作用。拟南芥的转基因实验也验证了GRF6 Lys56的乙酰化在植物碱胁迫应答中的负调控作用(图8D、E)。在grf6 grf8双突变背景中,转入GRF6 K56Q无法恢复植物在高pH环境下的正常生长,并且在高pH环境中GRF6的乙酰化水平降低(图8F、G)。因此,该研究揭示了蛋白质乙酰化通过调节14-3-3蛋白与AHA2的相互作用来调节植物碱胁迫应答的新机制。
王旭初, 阮松林, 徐平等. 植物蛋白质组学[M]. 北京:科学出版社, 2022.1.
张赫, 张士猛, 周平坤. 乙酰基转移酶Tip60 (KAT5)的功能研究进展[J]. 生物化学与生物物理进展, 2015, 42(1): 25-31.
吕斌娜, 梁文星. 蛋白质乙酰化修饰研究进展[J]. 生物技术通报, 2015, 31(4): 166.
Aksnes H, Drazic A, Marie M, et al. First things first: vital protein marks by N-terminal acetyltransferases[J]. Trends in biochemical sciences, 2016, 41(9): 746-760.
Nagy Z, Tora L. Distinct GCN5/PCAF-containing complexes function as co-activators and are involved in transcription factor and global histone acetylation[J]. Oncogene, 2007, 26(37): 5341-5357.
Fournier M, Orpinell M, Grauffel C, et al. KAT2A/KAT2B-targeted acetylome reveals a role for PLK4 acetylation in preventing centrosome amplification[J]. Nature communications, 2016, 7(1): 13227.
Barneda-Zahonero B, Parra M. Histone deacetylases and cancer[J]. Molecular oncology, 2012, 6(6): 579-589.
Zhang M, Tan F Q, Fan Y J, et al. Acetylome reprograming participates in the establishment of fruit metabolism during polyploidization in citrus[J]. Plant Physiology, 2022, 190(4): 2519-2538.
Zhang N, Hu J, Liu Z, et al. Sir2‐mediated cytoplasmic deacetylation facilitates pathogenic fungi infection in host plants[J]. New Phytologist, 2023.
Choi S, Prokchorchik M, Lee H, et al. Direct acetylation of a conserved threonine of RIN4 by the bacterial effector HopZ5 or AvrBsT activates RPM1-dependent immunity in Arabidopsis[J]. Molecular Plant, 2021, 14(11): 1951-1960.
Guo J, Chai X, Mei Y, et al. Acetylproteomics analyses reveal critical features of lysine-ε-acetylation in Arabidopsis and a role of 14-3-3 protein acetylation in alkaline response[J]. Stress Biology, 2022, 2(1): 1.
Wang R, Sun H, Wang G, et al. Imbalance of lysine acetylation contributes to the pathogenesis of Parkinson’s disease[J]. International Journal of Molecular Sciences, 2020, 21(19): 7182.
Chen X, Duan Y, Qiao F, et al. A secreted fungal effector suppresses rice immunity through host histone hypoacetylation[J]. New Phytologist, 2022, 235(5): 1977-1994.