藜麦(Chenopodium quinoa)是一种原产于南美中部安第斯山脉的重要农作物。其最为显著的表型特征之一是种子颜色。种子颜色的变化主要受到甜菜碱对比丰度的调控,这是一类强效抗氧化剂和清除自由基的色素,仅在石竹目植物中发现。然而,种子中这些色素的遗传基础仍有待确定。
近日,奥地利自然资源与生命科学大学计算生物学研究所Felix在Plant Biotechnology Journal上发表了一篇题为“Genomic basis of seed colour in quinoa inferred from variant patterns using extreme gradient boosting”的研究论文。该研究通过机器学习(极端梯度提升)的方法预测了种子颜色的遗传变异,并表明极端梯度提升在性能上优于传统的全基因组关联分析。展示了机器学习从大规模测序数据集中提取有效信息的强大功能。
 作者对源自玻利维亚和秘鲁的106个藜麦种质进行了测序,并将其与之前测序的50个种质合并。这156个种质的测序覆盖范围从3.5倍(20,050,178条reads,读长125bp)到7.0倍(40,770,980条reads),平均覆盖为5.2倍。为了最大程度的提高多样性,选择了不同的采样地点,并根据IPK基因库提供的数据信息选择了不同的种子颜色。在测序后,作者对测序数据进行了严格的质量控制和修剪,然后将所有156个种质与玻利维亚参考基因型CHEN125的组装基因组进行比对,将其命名为“RefCHEN125”,随后进行了变异调用。接下来,作者对所有种质进行了联合基因分型,过滤了次等位基因频率和测序覆盖,并创建了一个基因型矩阵。
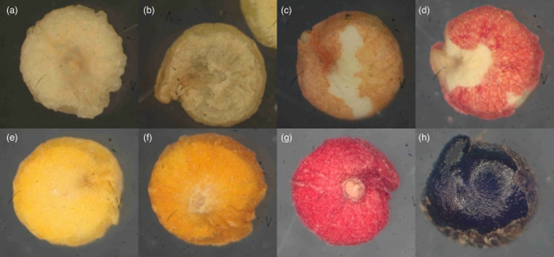
根据IPK基因库每份种质的数据信息,作者(每个种质约20-50个种子)修订了这13种不同的颜色描述,并将它们重新分为9个不同的种子颜色组。虽然白色、米色和橙色种子的颜色相对一致,但黄色种子的种质呈现出较广泛的黄色变异,其中一些种质呈鲜艳的黄色,更接近橙色种质,一些稍微呈微绿色,但大多数种质呈适度的黄色,更接近白色种质。作者只有少量的具有褐色、微绿红色、红色和黑色种子的种质。总体而言,这导致了一个不平衡的数据集,即并非所有组别都有相等的代表性。“微绿红色”的组别包括那些在每个样本中显示相似结构和大小的种子,并呈现出混合的红色和绿色色调的种质。最后,有一组种质,其种子呈现出各种不同的颜色,作者称之为“混合颜色”。
作者比较了米色、橙色和白色三组之间 129 个变异位点的基因型。区分橙色、白色和米色种质的图案清晰可见,然而,这些图案也表明,藜麦的种子颜色不能简化为单一的变异位点,而是复杂的变异位点组合。随后,作者将剩余的小组,包括黑色、红色、褐色和微绿红色(每组六到七个种质),纳入线性判别分析(LDA)中。基于之前确定的129个变异位点,得到的聚类清晰地将所有八个颜色组彼此分隔开。当包括具有混合种子颜色特征的种质时,它们无法被分配到明确的组别中。
作者将这些结果与使用线性混合模型(LMM)的经典单变量GWAS进行了比较,如GEMMA。所有常见的单变量 LMM 算法都不允许对非有序性状进行多类分类数据分析,因此作者进行了多轮GWAS,通过执行三个主要种子颜色(米色、橙色和白色)之间的成对比较。随后,作者对所有可能的组合进行了严格的Bonferroni校正,然而即使在校正之后,GWAS仍然鉴定出1073个显著变异位点(比XGBoost多6.8倍)。
总体而言,作者通过从基因组测序数据中调用变异位点信息,可以对植物进行基于其种子颜色的分类,并且提出的工作流程可用于分析各种表型特征。极端梯度提升不仅限于分类特征,也可以应用于诸如产量或生物量等重要的植物性状。
作者对源自玻利维亚和秘鲁的106个藜麦种质进行了测序,并将其与之前测序的50个种质合并。这156个种质的测序覆盖范围从3.5倍(20,050,178条reads,读长125bp)到7.0倍(40,770,980条reads),平均覆盖为5.2倍。为了最大程度的提高多样性,选择了不同的采样地点,并根据IPK基因库提供的数据信息选择了不同的种子颜色。在测序后,作者对测序数据进行了严格的质量控制和修剪,然后将所有156个种质与玻利维亚参考基因型CHEN125的组装基因组进行比对,将其命名为“RefCHEN125”,随后进行了变异调用。接下来,作者对所有种质进行了联合基因分型,过滤了次等位基因频率和测序覆盖,并创建了一个基因型矩阵。
根据IPK基因库每份种质的数据信息,作者(每个种质约20-50个种子)修订了这13种不同的颜色描述,并将它们重新分为9个不同的种子颜色组。虽然白色、米色和橙色种子的颜色相对一致,但黄色种子的种质呈现出较广泛的黄色变异,其中一些种质呈鲜艳的黄色,更接近橙色种质,一些稍微呈微绿色,但大多数种质呈适度的黄色,更接近白色种质。作者只有少量的具有褐色、微绿红色、红色和黑色种子的种质。总体而言,这导致了一个不平衡的数据集,即并非所有组别都有相等的代表性。“微绿红色”的组别包括那些在每个样本中显示相似结构和大小的种子,并呈现出混合的红色和绿色色调的种质。最后,有一组种质,其种子呈现出各种不同的颜色,作者称之为“混合颜色”。
作者比较了米色、橙色和白色三组之间 129 个变异位点的基因型。区分橙色、白色和米色种质的图案清晰可见,然而,这些图案也表明,藜麦的种子颜色不能简化为单一的变异位点,而是复杂的变异位点组合。随后,作者将剩余的小组,包括黑色、红色、褐色和微绿红色(每组六到七个种质),纳入线性判别分析(LDA)中。基于之前确定的129个变异位点,得到的聚类清晰地将所有八个颜色组彼此分隔开。当包括具有混合种子颜色特征的种质时,它们无法被分配到明确的组别中。
作者将这些结果与使用线性混合模型(LMM)的经典单变量GWAS进行了比较,如GEMMA。所有常见的单变量 LMM 算法都不允许对非有序性状进行多类分类数据分析,因此作者进行了多轮GWAS,通过执行三个主要种子颜色(米色、橙色和白色)之间的成对比较。随后,作者对所有可能的组合进行了严格的Bonferroni校正,然而即使在校正之后,GWAS仍然鉴定出1073个显著变异位点(比XGBoost多6.8倍)。
总体而言,作者通过从基因组测序数据中调用变异位点信息,可以对植物进行基于其种子颜色的分类,并且提出的工作流程可用于分析各种表型特征。极端梯度提升不仅限于分类特征,也可以应用于诸如产量或生物量等重要的植物性状。