最初采用84种细菌和22种真菌组成多合成群落(SynComs)对无菌拟南芥的根系培养,采用宏基因组学分析土壤-根系表面的细菌和真菌群落的转录组结构的变化。通过宏基因组的测序结果表明:土壤基质样品的41.9%的来源于微生物基因组,根系样品的8.7%来源于微生物基因组。其中7300万条reads可以映射到总84种细菌中的73种细菌基因组中;真菌中发现只有特定的几种高转录覆盖率。初步表明植物根系定殖微生物群落的的现象。
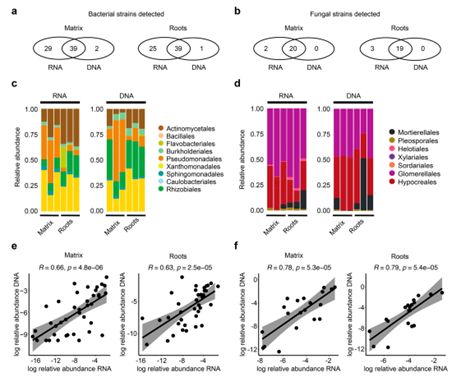
图2:对多合成群落(SynComs)的RNA和DNA的分析探究不同菌株之间的比较。
分别对土壤基质和根系中获取的DNA和RNA的数据直接反应菌株的丰富度,图2显示土壤基质和根系的细菌和真菌的相对丰富度的差异。

通过图3a热图显示根系20种高覆盖的菌株和土壤基质比较。根系的微生物群落的表达差异性分析显示前200个高表达基因(OG)进行聚类分析发现,其中几个基因的高表达和引起根系诱导群落丰富度高度相关,因此可能解释了细菌的丰富度的变化原因。通过图3b、c的研究表明:在植物的表达激活和根部微生物丰富度密切相关,不同的细菌利用根系的初级代谢物的能力和根系微生物菌株占主导地位之间存在联系。
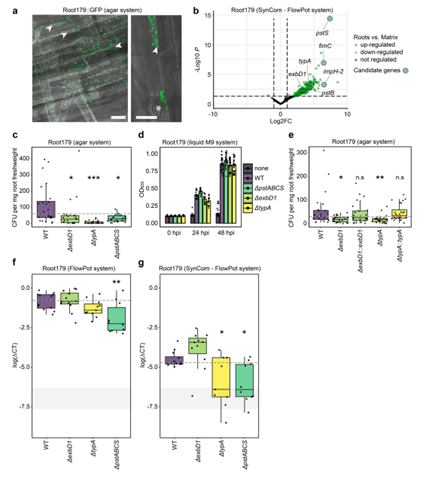
图4:参与拟南芥根系定殖定候选基因验证,利用罗丹杆菌的Root179标记验证
采用GFP荧光对Root179基因进行标记,在14dpi下测定拟南芥的根系表面的荧光。通过荧光显示细菌中植物根系表皮凹槽处积累。对根系和土壤基质样品的基因差异表达比较,Root179中差异表达基因的情况。对Root179野生性和相应缺失突变体的根系定殖能力的比较,在土壤基质下发现缺失突变体的Root179在根系的定殖性低于野生性。在液体培养中发现Root179野生性和相应缺失突变体的根系定殖能力差异不大,可能是磷酸盐对细菌环境的伤害。
根系相关微生物群落的结构和功能结构是调节自然界中植物生长和性能的驱动力。我们对根系定殖过程中复杂微生物群落中激活的微生物过程和功能的理解仍然不足。通过实验验证了Root179的两个基因(typA、exbD)和一个完整的操纵子(pstABCS)作为根定植决定簇的重要性。typA和pst基因(A、B、C和S)都是细菌群落中最常见的上调基因之一,而exbD在Root179中特异性上调。编码磷酸盐转运系统的整个pstABCS操纵子(但不是单独的pstS或pstB)促进了Root179根定植,这表明成功的根定植需要有效的磷酸盐摄取。
文章来源:植物生物技术Pbj
