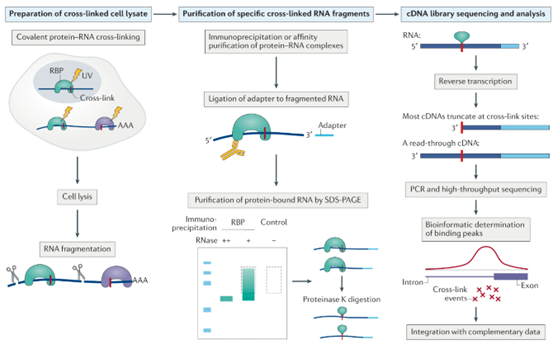
图一.传统CLIP实验流程
(Hafner, M., Katsantoni, M., Köster, T. et al. CLIP and complementary methods. Nat Rev Methods Primers 1, 20 (2021). https://doi.org/10.1038/s43586-021-00018-1)
近日,来自美国加州大学旧金山分校的K. Mark Ansel和David J. Erle团队,在《Genome Biology》杂志发表了题为“GCLiPP: global crosslinking and protein purification method for constructing high-resolution occupancy maps for RNA binding proteins”的文章。作者开发了全局交联蛋白纯化 (GCLiPP)的方法,并创建了小鼠原代T细胞和人类Jurkat T细胞系的全局RBP结合图谱。
GCLiPP方法与eCLIP具有许多相同的技术特征,并且无需RBP特异性免疫沉淀即可产生高分辨率转录组范围内的蛋白质结合数据。作者利用GCLiPP序列reads密度的峰,并测量这些峰内 GCLiPP 读数的分布,以评估该技术的重现性。与其他转录组特征相比,Jurkat GCLiPP文库的reads在成熟 mRNA和长非编码RNA中高度富集。
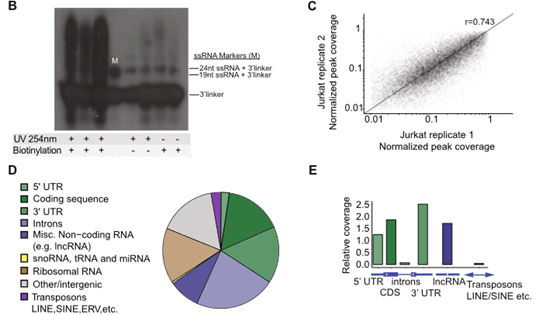 图二.GCLiPP测序揭示的RBP结合情况
为了评估GCliPP的性能,作者把GCliPP数据与eCLIP和基于相分离的方法进行比较。相分离方法产生更宽的峰,可能表明RBP位点图谱的分辨率较低。为了更好地评估分辨率,作者确定了每种技术检测到的RBP占据区域的系统发育保守性,推断功能性RBP-RNA相互作用位点比中性3’UTR 序列更保守。以峰为中心的200 nt区域的PhyloP分数对所有结合位点进行平均,然后对每种方法的平均值进行归一化(图 3E)。GCLiPP和eCLIP峰在峰中心显示出较高的序列保守性。eCLIP和GCLiPP(图 3F,上图)之间的归一化PhyloP分数在距离峰中心的每个核苷酸距离上的相关性比eCLIP和相分离方法之间的相关性更好(图 3F,中图和下图)。因此,GCLiPP以全局高分辨率地检测整个转录组中的RBP结合位点,与eCLIP数据非常相似。
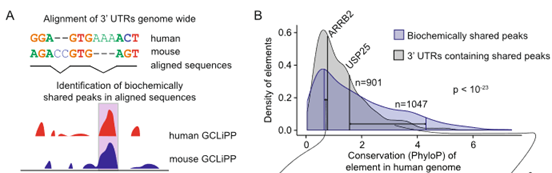
图四.跨物种RBP位点比较
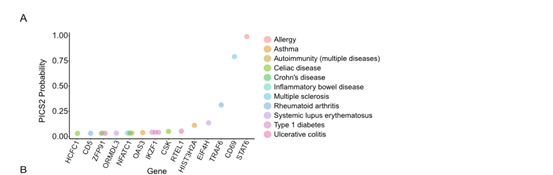
作者认为RBP结合图谱可以用来指导位于顺式调控元件中序列变异的功能注释。为了测试这种可能性,作者将Jurkat GCLiPP峰与可能与人类免疫介导性疾病相关的随机单核苷酸多态(SNPs)取交集。利用先前开发的算法,在这些变异中鉴定出63个出现在3‘UTR GCLiPP峰内的SNP。这些变异与多种免疫介导的疾病有关,并出现在T细胞中表达的各种基因中。
图五.GCLiPP峰与SNP位点交集
综上所述,作者开发了GCLiPP的方法构建全局高分辨率的RBP结合图谱,可以检测整个转录组范围内的RBP结合情况。GCLiPP以比基于相分离的技术更高的分辨率映射RBP占据的位点。GCLiPP序列标签与已知的RBP结合位点相对应,并在RBP特异性交联免疫沉淀 (CLIP) 检测到的位点富集。人类Jurkat T细胞和小鼠原代T细胞的比较揭示了人类和小鼠3’ UTR同源区域的GCLiPP信号共享峰。并且GCLiPP信号与免疫相关的SNPs重叠,说明了GCLiPP可以用于指导顺式调控元件中序列变异的功能注释。
图二.GCLiPP测序揭示的RBP结合情况
为了评估GCliPP的性能,作者把GCliPP数据与eCLIP和基于相分离的方法进行比较。相分离方法产生更宽的峰,可能表明RBP位点图谱的分辨率较低。为了更好地评估分辨率,作者确定了每种技术检测到的RBP占据区域的系统发育保守性,推断功能性RBP-RNA相互作用位点比中性3’UTR 序列更保守。以峰为中心的200 nt区域的PhyloP分数对所有结合位点进行平均,然后对每种方法的平均值进行归一化(图 3E)。GCLiPP和eCLIP峰在峰中心显示出较高的序列保守性。eCLIP和GCLiPP(图 3F,上图)之间的归一化PhyloP分数在距离峰中心的每个核苷酸距离上的相关性比eCLIP和相分离方法之间的相关性更好(图 3F,中图和下图)。因此,GCLiPP以全局高分辨率地检测整个转录组中的RBP结合位点,与eCLIP数据非常相似。
图四.跨物种RBP位点比较
作者认为RBP结合图谱可以用来指导位于顺式调控元件中序列变异的功能注释。为了测试这种可能性,作者将Jurkat GCLiPP峰与可能与人类免疫介导性疾病相关的随机单核苷酸多态(SNPs)取交集。利用先前开发的算法,在这些变异中鉴定出63个出现在3‘UTR GCLiPP峰内的SNP。这些变异与多种免疫介导的疾病有关,并出现在T细胞中表达的各种基因中。
图五.GCLiPP峰与SNP位点交集
综上所述,作者开发了GCLiPP的方法构建全局高分辨率的RBP结合图谱,可以检测整个转录组范围内的RBP结合情况。GCLiPP以比基于相分离的技术更高的分辨率映射RBP占据的位点。GCLiPP序列标签与已知的RBP结合位点相对应,并在RBP特异性交联免疫沉淀 (CLIP) 检测到的位点富集。人类Jurkat T细胞和小鼠原代T细胞的比较揭示了人类和小鼠3’ UTR同源区域的GCLiPP信号共享峰。并且GCLiPP信号与免疫相关的SNPs重叠,说明了GCLiPP可以用于指导顺式调控元件中序列变异的功能注释。