蛋白互作在细胞生命活动中起着关键作用,在不同时空层面上参与多种细胞过程,例如,调控细胞周期、蛋白质的合成与分泌、信号转导与代谢等。因此了解与研究蛋白质间的相互作用,是理解细胞内发生若干生物化学活动的第一步。在植物中研究蛋白互作时,大家常用的方法有:双分子荧光互补实验(BiFC)、荧光素酶互补实验(LCA)以及免疫共沉淀(Co-IP)等,那么除了这些常用的技术之外,还有其它的蛋白互作研究方法吗?答案当然是肯定的!在本期的推文中,小远就和大家一起来盘点一下在植物中研究蛋白互作的方法都有哪些吧!
本篇文章速览:
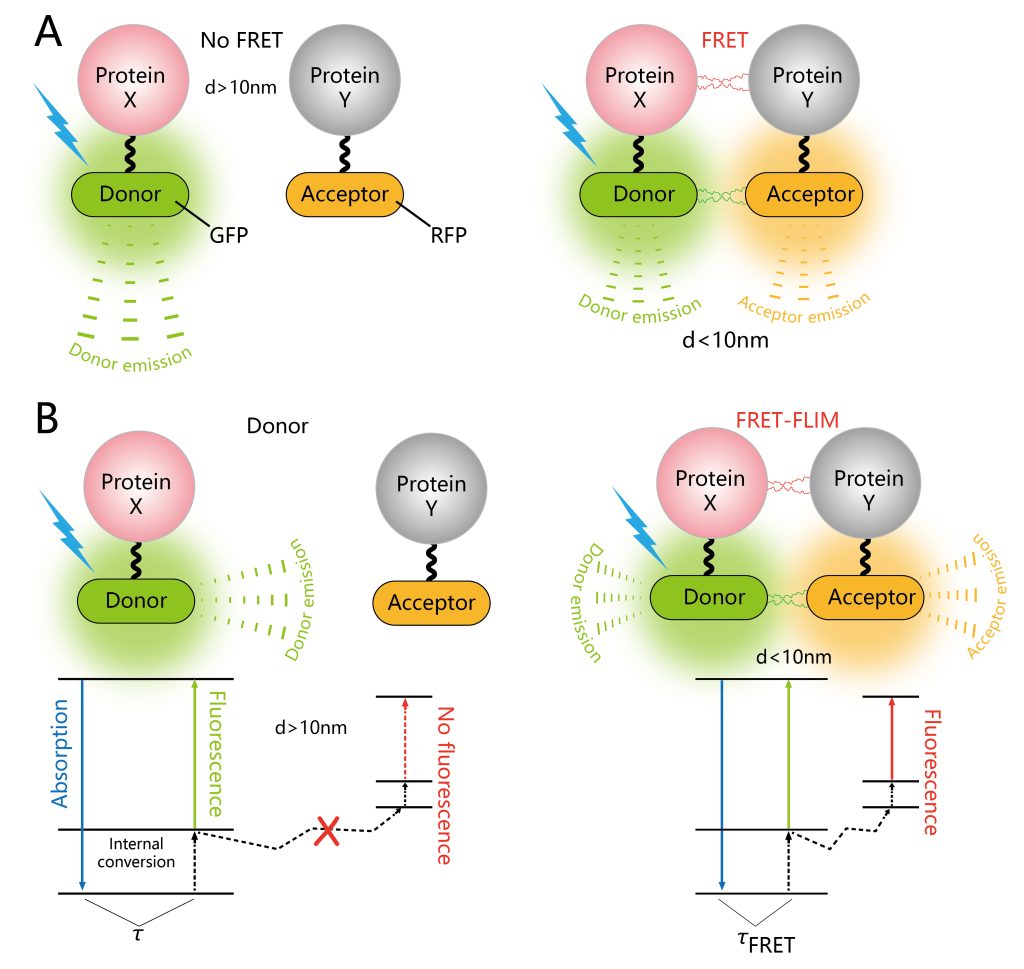
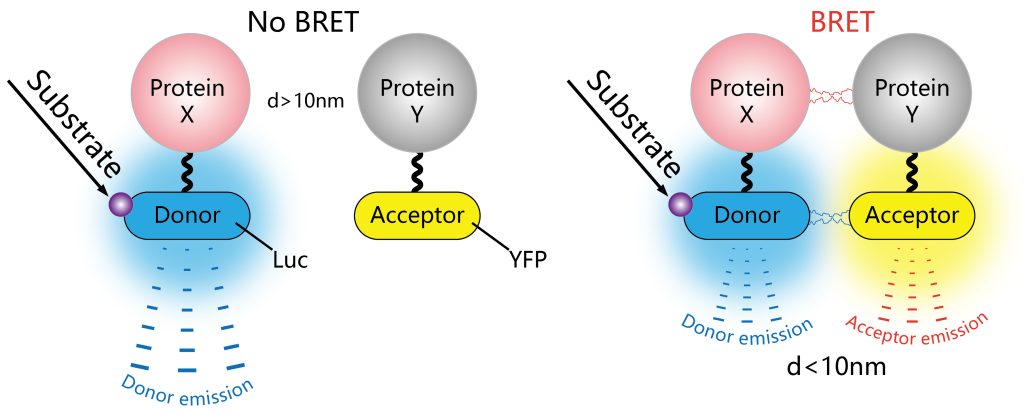
荧光共振能量转移(Förster resonance energy transfer,FRET)是一种物理过程,指两个携带不同荧光基团的大分子之间或同一分子的不同荧光团之间通过电偶极相互作用所发生的非辐射能量转移(Förster, 2012; Margineanu et al., 2016)。其依赖于两个分子之间密切的物理相互作用,例如标记蛋白质,如果它们之间的距离小于10nm,则可以进行非辐射(偶极-偶极)能量转移(Cui et al., 2019)(图9A)。传统的FRET过程分析通常是基于荧光强度成像来实现,分析的结果容易受光谱串扰的影响,因而灵敏度不高。荧光寿命显微成像(Fluorescence Life-time Imaging Microscopy,FLIM)技术是研究细胞内生命活动状态的一种非常可靠的方法。荧光寿命是指荧光团在返回基态之前处于激发态的平均时间,是荧光团的固有性质,它能十分灵敏地反映激发态分子与周围微环境的相互作用及能量转移。将FLIM技术应用到FRET过程分析中,利用FLIM技术可定量测量这一优势,可以非常灵敏地反映供体荧光分子与受体荧光分子之间的能量转移过程(图9B)。因此,利用FRET-FLIM能够实时检测植物细胞内的蛋白互作动态。
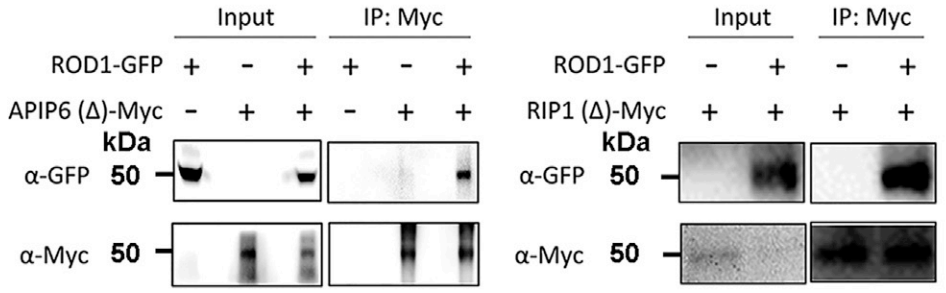
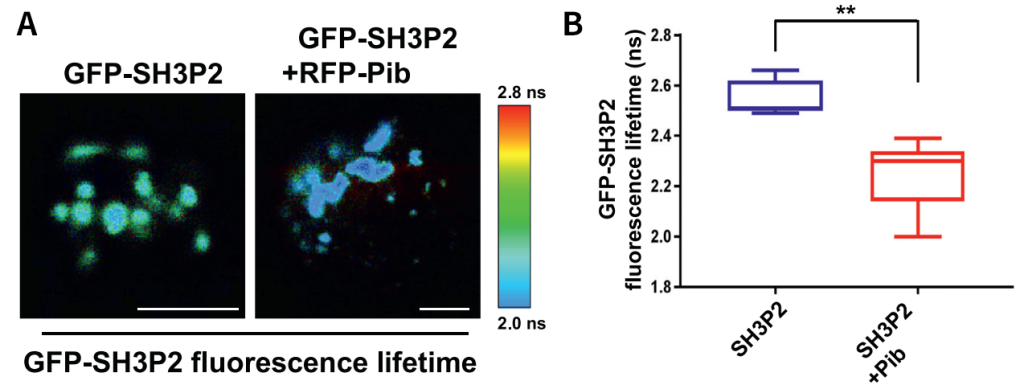
为了研究Pib蛋白触发对稻瘟病菌防御的机制,作者首先利用酵母双杂筛库鉴定出了多个与Pib结合的候选蛋白,并结合酵母双杂点对点实验,证实了SH3P2与Pib相互作用。接着,作者利用FRET-FLIM技术进一步分析了SH3P2与Pib在植物体内的相互作用。在水稻原生质体中通过FLIM-FRIT测量供体GFP-SH3P2的荧光寿命,发现供体单独表达时的平均荧光寿命为t=2.56ns,在与受体RFP-Pib共表达后其荧光寿命下降至t=2.24ns,这一结果表明SH3P2在体内与Pib相互作用(图10)。
另外,将荧光素酶代替荧光蛋白质作为能量供体,开发出的生物发光共振能量转移(Bioluminescence resonance energy transfer, BRET)技术(图11),可以有效解决传统FRET技术由于激发光导致的光毒性、光漂白等问题,不仅能降低样品背景噪音,还可提高检测灵敏度。不过,目前BRET技术在动物细胞中应用的更为广泛。
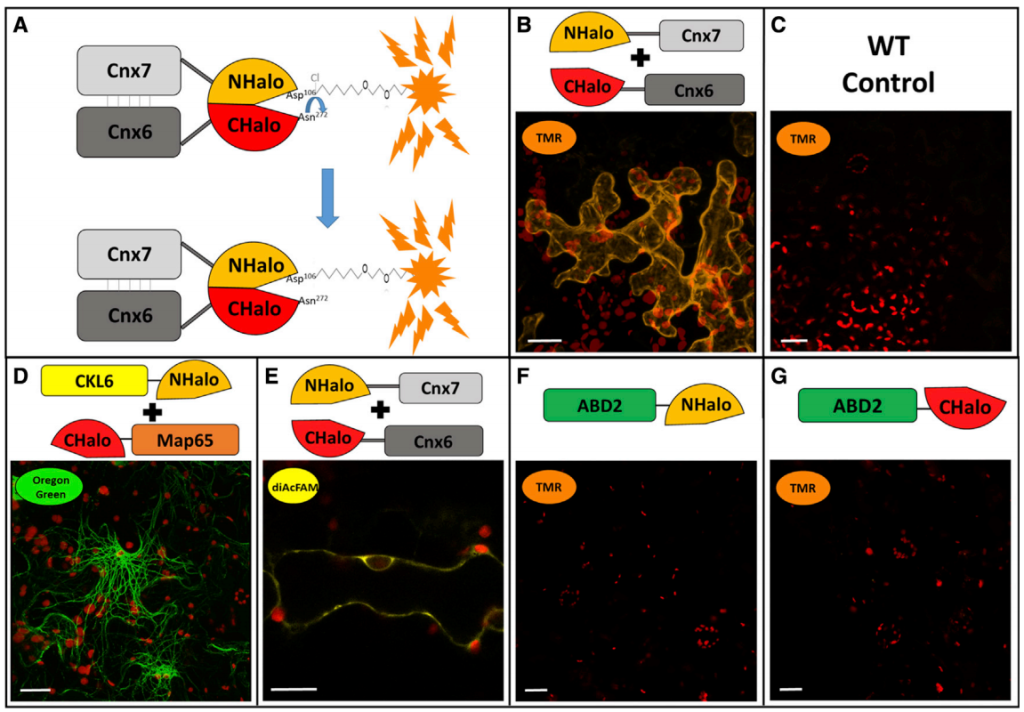
2021年6月,Minner-Meinen等人在Plant Communications杂志上发表了题为“Split-HaloTag imaging assay for sophisticated microscopy of protein–protein interactions in planta”的研究论文。该研究建立起了检测植物体内蛋白互作的新方法Split-HaloTag,克服了BiFC存在的假阳性高、容易光漂白等缺点。在本研究中,作者利用拟南芥Cnx6和Cnx7亚基组成的异源四聚体钼蝶呤合酶(MPT)复合物的形成来证明植物中Split‑HaloTag的重组。将NHalo、CHalo分别与Cnx7和Cnx6融合在一起,由于这两个亚基的相互作用使得NHalo和Chalo在空间上非常接近并重组成完整的蛋白,将Cnx6-NHalo和Cnx7-CHalo转化到烟草表皮细胞中并用荧光配体TMR染色后,就可以观察到明显的荧光信号。此外,重组的HaloTag还能够结合绿色荧光配体Oregon Green和黄色荧光配体DiAcFAM,并且单独表达NHalo或CHalo片段时无法实现与荧光配体的结合(图12)。
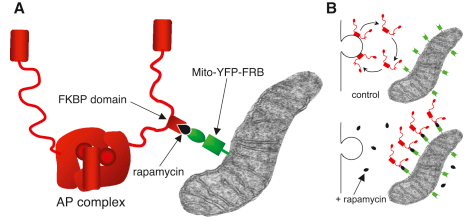
图13 Knocksideways系统工作原理(Robinson et al., 2010)。在雷帕霉素的作用下,锚定在线粒体上的FRB融合蛋白与FKBP融合蛋白形成异二聚体,从而使AP复合体定位在线粒体上。
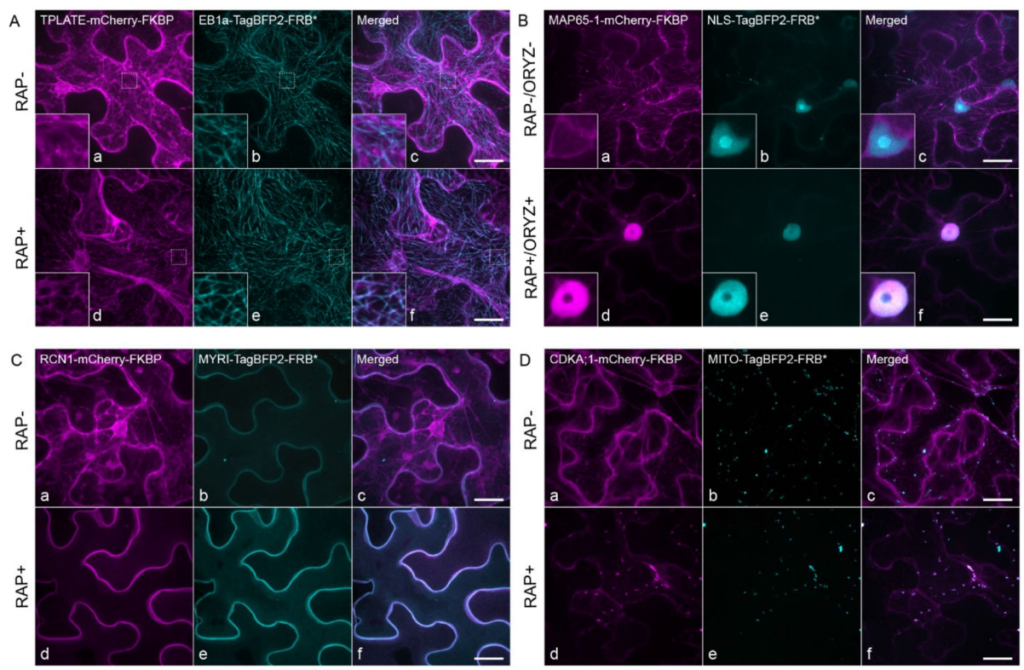
2021年1月,Winkler等人在The Plant Cell杂志上发表了题为“Visualizing protein-protein interactions in plants by rapamycin-dependent delocalization”的研究论文。在该论文中,作者基于Knocksideways系统,建立起了可应用于植物细胞中的KSP(Knocksideways in plants)蛋白互作检测系统,该系统与传统的蛋白互作研究技术BiFC相比,不再依赖于荧光基团的互补或基团之间的空间距离,并且可以在极短的时间内观察到稳定的结果。在KSP蛋白互作检测系统的创建过程中,作者首先将亚细胞定位信号、荧光蛋白与FRB融合在一起,获得了多种不同亚细胞定位的FRB融合蛋白。接着作者将亚细胞定位与FRB融合蛋白不同的诱饵蛋白与FKBP蛋白连接,通过在烟草中共表达这两个融合蛋白,并施用雷帕霉素后,FKBP融合蛋白会重新定位在FRB融合蛋白的细胞结构上(图14)。这一结果表明雷帕霉素在植物细胞中,也能够介导异二聚体的形成,说明KSP系统适用于植物细胞。
图14 雷帕霉素使FKBP融合蛋白重新定位在FRB融合蛋白的细胞结构上(Winkler et al., 2021)。未添加雷帕霉素(RAP-)和添加雷帕霉素(RAP+)的情况下瞬时表达mCherry-FKBP和TagBFP2-FRB*融合蛋白在烟草叶片中的亚细胞定位情况。
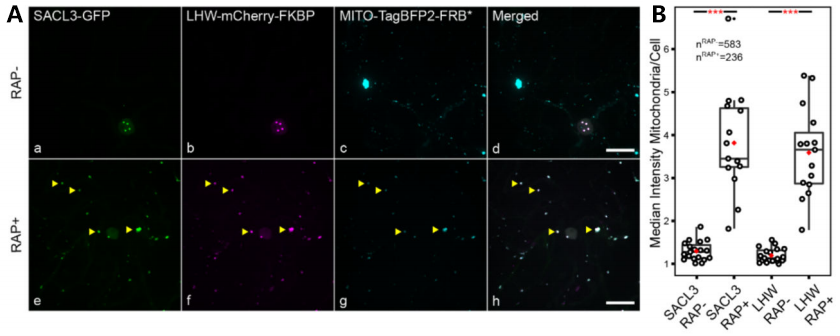
确定KSP检测系统的可行性之后,作者利用MITO-TagBFP2-FRB*(定位在线粒体上)在KSP检测系统中测试了SACL和LHW的相互作用。在没有添加雷帕霉素的条件下,SACL-GFP和LHW-mCherry-FKBP定位在细胞核上,使用雷帕霉素处理后,这两个融合蛋白离域到了线粒体,从而证实了这两个蛋白在KSP检测系统中的相互作用,这一结果也进一步验证了通过其它蛋白互作技术,如:免疫沉淀-串联质谱、酵母双杂得到的互作结果(图15)。
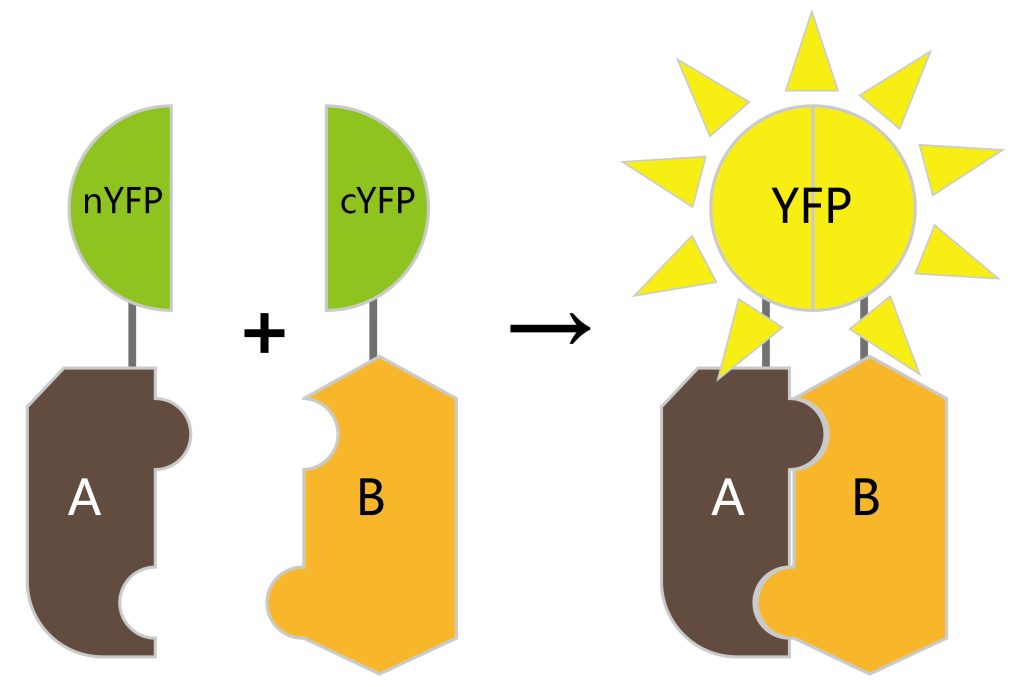
(1)BiFC技术不仅可以直观、快速判断目标蛋白在活细胞中的定位和相互作用,还能用于体外的蛋白质相互作用。该技术对仪器的要求没那么高,且背景干净。但是它对温度敏感,无法实时观察分子间的相互作用,容易光漂白,且存在假阳性和假阴性问题,需要在实验中反复验证。
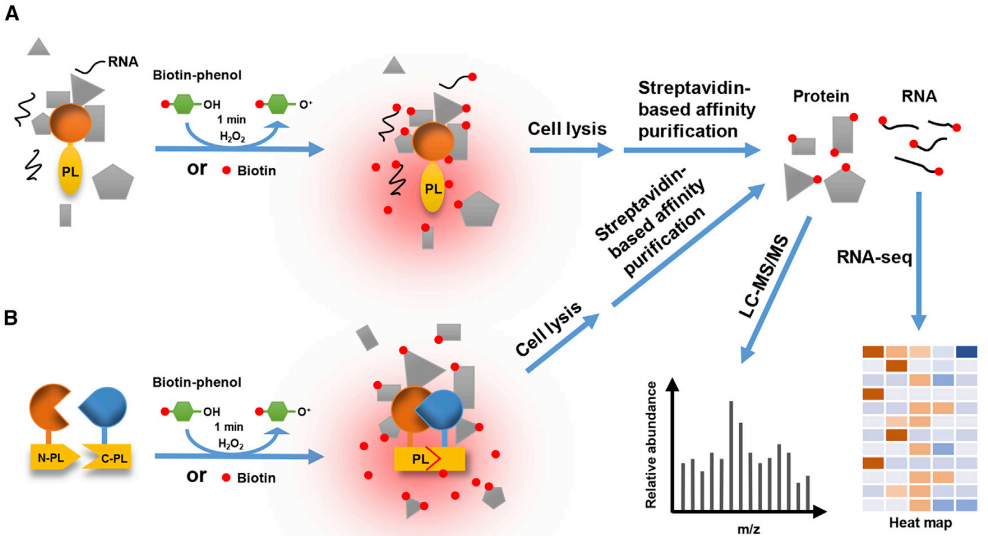
(4)邻近标记技术不仅无需构建文库或使用抗体,还对疏水性和低丰度蛋白的互作蛋白鉴定以及弱/瞬时互作蛋白的分析具有独特的优势。但是邻近标记使用的酶大多数最适温度为37℃,而这一温度不利于大部分植物的生长,另外,生物素连接酶蛋白表达水平、外源生物素浓度以及孵育时间都需要优化。
(5)FRET-FLIM技术具备高时空分辨率,能够实时监测植物活细胞中蛋白质的动态,分析程序简单快速,结果不需要光谱矫正。但是需要目标蛋白高表达,另外激发光源可能会引起光漂白,该技术的成本相对比较高,当供体蛋白与受体蛋白空间结构较大时,容易导致FRET发生效率低,易出现假阳性。
(6)Split-HaloTag技术因为TMR染料的稳定性,可以用于长时间扫描观察,并且Split-HaloTag片段不具有内在亲合性,降低了假阳性。但是该技术的实验方案和操作相对复杂,对实验者的操作要求高。
(7)KSP检测系统可以在极短的时间内观察到结果,且结果并不依赖荧光基团之间的互补或基团之间的空间距离,但是对于一些移动性强或具有多种亚细胞定位的蛋白来说,使用该技术效果不佳。
References:
Cui Y, Zhang X, Yu M, et al. Techniques for detecting protein-protein interactions in living cells: principles, limitations, and recent progress. Sci China Life Sci. 2019, 62(5): 619-632.
Förster T. Energy migration and fluorescence. 1946. J Biomed Opt. 2012, 17(1): 011002.
Gao M, He Y, Yin X, et al. Ca2+ sensor-mediated ROS scavenging suppresses rice immunity and is exploited by a fungal effector. Cell. 2021, 84(21): 5391-5404.
Ishikawa H, Meng F, Kondo N, et al. Generation of a dual-functional split-reporter protein for monitoring membrane fusion using self-associating split GFP. Protein Eng Des Sel. 2012, 25(12): 813-20.
Lang C, Schulze J, Mendel RR, et al. HaloTag: a new versatile reporter gene system in plant cells. J Exp Bot. 2006, 57(12): 2985-92.
Liu Y, Liu Q, Li X, et al. MdERF114 enhances the resistance of apple roots to Fusarium solani by regulating the transcription of MdPRX63. Plant Physiol. 2023, 192(3): 2015-2029.
Margineanu A, Chan JJ, Kelly DJ, et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Sci Rep. 2016, 6: 28186.
Minner-Meinen R, Weber JN, Albrecht A, et al. Split-HaloTag imaging assay for sophisticated microscopy of protein-protein interactions in planta. Plant Commun. 2021, 2(5): 100212.
Robinson MS, Sahlender DA, Foster SD. Rapid inactivation of proteins by rapamycin-induced rerouting to mitochondria. Dev Cell. 2010, 18(2): 324-31.
Shang S, Liu G, Zhang S, et al. A fungal CFEM-containing effector targets NPR1 regulator NIMIN2 to suppress plant immunity. Plant Biotechnol J. 2023.
Winkler J, Mylle E, De Meyer A, et al. Visualizing protein-protein interactions in plants by rapamycin-dependent delocalization. Plant Cell. 2021, 33(4): 1101-1117.
Xie Y, Wang Y, Yu X, et al. SH3P2, an SH3 domain-containing protein that interacts with both Pib and AvrPib, suppresses effector-triggered, Pib-mediated immunity in rice. Mol Plant. 2022, 15(12): 1931-1946.
Yang X., Wen Z., Zhang D., et al. Proximity labeling: an emerging tool for probing in planta molecular interactions. Plant Commun. 2020, 2(2): 100137.
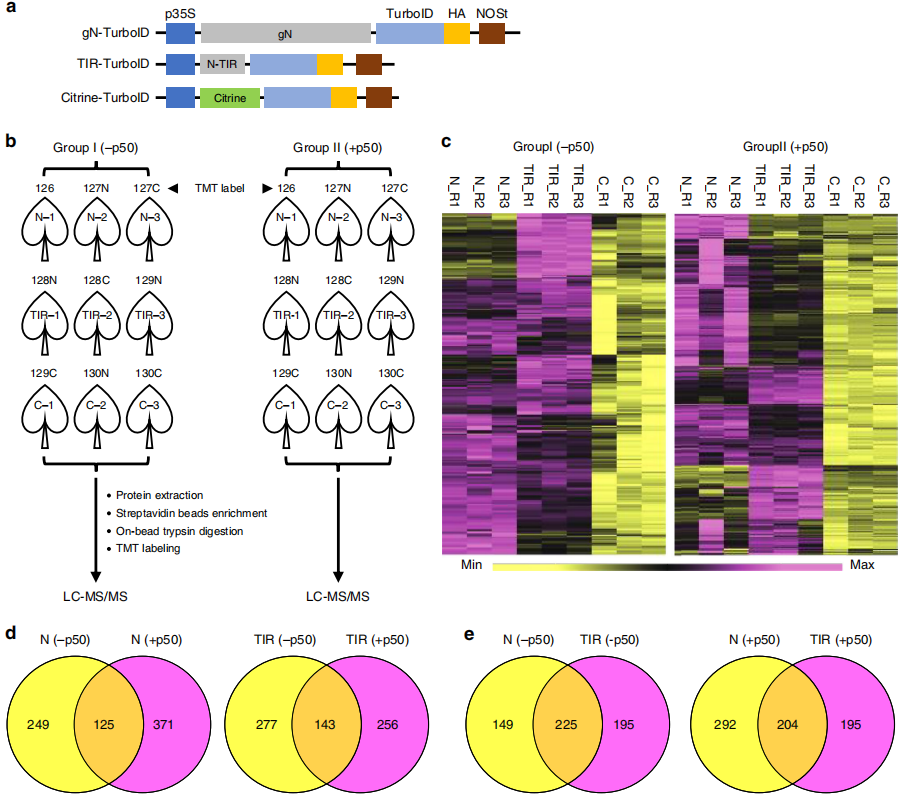
Zhang Y., Song G., Lal N.K., et al. TurboID-based proximity labeling reveals that UBR7 is a regulator of N NLR immune receptor-mediated immunity. Nat Commun. 2019, 10(1): 3252.
苏田, 韩笑, 刘华东. 邻近标记在蛋白质组学中的发展及应用. 中国生物化学与分子生物学报, 2020, 36(01): 36-41.