本文内容速览:
生长素参与植物细胞的伸长和分裂、主侧根和下胚轴的生长、植株向地性和向光性的形成以及根毛和花器官的形成等生命过程,对植物早期生长发育和形态建成均具重要意义。生长素不仅在植物的生长发育和环境适应中发挥作用,而且还是协调不同组织甚至不同细胞之间交流的重要信号分子。
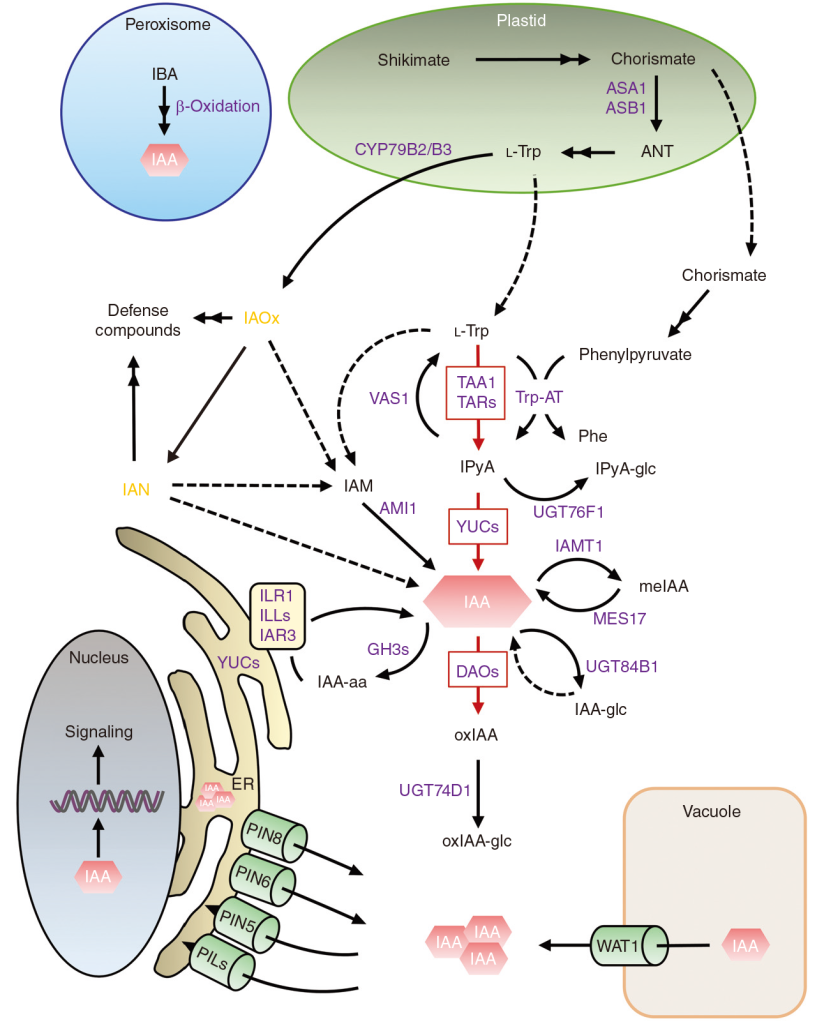
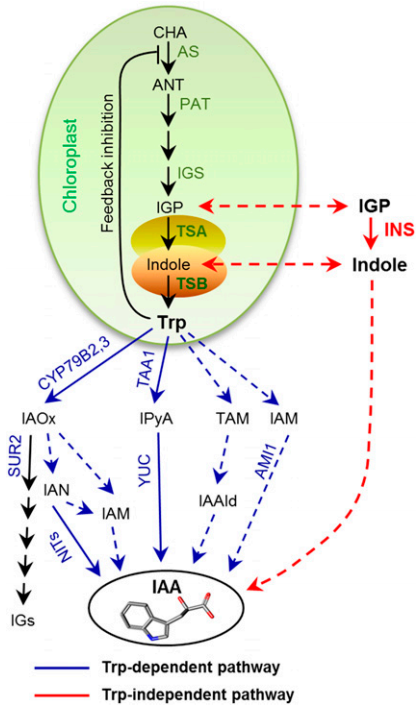
目前普遍认为IAOx途径不是生长素合成的主流途径,主要有以下几个观点:(1)在水稻和玉米等植物中并没有发现CYP79B的同源基因;(2)cy79b2 cy79b3双突变体生长素缺陷的表型相比于其它信号转导途径成员并不明显。
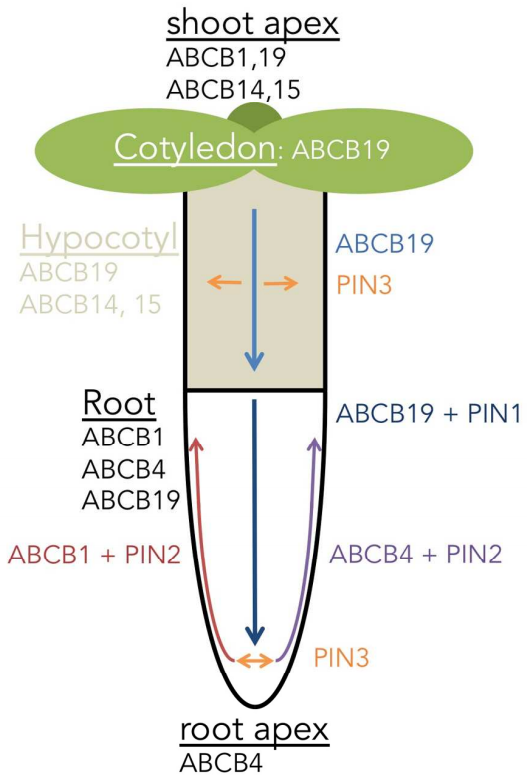
生长素极性运输由3类转运蛋白介导完成,包括生长素输入载体Aux1/LAX、ABCB 转运蛋白和生长素输出载体PIN。局部生长素的新陈代谢导致其含量与浓度升高,引起具有显著方向性的细胞—细胞间的运输,植物体中的生长素输入载体与输出载体通过协调作用从而实现生长素的跨膜运输。其中,PIN 蛋白的质膜极性分布模式在调节生长素不对称分布方面起着重要作用,是决定生长素极性运输方向的关键调控因子(Park et al., 2017)。
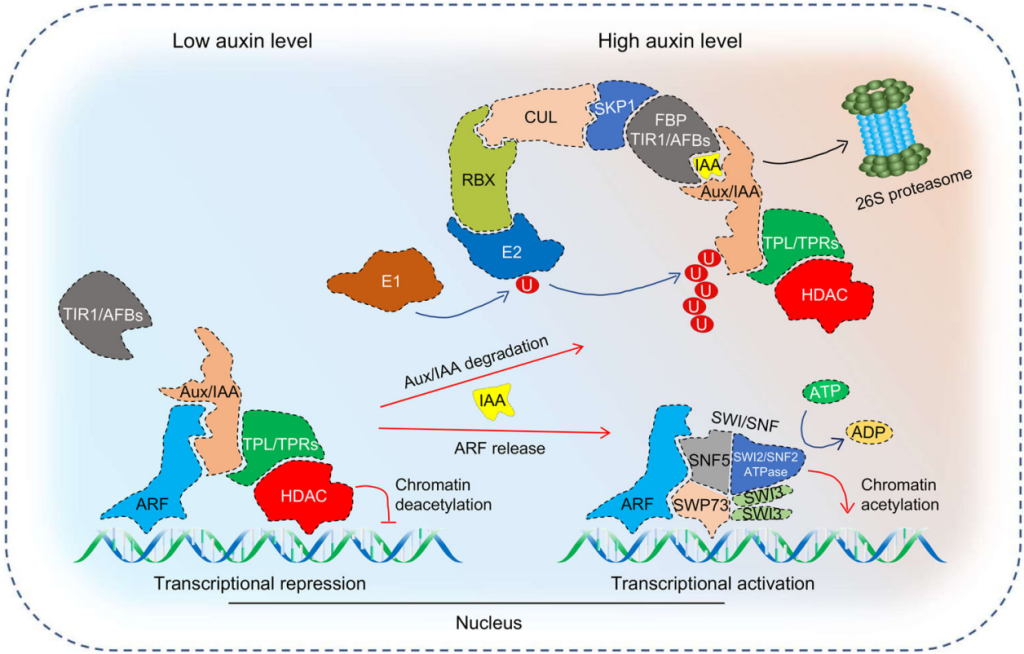
图4. 经典的生长素信号转导途径(Yu et al., 2022)。
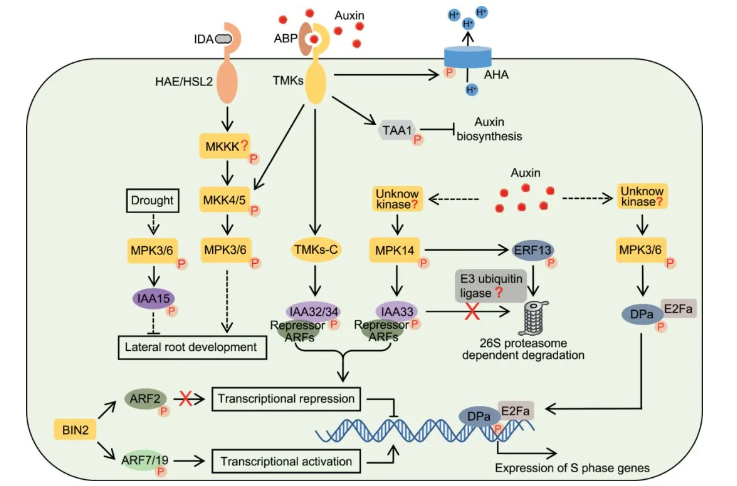
在Aux/IAAs家族中存在部分蛋白由于缺失结构域而无法被TIR1/AFBs识别,因此它们不能参与经典的生长素信号转导途径。例如TMK1-IAA32/34-ARFs途径和MPK14-IAA33-ARFs途径等,它们主要由蛋白质磷酸化来介导。在TMK1-IAA32/34-ARFs途径中,高浓度生长素诱导质膜定位的跨膜激酶(transmembrane kinase 1,TMK1)羧基端(C-)发生剪切,导致TMK1的C末端转移到细胞核中,非经典Aux/IAAs家族成员IAA32/34被TMK1的C末端特异性磷酸化,磷酸化的IAA32/34与ARFs互作,参与调控下游基因表达;在MPK14-IAA33-ARFs途径中,高浓度生长素激活丝裂原活化蛋白激酶14(mitogen-activated protein kinase14,MPK14)磷酸化并稳定非经典IAA33,提高了IAA33与经典IAA5竞争性结合下游抑制因子ARF10/ARF16的能力(Cui et al., 2023)。
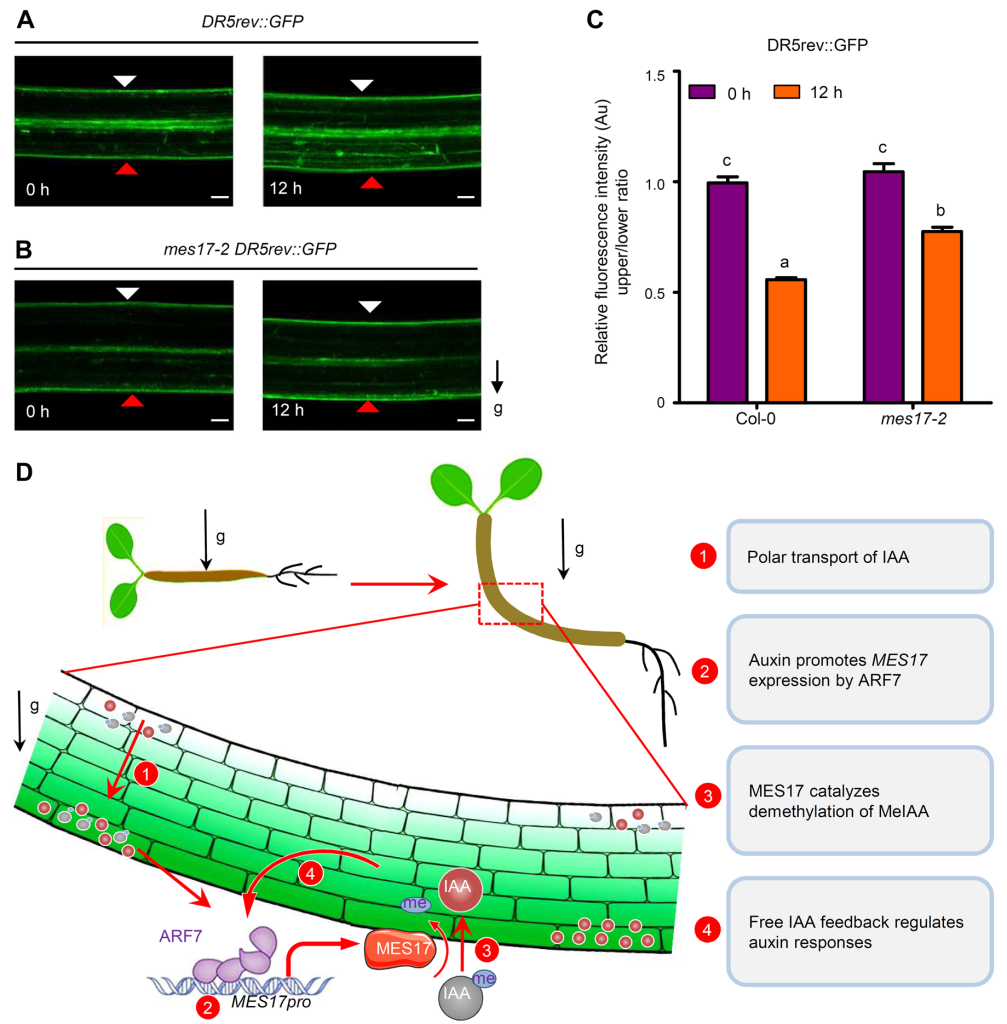
图6. MES17对生长素信号的反馈调控(Zhang et al., 2022)。(A-B)重力刺激前(0h)和重力刺激后(12h),Col-0(A)和 mes17-2(B)幼苗下胚轴中的 DR5rev:GFP 报告者的荧光模式。白色三角形表示下胚轴上部,红色三角形表示下部。g 代表重力,箭头表示重力方向。(C)、(A)和(B)中所示 Col-0 和 mes17-2 下胚轴在重力刺激 12 小时前后的相对绿色荧光蛋白(GFP)信号强度。(D)MES17 在下胚轴重力定向过程中的作用模型。
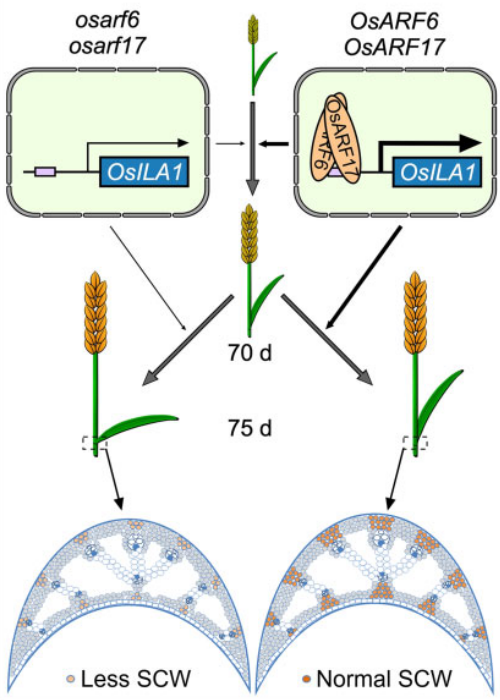
2021年,上海交通大学生命科学技术学院张大兵课题组在The Plant Cell上发表了题为“AUXIN RESPONSE FACTOR 6 and 7 control the flag leaf angle in rice by regulating secondary cell wall biosynthesis of lamina joints”的研究论文。作者通过对osarf6和osarf17突变体进行观察,发现突变体叶片关节细胞次生细胞壁水平降低,导致旗叶角度夸大。进一步研究发现生长素响应因子OsARF6和OsARF17通过独立或协同的方式激活水稻旗叶夹角调控基因ILA1的表达参与水稻旗叶角度调控(Huang et al., 2021)。
图7. OsARF6和OsARF17调控水稻旗叶角度的模型(Huang et al., 2021)。
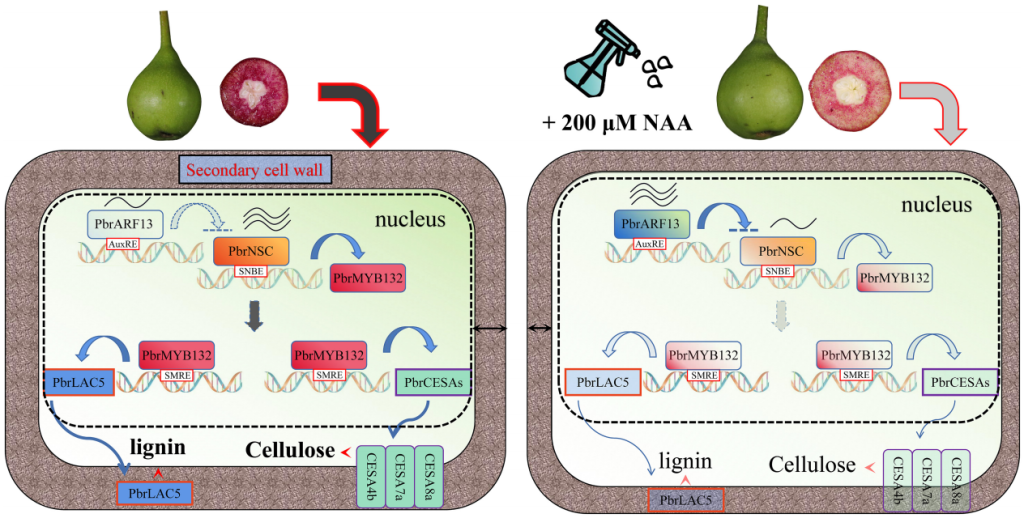
2023年,南京农业大学园艺学院吴俊老师课题组在Plant Biotechnology Journal上发表了题为“Auxin inhibits lignin and cellulose synthesis in stone cells of pear fruit via the PbrARF13-PbrNSC-PbrMYB132 transcriptional regulatory cascade”的研究论文。该研究发现外源喷施NAA能够抑制梨果实中石细胞木质素和纤维素合成关键基因PbrNSC 的表达,此外PbrNSC的表达还受到生长素反应因子PbrARF13的抑制,PbrNSC还能抑制PbrMYB132的表达。基于此,作者最终阐述了‘PbrARF13-PbrNSC-PbrMYB132’ 级联信号途径在NAA抑制梨果实石细胞形成的过程中发挥着重要的作用(Xu et al., 2023)。
References:
Casanova-Sáez R, Mateo-Bonmatí E, Ljung K. Auxin Metabolism in Plants. Cold Spring Harb Perspect Biol. 2021;13(3):a039867.
Hull AK, Vij R, Celenza JL. Arabidopsis cytochrome P450s that catalyze the first step of tryptophan-dependent indole-3-acetic acid biosynthesis. Proc Natl Acad Sci U S A. 2000;97(5):2379-2384.
Sugawara S, Hishiyama S, Jikumaru Y, et al. Biochemical analyses of indole-3-acetaldoxime-dependent auxin biosynthesis in Arabidopsis. Proc Natl Acad Sci U S A. 2009;106(13):5430-5435.
Patten CL, Blakney AJ, Coulson TJ. Activity, distribution and function of indole-3-acetic acid biosynthetic pathways in bacteria. Crit Rev Microbiol. 2013;39(4):395-415.
Tao Y, Ferrer JL, Ljung K, et al. Rapid synthesis of auxin via a new tryptophan-dependent pathway is required for shade avoidance in plants. Cell. 2008;133(1):164-176.
Woodward AW, Bartel B. Auxin: regulation, action, and interaction. Ann Bot. 2005;95(5):707-735.
Mashiguchi K, Tanaka K, Sakai T, et al. The main auxin biosynthesis pathway in Arabidopsis. Proc Natl Acad Sci U S A. 2011;108(45):18512-18517.
Wang B, Chu J, Yu T, et al. Tryptophan-independent auxin biosynthesis contributes to early embryogenesis in Arabidopsis. Proc Natl Acad Sci U S A. 2015;112(15):4821-4826.
Yu Z, Zhang F, Friml J, et al. Auxin signaling: Research advances over the past 30 years. J Integr Plant Biol. 2022;64(2):371-392.
Cui X, Wang J, Li K, et al. Protein post-translational modifications in auxin signaling. J Genet Genomics. 2023;S1673-8527(23)00154-6.
Park J, Lee Y, Martinoia E, et al. Plant hormone transporters: what we know and what we would like to know. BMC Biol. 2017;15(1):93.
Geisler M, Aryal B, di Donato M, et al. A Critical View on ABC Transporters and Their Interacting Partners in Auxin Transport. Plant Cell Physiol. 2017;58(10):1601-1614.
Zhang F, Li C, Qu X, et al. A feedback regulation between ARF7-mediated auxin signaling and auxin homeostasis involving MES17 affects plant gravitropism. J Integr Plant Biol. 2022;64(7):1339-1351.
Huang G, Hu H, van de Meene A, et al. AUXIN RESPONSE FACTORS 6 and 17 control the flag leaf angle in rice by regulating secondary cell wall biosynthesis of lamina joints. Plant Cell. 2021;33(9):3120-3133.
Xu S, Sun M, Yao JL, et al. Auxin inhibits lignin and cellulose biosynthesis in stone cells of pear fruit via the PbrARF13-PbrNSC-PbrMYB132 transcriptional regulatory cascade. Plant Biotechnol J. 2023;21(7):1408-1425.
Wang Q, Huang D, Tu W, et al. Overexpression of auxin/indole-3-acetic acid gene MdIAA24 enhances Glomerella leaf spot resistance in apple (Malus domestica). Horticultural Plant Journal, 2022.
Xie Y, Lv Y, Jia L, et al. Plastid-localized amino acid metabolism coordinates rice ammonium tolerance and nitrogen use efficiency. Nat Plants. 2023;9(9):1514-1529.
Li HL, Liu ZY, Wang XN, et al. E3 ubiquitin ligases SINA4 and SINA11 regulate anthocyanin biosynthesis by targeting the IAA29-ARF5-1-ERF3 module in apple. Plant Cell Environ. 2023;10.1111.
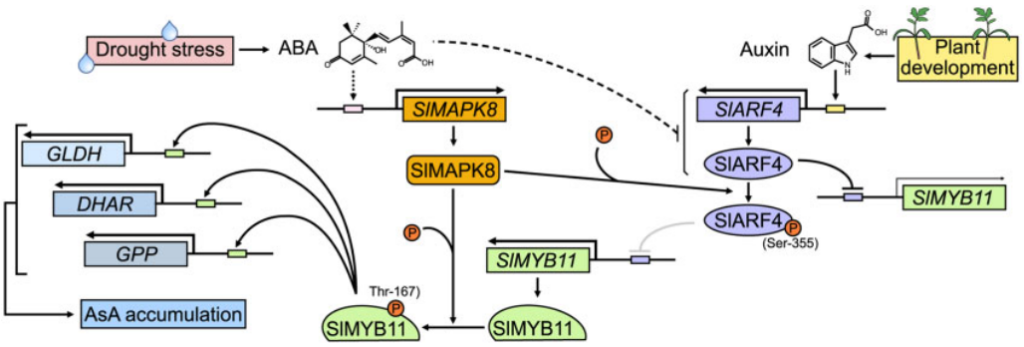
Xu X, Zhang Q, Gao X, et al. Auxin and abscisic acid antagonistically regulate ascorbic acid production via the SlMAPK8-SlARF4-SlMYB11 module in tomato. Plant Cell. 2022;34(11):4409-4427.
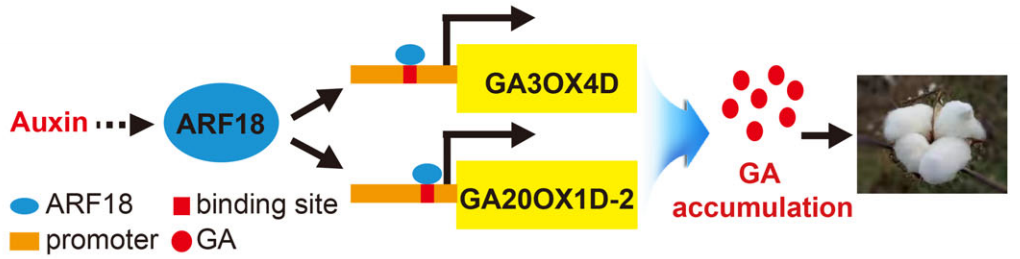
Zhu L, Jiang B, Zhu J, et al. Auxin promotes fiber elongation by enhancing gibberellic acid biosynthesis in cotton. Plant Biotechnol J. 2022;20(3):423-425.
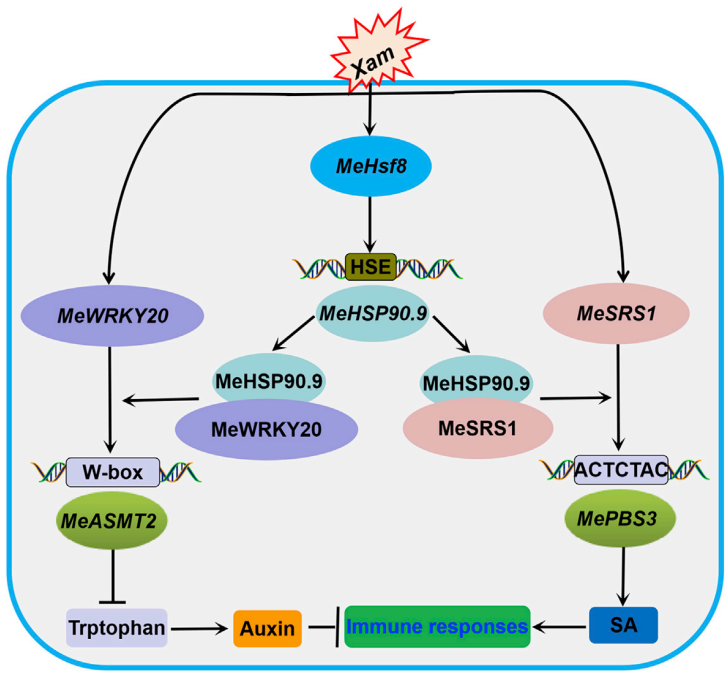
Wei Y, Zhu B, Liu W, et al. Heat shock protein 90 co-chaperone modules fine-tune the antagonistic interaction between salicylic acid and auxin biosynthesis in cassava. Cell Rep. 2021;34(5):108717.