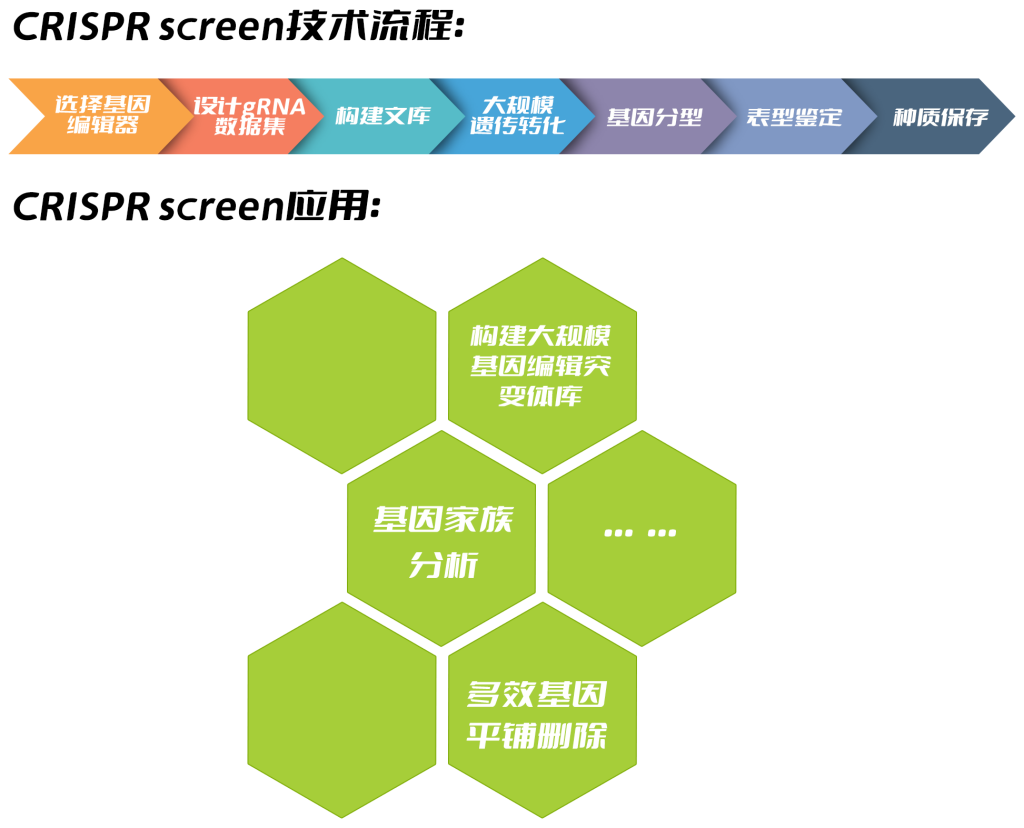
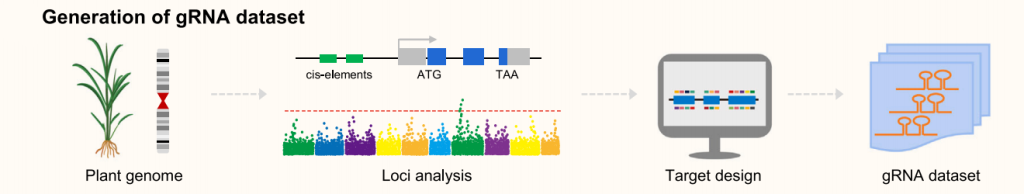
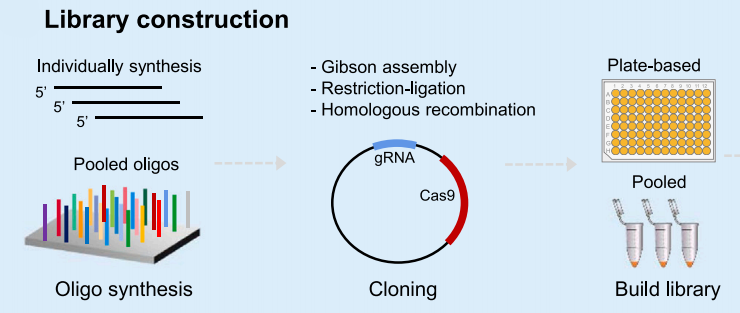
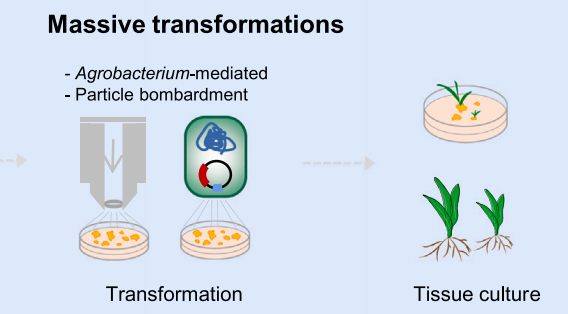
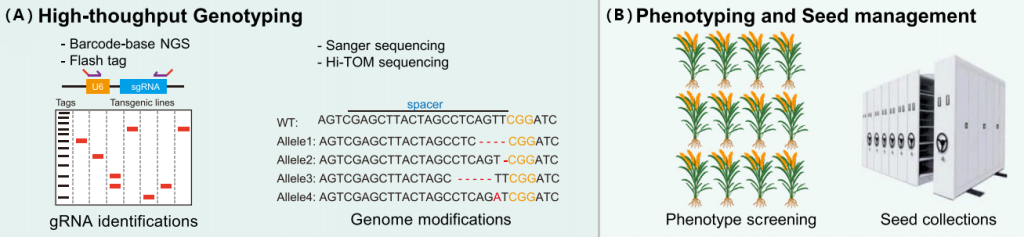
本次推文内容速览:
作为一种强大的基因编辑工具,CRISPR/Cas正在彻底改变基础研究和作物育种,目前已发展成为高效、简单和可编程的大规模编辑(CRISPR screen)技术。CRISPR screen又可以称为全基因组基因编辑或者高通量基因编辑技术,目前在哺乳动物研究领域特别流行,且成效显著。随着该技术的不断发展,CRISPR screen也被报道用于作物,包括水稻、番茄、大豆和玉米等(Jacobs et al., 2017;Lu et al., 2017;Meng et al., 2017;Bai et al., 2020)。各种作物大规模编辑的广泛运用很可能会导致未来几年基因编辑种质的激增。此外,高质量、高覆盖率和均匀分布的编辑突变体文库的开发将为功能基因组学研究和作物改良提供宝贵的遗传资源。
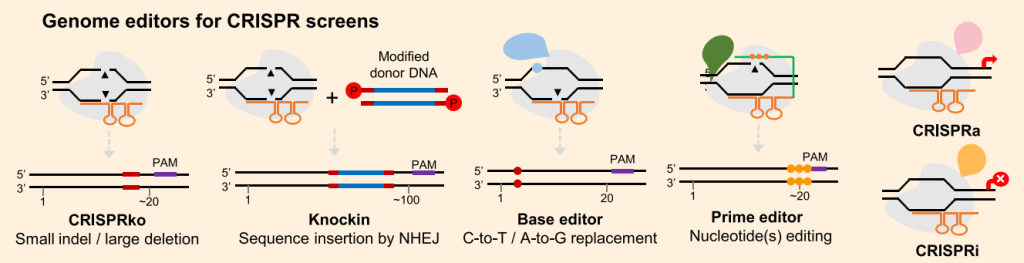
尽管SpCas9介导的基因敲除是目前流行的CRISPR筛选工具,但也存在其它值得推荐的基因编辑工具。例如:1)具有更多可识别PAM的SpCas9变体Cas9 NG(将PAM识别序列由NGG简化为NG,还能识别NAC、NTG、NT和NCG等PAM序列)和SpRY(几乎不受PAM序列的限制,但它对NNR和NRN PAM具有较大的偏好性,N=任意碱基,R=A/G)(Tang et al., 2017;Manghwar et al., 2019);2)识别富含AT PAM的Cpf1,因为Cpf1/Cas12a的切割位点位于其PAM的远端,所以它通常产生大的缺失(例如20bp)。SpCas9与某些酶(如APOBEC3A和UNG)的融合也可以诱导类似的缺失,这些工具特别适合于筛选启动子内的顺式调节元件(Wang et al., 2020;Tian et al., 2022)。
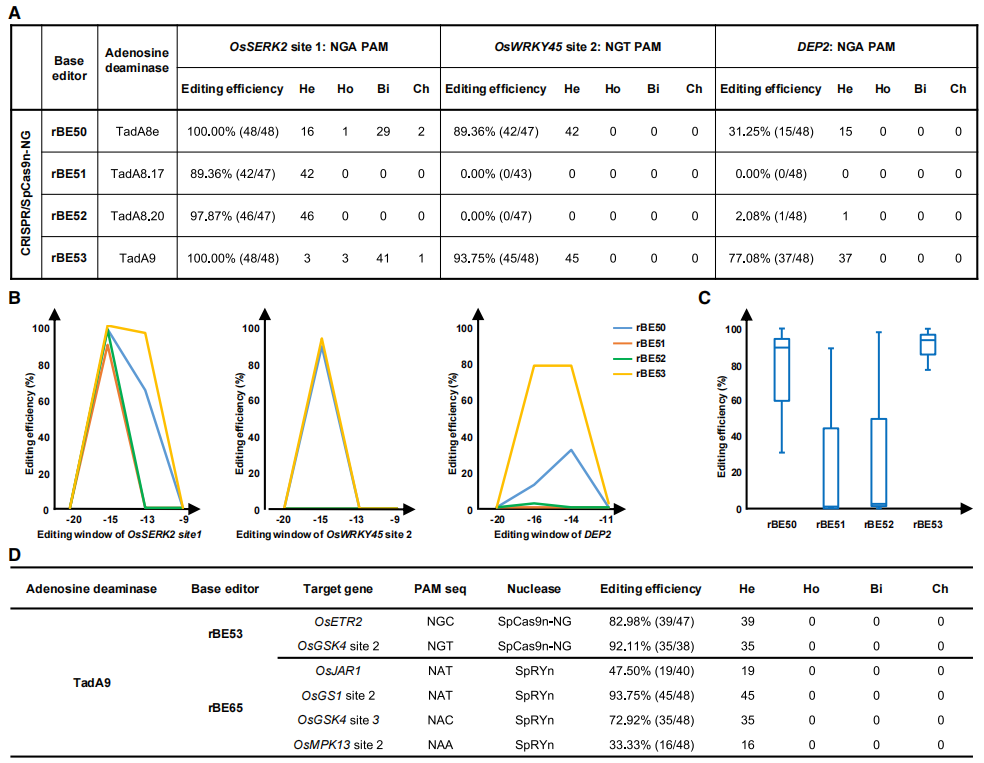
除了ABE和CBE单碱基编辑外,中科院上海分子植物卓越中心逆境中心朱健康研究组与陆钰明研究组共同优化得到的CGBE碱基编辑器,它可以在水稻中实现高效的C到G的颠换编辑。中国科学院遗传与发育生物学研究所高彩霞团队和李家洋团队合作开发了一种内源基因突变碱基编辑器STEME,它将胞嘧啶脱氨酶APOBEC3A和腺嘌呤脱氨酶ecTadA-ecTadA7.10同时融合在nCas9的N端,一起实现C到T和A到G的转换,这项技术目前主要用于植物基因的定向进化。随着这些碱基编辑器的进一步的改造,或许将来可以将它们应用扩展到CRISPR screen技术中。
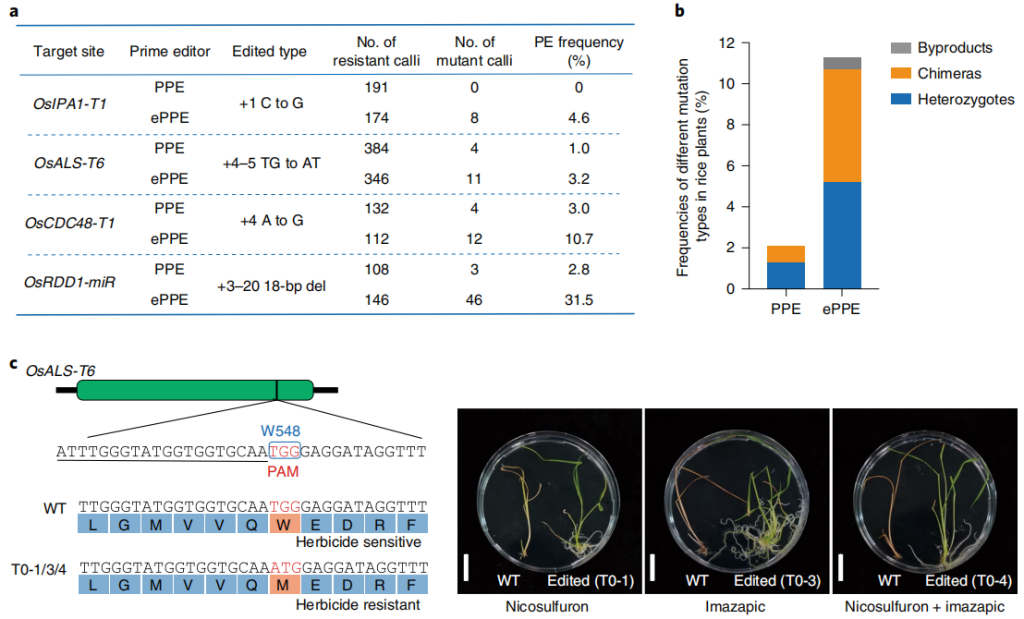
图5 PPE和ePPE在植物中产生突变(Zong et al., 2022)。(a)PPE和ePPE在四个目标位点诱导抗性水稻愈伤组织编辑效率的比较;(b)PPE和ePPE在OsALS-T6位点上诱导的T0突变体的不同突变类型的效率;(c)OsALS-T6位点发生Trp548Met突变会赋予水稻除草剂抗性。将WT OsALS-T6与T0-1、T0-3和T0-4突变体中的序列进行比较。PAM以红色突出显示,sgRNA以下划线突出显示。野生型(左)和杂合OsALS W548M突变体(右)的表型。
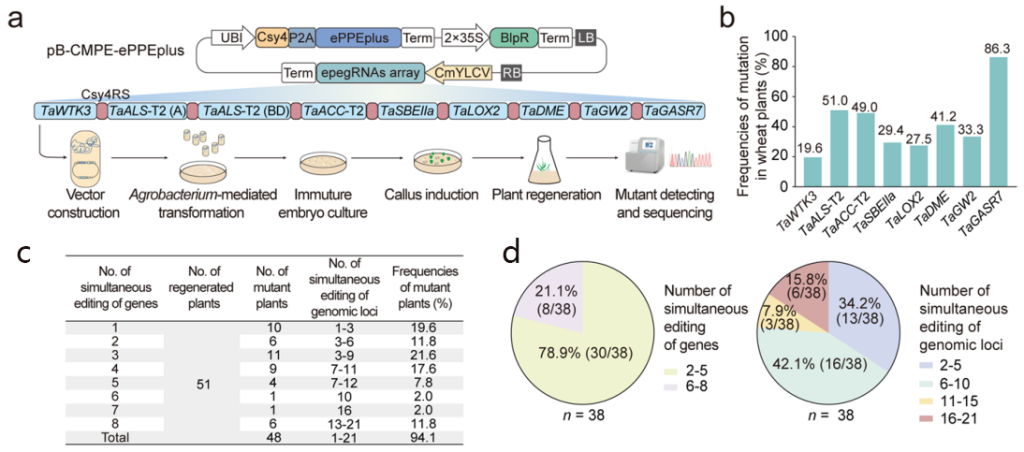
2023年6月29日,中国农业大学小麦研究中心宗媛团队在Genome Biology上发表了题为“Efficient and versatile multiplex prime editing in hexaploid wheat”的研究论文。研究者多角度对植物Prime editor系统进行了全方位的优化,在六倍体小麦中开发了一种升级版先导编辑器ePPEplus,在逆转录酶中引入新的氨基酸突变位点V223A。ePPEplus与PPE和ePPE相比,效率分别平均提高了33.0倍和6.4倍。重要的是,建立了一个强大的多重引物编辑平台,在小麦中实现了2~8个基因的同时精准编辑(最高可同时编辑21个基因组的位点(Genomic loci)),总编辑效率高达74.5%(图6a和b)。值得注意的是,51株T0再生植株中有6株(11.8%)发生了8个基因的同时编辑,实现了小麦植株中不同有利等位基因的聚合(图6c和d)(Ni et al., 2023)。这拓宽了引导编辑系统的应用范畴,为作物优势性状叠加提供新的技术支持。
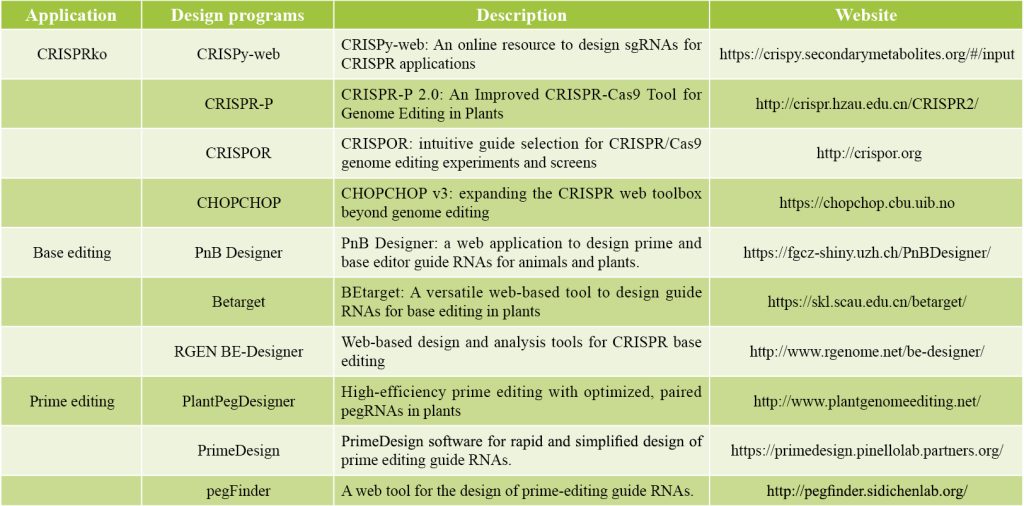
表1 gRNA设计网站(Liu et al., 2023)。
在哺乳动物中,混合转染策略的关键点是必须保证一个细胞接收一个gRNA,否则多个sgRNA的存在将导致多重编辑。农杆菌和慢病毒的作用相似,通常会将多个外源DNA整合到宿主基因组中。目前,这些问题已经在哺乳动物中通过控制转染的多重性(MOI,每个细胞递送的病毒粒子的数量)得到了解决。通常使用相对较低的MOI(0.3-0.5)来确保一个细胞主要只接收一个病毒粒子(Shalem et al., 2015)。在使用低“MOI”的农杆菌的水稻愈伤组织的混合转化中也实现了这种“一对一”的效果(Lu et al., 2017)。然而,最佳农杆菌浓度在不同植物物种(如番茄和拟南芥)之间可能有很大差异,因此需要在进行大规模混合遗传转化之前确定。
与哺乳动物中直接对细胞进行检测不同,植物中的CRISPR screen通常需要通过组织培养来获得编辑的植株集合,再进行检测。因此,在植物中进行CRISPR screen要困难得多。
早在2017年,中科院遗传发育所李家洋及高彩霞研究团队和中科院上海分子植物卓越中心陆钰明以背靠背的形式分别在Molecular Plant发表了以利用CRISPR/Cas9方法创制水稻突变体库的研究论文(Lu et al., 2017;Meng et al., 2017)。
2021年10月,华中农业大学谢卡斌课题组在Molecular Plant期刊上发表了题为“A FLASH pipeline for arrayed CRISPR library construction and the gene function discovery of rice receptor-like kinases”的研究论文。该研究设计了一种以PCR片段长度作为Cas9/gRNA载体标签的策略(PCR fragment-length markers for gRNAs distinguishing,简称FLASH标签),它可以通过常规PCR和凝胶电泳读取每个载体和转化植株的gRNA信息(Chen et al., 2020)。利用此方法,该课题组创建了靶向敲除1072个水稻类受体激酶的基因编辑材料,为快速鉴定抗病、抗逆相关的基因提供了新资源。
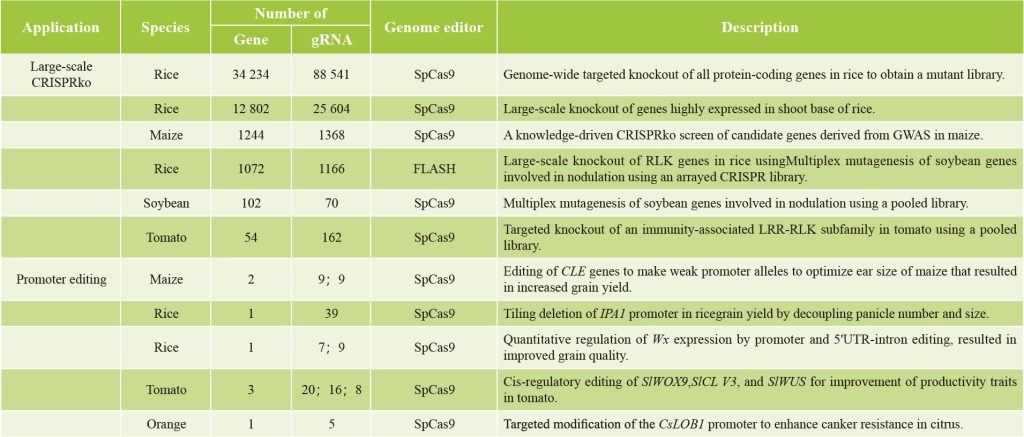
表2 植物CRISPR screen汇总(Liu et al., 2023)。
Meng X, Yu H, Zhang Y, et al. Construction of a genome-wide mutant library in rice using CRISPR/Cas9[J]. Molecular plant, 2017, 10(9): 1238-1241.
Jacobs T B, Zhang N, Patel D, et al. Generation of a collection of mutant tomato lines using pooled CRISPR libraries[J]. Plant physiology, 2017, 174(4): 2023-2037.
Bai M, Yuan J, Kuang H, et al. Generation of a multiplex mutagenesis population via pooled CRISPR‐Cas9 in soya bean[J]. Plant Biotechnology Journal, 2020, 18(3): 721-731.
Lu Y, Ye X, Guo R, et al. Genome-wide targeted mutagenesis in rice using the CRISPR/Cas9 system[J]. Molecular plant, 2017, 10(9): 1242-1245.
Liu H J, Jian L, Xu J, et al. High-throughput CRISPR/Cas9 mutagenesis streamlines trait gene identification in maize[J]. The Plant Cell, 2020, 32(5): 1397-1413.
Manghwar H, Lindsey K, Zhang X, et al. CRISPR/Cas system: recent advances and future prospects for genome editing[J]. Trends in plant science, 2019, 24(12): 1102-1125.
Tang X, Lowder L G, Zhang T, et al. A CRISPR–Cpf1 system for efficient genome editing and transcriptional repression in plants[J]. Nature plants, 2017, 3(3): 1-5.
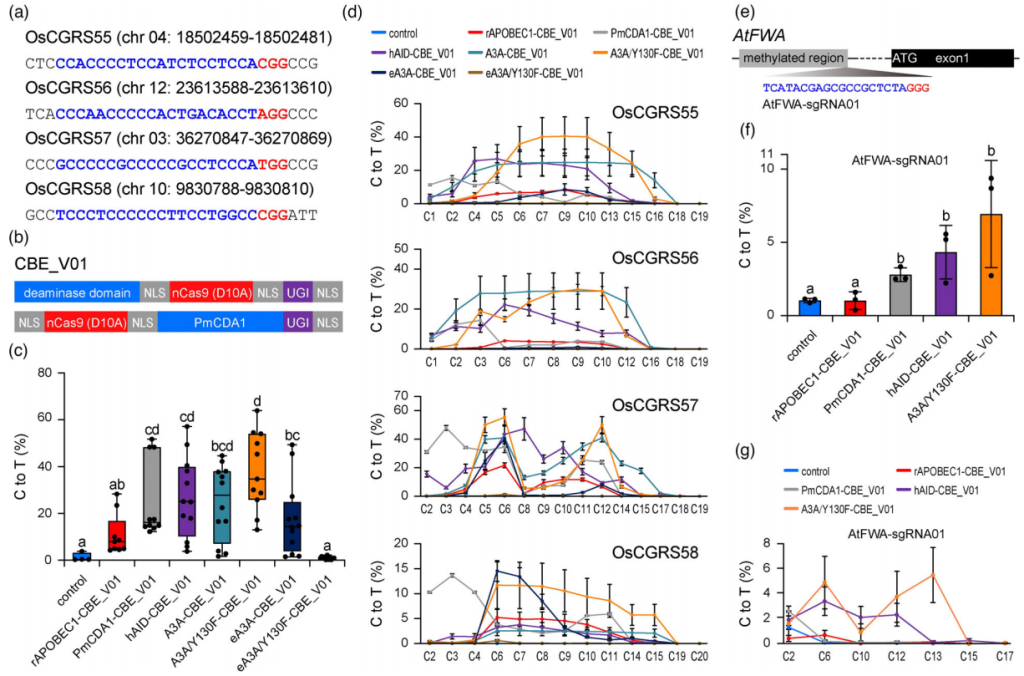
Tian Y, Shen R, Li Z, et al. Efficient C‐to‐G editing in rice using an optimized base editor[J]. Plant Biotechnology Journal, 2022, 20(7): 1238.
Wang S, Zong Y, Lin Q, et al. Precise, predictable multi-nucleotide deletions in rice and wheat using APOBEC–Cas9[J]. Nature biotechnology, 2020, 38(12): 1460-1465.
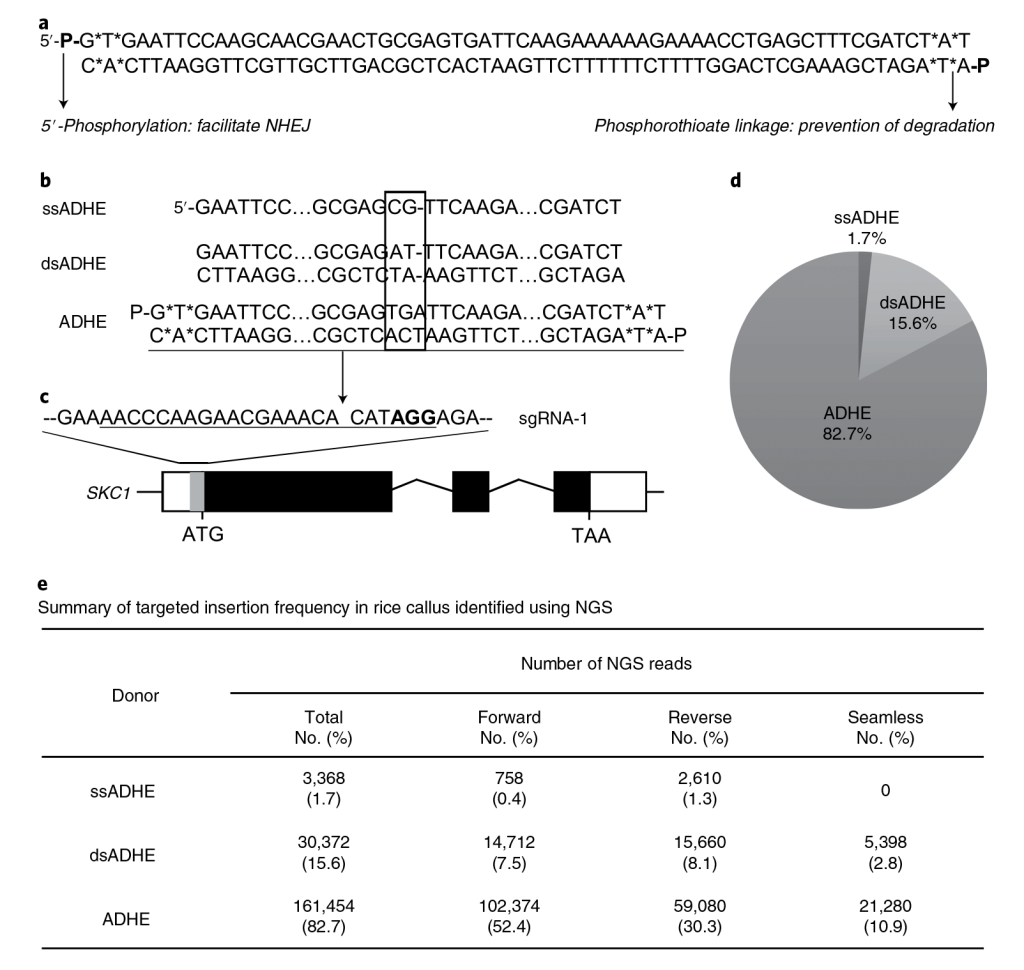
Lu Y, Tian Y, Shen R, et al. Targeted, efficient sequence insertion and replacement in rice[J]. Nature Biotechnology, 2020, 38(12): 1402-1407.
Lu Y, Ronald P C, Han B, et al. Rice protein tagging project: A call for international collaborations on genome-wide in-locus tagging of rice proteins[J]. Molecular Plant, 2020, 13(12): 1663-1665.
Komor A C, Kim Y B, Packer M S, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424.
Ren Q, Sretenovic S, Liu G, et al. Improved plant cytosine base editors with high editing activity, purity, and specificity[J]. Plant Biotechnology Journal, 2021, 19(10): 2052-2068.
Yan D, Ren B, Liu L, et al. High-efficiency and multiplex adenine base editing in plants using new TadA variants[J]. Molecular Plant, 2021, 14(5): 722-731.
Zong Y, Liu Y, Xue C, et al. An engineered prime editor with enhanced editing efficiency in plants[J]. Nature Biotechnology, 2022, 40(9): 1394-1402.
Ni P, Zhao Y, Zhou X, et al. Efficient and versatile multiplex prime editing in hexaploid wheat[J]. Genome Biology, 2023, 24(1): 1-19.
Tang X, Lowder L G, Zhang T, et al. A CRISPR–Cpf1 system for efficient genome editing and transcriptional repression in plants[J]. Nature plants, 2017, 3(3): 1-5.
Pan C, Wu X, Markel K, et al. CRISPR–Act3. 0 for highly efficient multiplexed gene activation in plants[J]. Nature plants, 2021, 7(7): 942-953.
Zhang R X, Li B B, Yang Z G, et al. Dissecting plant gene functions using CRISPR toolsets for crop improvement[J]. Journal of Agricultural and Food Chemistry, 2022, 70(24): 7343-7359.
Blin K, Pedersen L E, Weber T, et al. CRISPy-web: An online resource to design sgRNAs for CRISPR applications[J]. Synthetic and Systems Biotechnology, 2016, 1(2): 118-121.
Heigwer F, Zhan T, Breinig M, et al. CRISPR library designer (CLD): software for multispecies design of single guide RNA libraries[J]. Genome biology, 2016, 17(1): 1-10.
Shalem O, Sanjana N E, Zhang F. High-throughput functional genomics using CRISPR–Cas9[J]. Nature Reviews Genetics, 2015, 16(5): 299-311.
Lu Y, Ye X, Guo R, et al. Genome-wide targeted mutagenesis in rice using the CRISPR/Cas9 system[J]. Molecular plant, 2017, 10(9): 1242-1245.
Liu T, Zhang X, Li K, et al. Large-scale genome editing in plants: Approaches, applications, and future perspectives[J]. Current Opinion in Biotechnology, 2023, 79: 102875.
Chen K, Ke R, Du M, et al. A FLASH pipeline for arrayed CRISPR library construction and the gene function discovery of rice receptor-like kinases[J]. Molecular plant, 2022, 15(2): 243-257.
Lu Y, Ye X, Guo R, et al. Genome-wide targeted mutagenesis in rice using the CRISPR/Cas9 system[J]. Molecular plant, 2017, 10(9): 1242-1245.
Meng X, Yu H, Zhang Y, et al. Construction of a genome-wide mutant library in rice using CRISPR/Cas9[J]. Molecular plant, 2017, 10(9): 1238-1241.