本次推文内容速览:
在众多的蛋白翻译后修饰(PTM)类型中,磷酸化修饰(Phosphorylation modification)的蛋白占到了所有蛋白质约三分之一的比例,是最普遍的修饰类型之一。蛋白质的磷酸化和去磷酸化作为一种“分子开关”(Olsen et al., 2006),通过影响蛋白质的结构、定位和功能,在细胞的信号转导、转录翻译、代谢和发育等生命过程中发挥着重要作用。总而言之,磷酸化作为生命活动最基础的修饰,蛋白的磷酸化研究一直受到研究者的广泛关注。因此,伯小远想为大家总结一下磷酸化在植物学研究中都发挥了哪些作用。
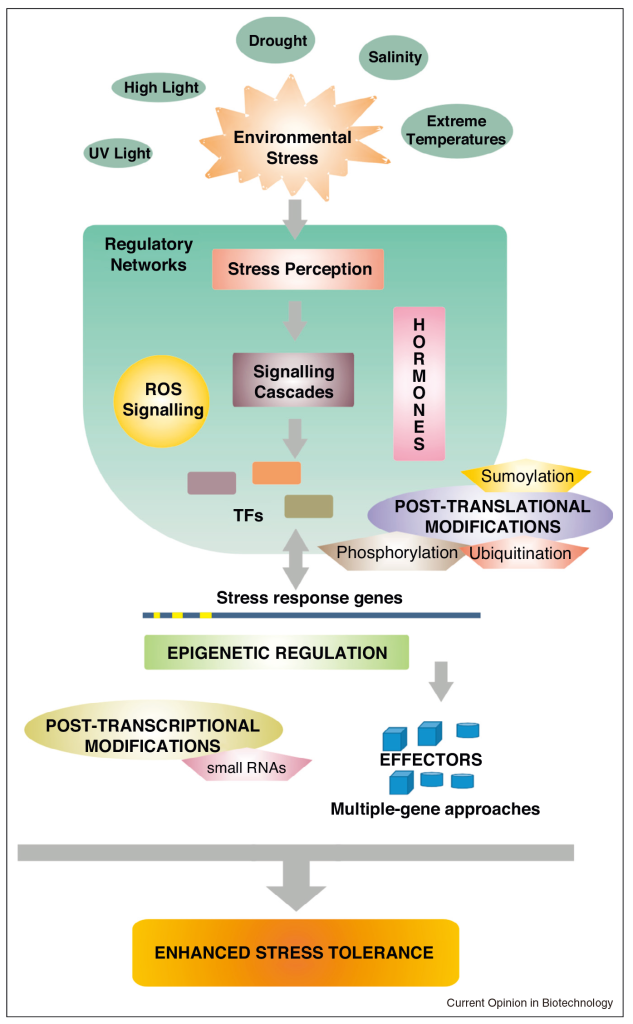
为了对抗环境胁迫的不利影响,植物进化出了适应机制或特定的生长习惯来避免胁迫(Dorothea Bartels and Ramanjulu Sunkar, 2005)。它们的反应是动态的,包括可逆和不可逆的变化、膜系统的调整、细胞壁结构的改变,以及细胞分裂的变化(Grant R. Cramer, 2010;Aleksandra Skirycz and Dirk Inze, 2010;Cramer et al., 2011)。此外,相容溶质(如脯氨酸或棉子糖)的产生有助于稳定蛋白质和细胞结构或维持细胞膨压,并降低过量的ROS水平(Dorothea Bartels and Ramanjulu Sunkar, 2005;Babu Valliyodan and Henry T Nguyen, 2006;Laszlo Szabados and Arnould Savoure, 2010;Julia Krasensky and Claudia Jonak, 2012)。非生物应激反应也受表观遗传(Khraiwesh et al., 2012;Yuan et al., 2013)和转录调控的影响,这些调控会改变大量基因的表达,包括参与渗透保护剂、解毒酶、转运蛋白和调节蛋白(如蛋白激酶、磷酸酶和转录因子)合成的基因(Julia Krasensky and Claudia Jonak, 2012;Chinnusamy et al., 2007;Kazuo Shinozaki and Kazuko Yamaguchi-Shinozaki, 2007)。
植物应对非生物胁迫的响应可以见图1,从图中可以看出磷酸化可以参与一些微调,从而来应对非生物胁迫。
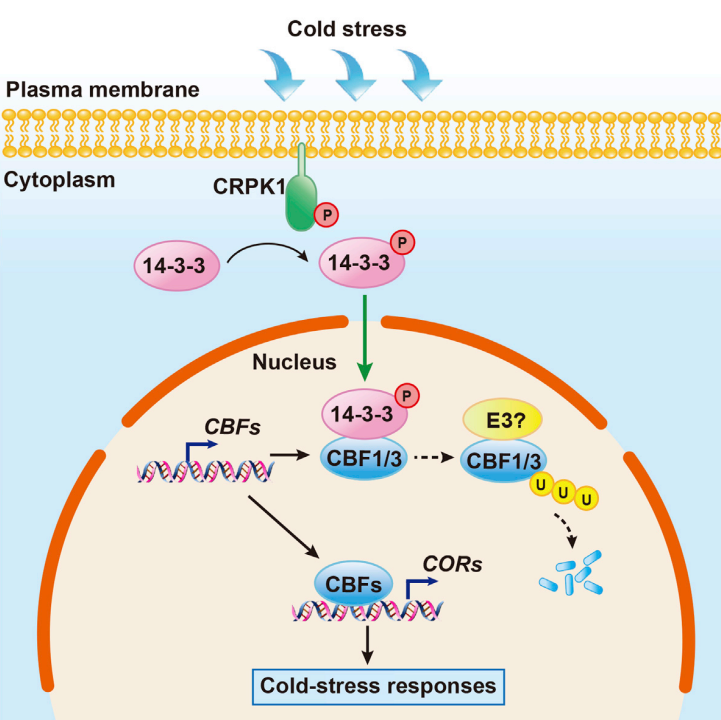
在“Plasma Membrane CRPK1-Mediated Phosphorylation of 14-3-3 Proteins Induces Their Nuclear Import to Fine-Tune CBF Signaling during Cold Response”一文中,作者发现冷激活质膜蛋白冷响应蛋白激酶1(CRPK1)磷酸化14-3-3蛋白。磷酸化的14-3-3蛋白从细胞质穿梭到细胞核,在那里它们与关键的冷反应C-重复结合因子(CBF)蛋白相互作用并破坏其稳定性。与此相一致的是,crpk1和14-3-3kλ突变体的抗冻性增强,而过表达14-3-3λ的转基因植株的抗冻性降低。进一步的研究表明,CRPK1在14-3-3蛋白的核易位和抗冻功能中起着至关重要的作用。因此,作者的研究表明,CRPK1-14-3-3模块将冷信号从质膜传导到细胞核,调节CBF的稳定性,从而确保植物对冷胁迫的调节反应。
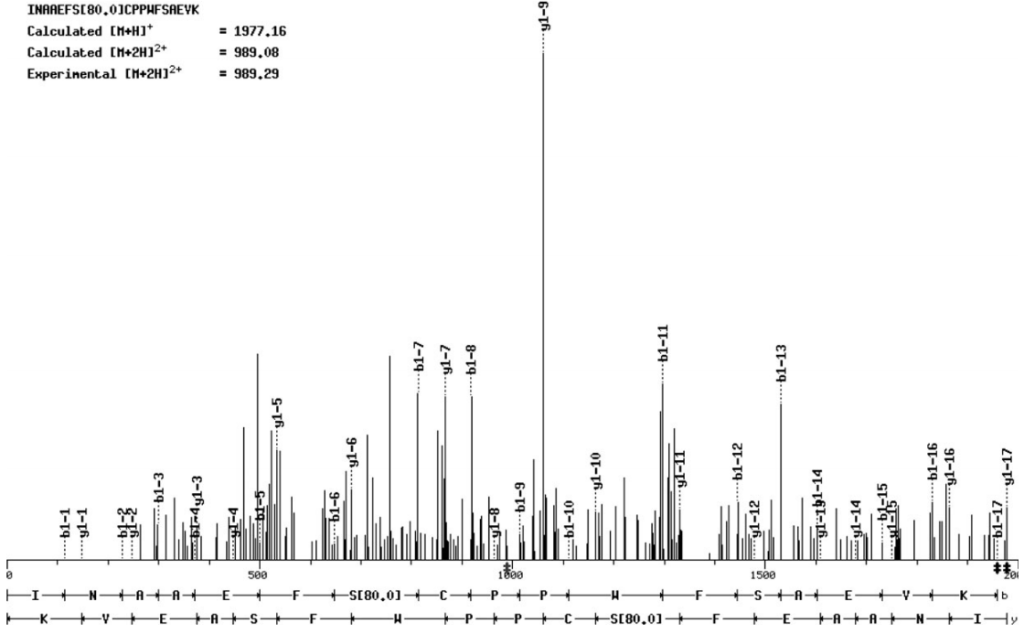
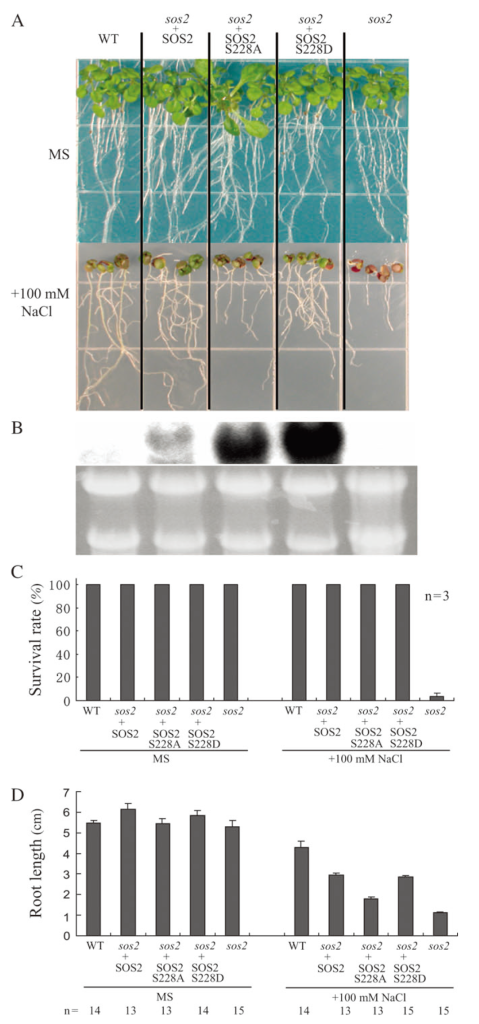
在“An Autophosphorylation Site of the Protein Kinase SOS2 Is Important for Salt Tolerance in Arabidopsis”中,作者鉴定了SOS2中的一个自磷酸化位点,并分析了其在拟南芥对盐胁迫响应中的作用。质谱分析显示SOS2的Ser 228是自磷酸化的。当该位点突变为Ala时,SOS2的自磷酸化速率降低。突变SOS2对底物的磷酸化程度也低于野生型SOS2。相反,将Ser 228转变为Asp来模拟自磷酸化,可以增强SOS2对底物的磷酸化。对sos2突变体的互补试验表明,S228A而非S228D突变部分破坏了SOS2的耐盐功能。作者还发现,Thr168位点的激活环磷酸化和Ser 228位点的自磷酸化不能相互替代,这表明两者都是耐盐所必需的。作者的研究结果表明,SOS2的Ser 228被自磷酸化,并且这种自磷酸化对盐胁迫下SOS2的功能很重要。
图3 在MS/MS肽序列数据中,Ser 228上存在一个磷酸基。N端和C端肽片段离子分别用b离子和y离子系列表示(Hiroaki Fujii and Jian-Kang Zhu, 2009)。
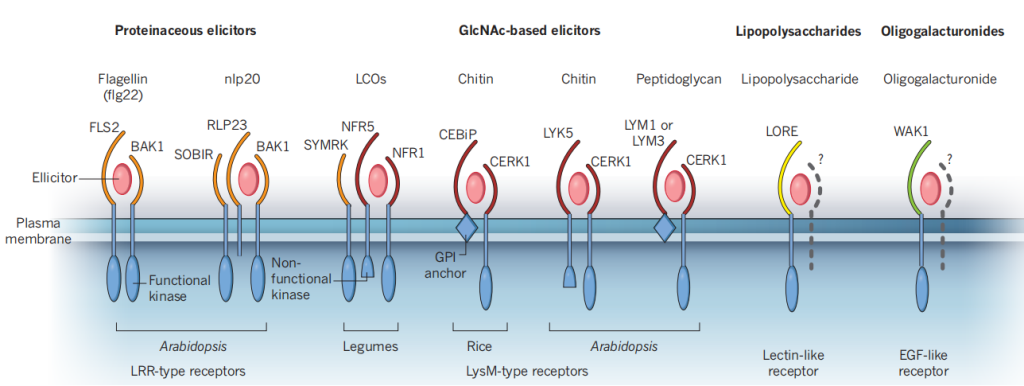
植物对微生物信号的识别,无论是病原体相关分子模式(PAMPs)还是共生信号,都涉及到细胞表面受体激酶复合物。PAMP识别的受体可以是受体激酶,即参与配体感知的胞外结构域、单通道跨膜结构域和胞内激酶结构域,也可以是具有胞外结构域但缺乏胞内信号结构域的受体样蛋白(Daniel Couto and Cyril Zipfel, 2016)。图5显示了参与微生物信号感知的主要受体复合物的类别。
图5 参与微生物感知的LysM-受体-激酶复合物(Cyril Zipfel and Giles E. D. Oldroyd, 2017)。显示了参与微生物信号感知的主要受体复合物的类别。一般来说,蛋白质诱导(例如,鞭毛蛋白flg22和nlp20,一个在细菌、真菌和卵菌中保守的表位)被具有LRR型胞外结构域(橙色)的受体感知,该受体也与LRR型共受体相关。基于GlcNAc的诱导子,如LCOs、几丁质和肽聚糖被存在于受体复合物中的LysM基序(红色)结合。与脂多糖识别相关的受体(类受体凝集素,黄色)和低聚半乳糖醛酸(EGF型受体,绿色)相关的受体也正在被发现。每个受体复合物包含至少一个具有功能性或非功能性激酶基序的受体样激酶。这些复合物还可以包括含有跨膜结构域和胞外结构域但缺乏胞内信号域的受体样蛋白(例如nlp20受体中的RLP23),或通过糖基磷脂酰肌醇(GPI)锚定在膜上的细胞外蛋白(例如水稻几丁质受体中的CEBiP和拟南芥肽聚糖受体中的LYM1和LYM3)。EGF,表皮生长因子。
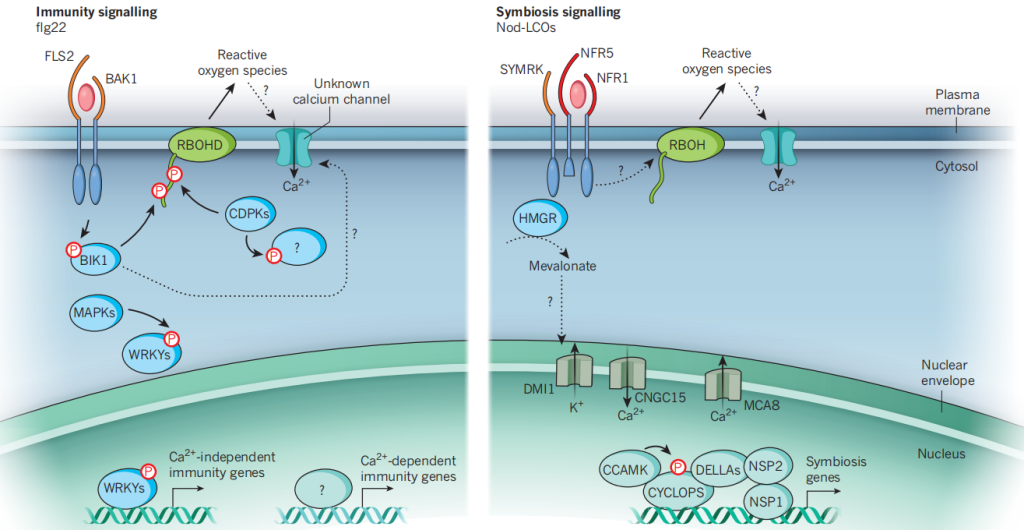
受体复合物的下游信号传递涉及多种过程,在免疫和共生信号通路之间有明显的相似之处。植物细胞对PAMPs的识别导致了一系列的细胞事件,包括外质体活性氧的产生,胞质钙浓度的增加,胞质丝裂原活化蛋白(MAP)激酶和钙依赖蛋白激酶(CDPKs)级联的激活,以及转录变化(Müller et al., 2016)。图6展示了植物免疫和共生细胞内信号通路,可以发现通路中是存在磷酸化作用的。
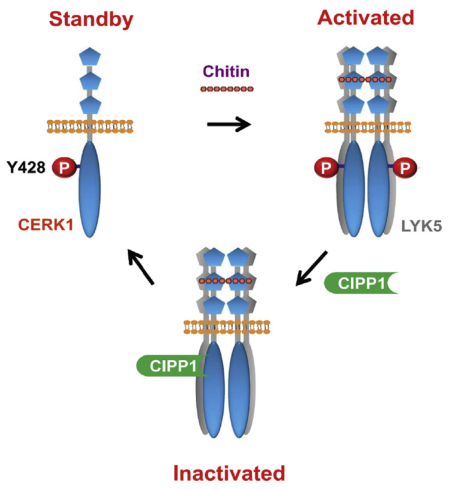
在“A Tyrosine Phosphorylation Cycle Regulates Fungal Activation of a Plant Receptor Ser/Thr Kinase”一文中作者报道了拟南芥PRR几丁质诱导子受体激酶1(CERK1)在未受刺激的细胞中酪氨酸428(Tyr428)位点自磷酸化,这是CERK1与真菌细胞壁成分几丁质结合时所必需的修饰。几丁质激活后,CERK1招募CERK1相互作用蛋白磷酸酶1(CIPP1),一种预测的丝氨酸/苏氨酸磷酸酶,去磷酸化Tyr428并抑制CERK1信号传导。CIPP1随后从Tyr428去磷酸化的CERK1中分离出来,使CERK1重新获得Tyr428的自磷酸化并恢复到备用状态。这项工作揭示了植物几丁质信号传导途径,并表明受体激酶和磷酸酶可以通过磷酸化循环协调调节受体激酶的信号转导。
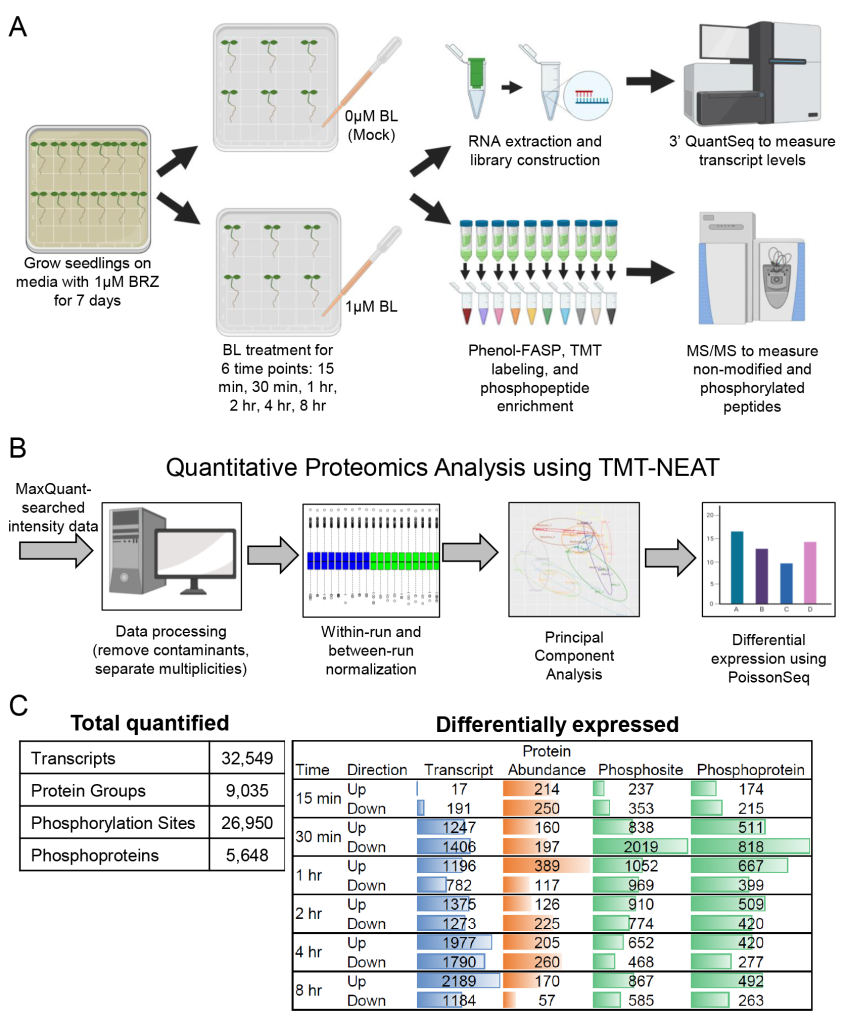
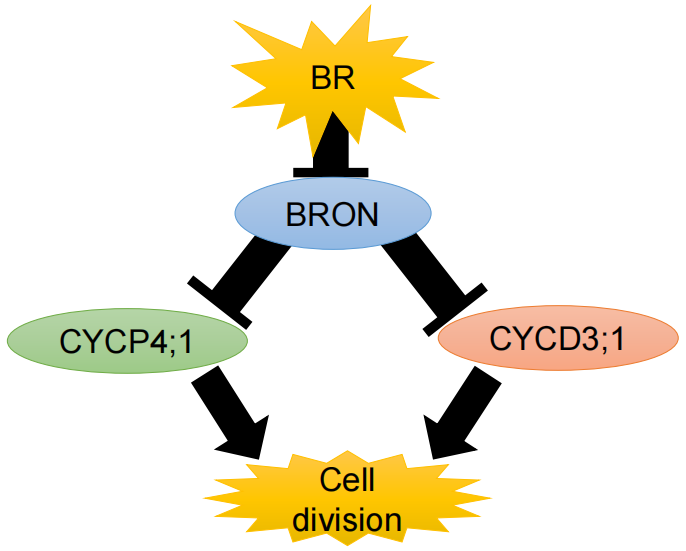
作者分析了BL处理6个不同时间长的拟南芥幼苗的32,549个转录本,9,035个蛋白质组和26,950个磷酸化位点的水平。然后,作者构建了一个称为组学网络时空聚类和组学推理(SC-ION)的网络推理管道,该管道能够将这些多组学数据集成到一个内聚的调控网络中。作者的网络说明了BR反应的信号级联,从激酶底物磷酸化开始,以转录调节结束。作者使用网络预测来确定TF BRI1-EMS抑制因子1(BES1)上假定的相关磷酸化位点;另外还通过实验验证了它的重要性。此外,作者还发现了一种未表征的TF,作者将其命名为BRON-TOSAURUS(BRON),它调节细胞分裂。此外,作者发现born突变根对BL敏感。总之,这项工作展示了应用于多组学数据的综合网络分析的力量,并为BR响应过程中发生的分子信号事件提供了基本的见解。
图8 BR响应的综合组学时间过程(Clark et al., 2021)。(A)收集分别在6个时间点(15min,30min,1h,2h,4h,8h)使用Mock和BL处理的幼苗作为样品。(B)TMT-NEAT定量蛋白质组学分析管道。(C)(左)转录本、蛋白质和磷酸化/磷酸化蛋白在每个时间点定量的总数。(右)每个时间点DE转录物、蛋白质和磷酸化位点/磷酸化蛋白的数量。彩色条形图表示每种数据类型中DE基因产物的相对数量。
References:
Bartels D, Sunkar R. Drought and salt tolerance in plants[J]. Critical reviews in plant sciences, 2005, 24(1): 23-58.
Boyer J S. Plant productivity and environment[J]. Science, 1982, 218(4571): 443-448.
Wang W, Vinocur B, Altman A. Plant responses to drought, salinity and extreme temperatures: towards genetic engineering for stress tolerance[J]. Planta, 2003, 218: 1-14.
Cabello J V, Lodeyro A F, Zurbriggen M D. Novel perspectives for the engineering of abiotic stress tolerance in plants[J]. Current Opinion in Biotechnology, 2014, 26: 62-70.
Chinnusamy V, Zhu J, Zhu J K. Cold stress regulation of gene expression in plants[J]. Trends in plant science, 2007, 12(10): 444-451.
Clark N M, Nolan T M, Wang P, et al. Integrated omics networks reveal the temporal signaling events of brassinosteroid response in Arabidopsis[J]. Nature Communications, 2021, 12(1): 5858.
Couto D, Zipfel C. Regulation of pattern recognition receptor signalling in plants[J]. Nature Reviews Immunology, 2016, 16(9): 537-552.
Cramer G R. Abiotic stress and plant responses from the whole vine to the genes[J]. Australian Journal of Grape and Wine Research, 2010, 16: 86-93.
Cramer G R, Urano K, Delrot S, et al. Effects of abiotic stress on plants: a systems biology perspective[J]. BMC plant biology, 2011, 11(1): 1-14.
Fujii H, Zhu J K. An autophosphorylation site of the protein kinase SOS2 is important for salt tolerance in Arabidopsis[J]. Molecular plant, 2009, 2(1): 183-190.
Khraiwesh B, Zhu J K, Zhu J. Role of miRNAs and siRNAs in biotic and abiotic stress responses of plants[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2012, 1819(2): 137-148.
Krasensky J, Jonak C. Drought, salt, and temperature stress-induced metabolic rearrangements and regulatory networks[J]. Journal of experimental botany, 2012, 63(4): 1593-1608.
Liu J, Liu B, Chen S, et al. A tyrosine phosphorylation cycle regulates fungal activation of a plant receptor Ser/Thr kinase[J]. Cell Host & Microbe, 2018, 23(2): 241-253. e6.
Liu Z, Jia Y, Ding Y, et al. Plasma membrane CRPK1-mediated phosphorylation of 14-3-3 proteins induces their nuclear import to fine-tune CBF signaling during cold response[J]. Molecular cell, 2017, 66(1): 117-128. e5.
Müller D B, Vogel C, Bai Y, et al. The plant microbiota: systems-level insights and perspectives[J]. Annual review of genetics, 2016, 50: 211-234.
Olsen J V, Blagoev B, Gnad F, et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks[J]. Cell, 2006, 127(3): 635-648.
Shinozaki K, Yamaguchi-Shinozaki K. Gene networks involved in drought stress response and tolerance[J]. Journal of experimental botany, 2007, 58(2): 221-227.
Skirycz A, Inzé D. More from less: plant growth under limited water[J]. Current Opinion in Biotechnology, 2010, 21(2): 197-203.
Szabados L, Savouré A. Proline: a multifunctional amino acid[J]. Trends in plant science, 2010, 15(2): 89-97.
Valliyodan B, Nguyen H T. Understanding regulatory networks and engineering for enhanced drought tolerance in plants[J]. Current opinion in plant biology, 2006, 9(2): 189-195.
Wang W X, Vinocur B, Shoseyov O, et al. Biotechnology of plant osmotic stress tolerance physiological and molecular considerations[C]//IV International Symposium on In Vitro Culture and Horticultural Breeding 560. 2000: 285-292.
Yuan L, Liu X, Luo M, et al. Involvement of histone modifications in plant abiotic stress responses[J]. Journal of Integrative Plant Biology, 2013, 55(10): 892-901.
Zipfel C, Oldroyd G E D. Plant signalling in symbiosis and immunity[J]. Nature, 2017, 543(7645): 328-336.