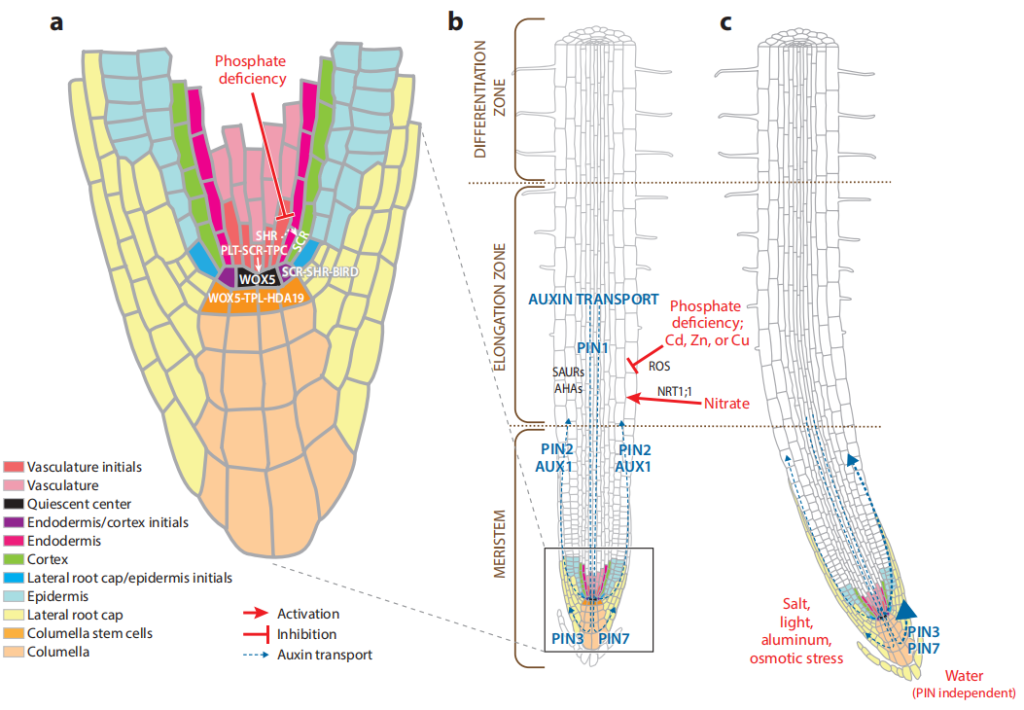
绝大多数真核生物由许多不同的细胞类型组成。在一个生物体中,不同的细胞类型可以从同一组染色体中产生。同时,生物体的所有细胞都能够对非生物和生物胁迫做出响应,如光、温度、化学物质和病原体等。基因表达的时间和空间调控对于成功产生高度特化的细胞类型及其对外部信号的响应至关重要(Motte et al., 2019)(图1)。
图1 真核生物由许多不同的细胞类型组成,生物体的所有细胞都能够对非生物或生物胁迫做出响应,如光、温度、化学物质或病原体等(Motte et al., 2019)。(a)组成植物根系的不同细胞;(b)正在生长的植物根系的不同细胞通过调控基因表达对非生物和生物胁迫做出反应;(c)受到定向刺激的根部。
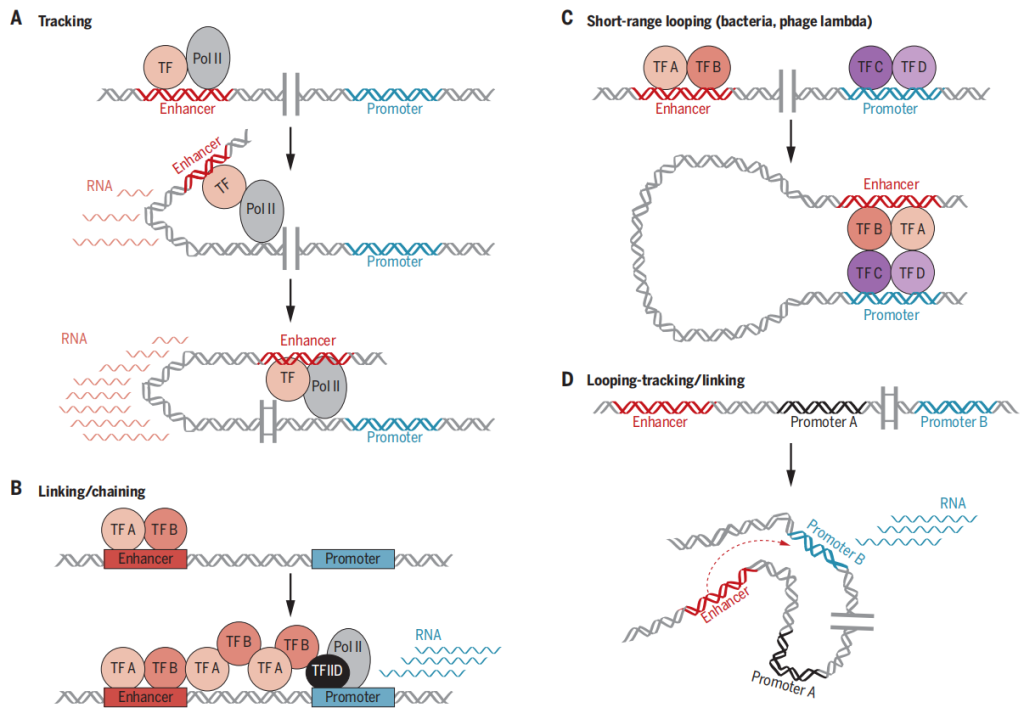
转录调控是基因表达的第一步和关键的调控步骤,也是最早受到关注和研究的最为透彻的分支。超过半个世纪的知识积累构建了一个由顺式调控元件(Cis-regulatory element)和反式作用因子(Trans-acting factor)两大要素相互作用形成的转录调控框架,真核生物mRNA的转录起始就是其中非常经典的模型(Lee et al., 2013;Andersson et al., 2020)(图2)。顺式调控元件主要包括启动子、增强子和沉默子等非编码DNA序列;而反式作用因子则包含RNA聚合酶、转录因子、染色质重塑因子(Chromatin remodeler)、甚至是一些RNA结合蛋白(RNA binding protein,RBP)等(Xiao et al., 2019)。顺式调控元件所蕴含的内在序列特征往往直接决定了反式作用因子的结合与作用模式,而反式作用因子在不同生物场景下的差异性行为也能够反过来影响顺式调控元件的激活。今天伯小远将要为大家介绍的内容就是顺式调控元件中的增强子(Zeitlinger et al., 2020)。
在过去的几十年里,科学家在不同的物种中(包括酵母、真菌、动物和植物中),鉴定和研究了不少增强子。这些增强子主要使用低通量的方法进行鉴定,如增强子捕获、启动子缺失分析、重组分析和数量性状基因座定位。但从上个世纪八十年代至今,对于增强子的研究仍然很大程度上停留在其定义本身,即增强子是什么、在哪里、具有什么功能以及基因组特征(Pennacchio et al., 2013) 。因为其本身多样灵活的空间位置和基因调控模式使得增强子成为了几乎可能遍布于整个基因组的“暗物质”。增强子的特点如下:
1)具有远距离性
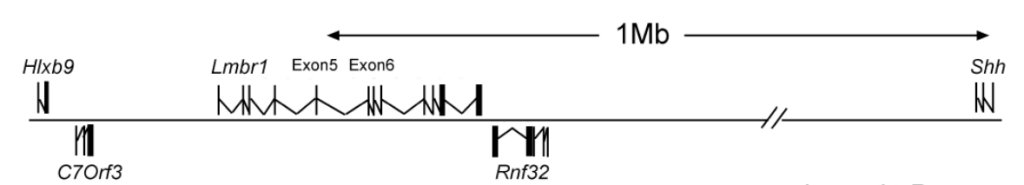
增强子一般位于靶基因上游-200bp处,它可以增强远处启动子的转录,甚至可以激活位于多达一百万bp外的靶基因的表达,如小鼠Shh基因与其增强子ZRS序列之间的调控关系。Shh的增强子ZRS位于1Mb以外的Lmbr1基因的第5个内含子中,ZRS通过与Shh形成loop的方式调节Shh的表达,这是一个典型的增强子案例(Lettice et al., 2003) 。
2)无方向性
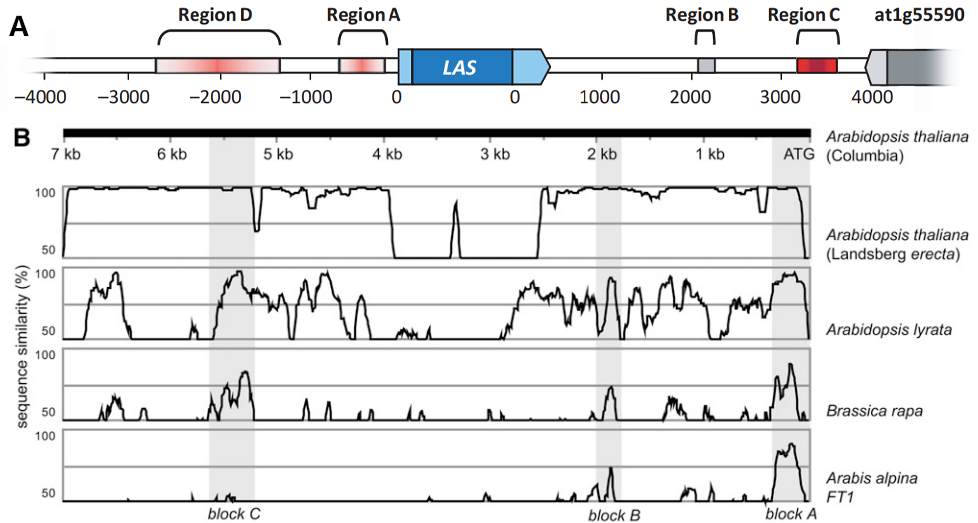
增强子无论位于靶基因的上游、下游或者内部都可以发挥增强转录的作用。例如:调控拟南芥基因LATERAL SUPPRESSOR(LAS)特异性表达的增强子位于LAS开放阅读框下游3.2kb处,而FLOWERING LOCUS T(FT)基因的增强子block C位于该基因的上游5kb左右(Adrian et al., 2010;Raatz et al., 2011) 。
图4 增强子的无方向性(Adrian et al., 2010;Raatz et al., 2011) 。(A)Region C为LAS特异性表达的进化上保守的3’增强子;(B)增强子block C在FLOWERING LOCUS T(FT)基因上游5kb左右。
3)具有组织特异性
在不同种属的细胞中,其增强转录的活性不同。许多增强子只在某些细胞或组织中表现活性,这是由细胞或组织中特异性蛋白质因子所决定的。例如免疫球蛋白基因的增强子只有在B淋巴细胞内活性最高。在胰岛素β细胞中有一种特异性蛋白因子可以作用于人类胰岛素基因的增强子,以增强胰岛素基因的转录,而在其它组织细胞中没有这种蛋白因子,所以胰岛素基因只有在胰岛素β细胞中才能很好地表达。
4)有相位性——其作用和DNA构象有关。
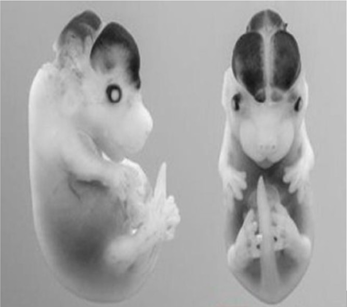
5)可以在异源物种上发挥作用——携带人类HARE5增强子的小鼠大脑发育出只有人类大脑才有的褶皱,并且比带有黑猩猩HARE5增强子的小鼠胚胎形成的大脑大12%。
6)有的增强子可以对外部信号产生反应——某些增强子可以被固醇类激素所激活;热休克基因的增强子在高温下被激活使该基因表达。
由于制备植物TF特异性抗体较为困难,同时TF表达量很低,在植物中ChIP-seq一般考虑构建标签(His、Flag、HA)融合目标蛋白的过表达质粒,使目标蛋白和标签蛋白在受体材料中融合表达,利用标签特异性抗体捕获标签蛋白融合目的蛋白的DNA的复合物。相比于ChIP-seq,CUT&Tag无需交联和片段化、实验简单、信噪比高。而DAP-seq是体外实验,无需制备特异性抗体,可用于无转化体系的植物物种。
活性增强子序列通常位于NDRs中,对核酸酶活性敏感,这样的序列也称为Dnase I 超敏(DH)位点。因此,脱氧核糖核酸酶I测序(DNase-seq)和微球菌核酸酶测序(MNase-seq)都是鉴定顺式调控元件的非常有价值的工具。另一种鉴定染色质开放性的方法是FAIRE-seq。FAIRE通过用甲醛交联组织或细胞,然后对染色质进行超声处理,并用苯酚/氯仿提取无核小体的DNA片段,来鉴定NDRs。ATAC-seq方法最初是作为MNase-seq、FAIRE-seq、DNase-seq的替代方法或补充开发出来的,相比于其他三个测序方法,ATAC-seq实验方法简单、时间短、需要的样本量少等诸多优点,想了解ATAC-seq的详细知识,可阅读我们的往期文章:解析表观遗传学的工具——ATAC-seq(一)。
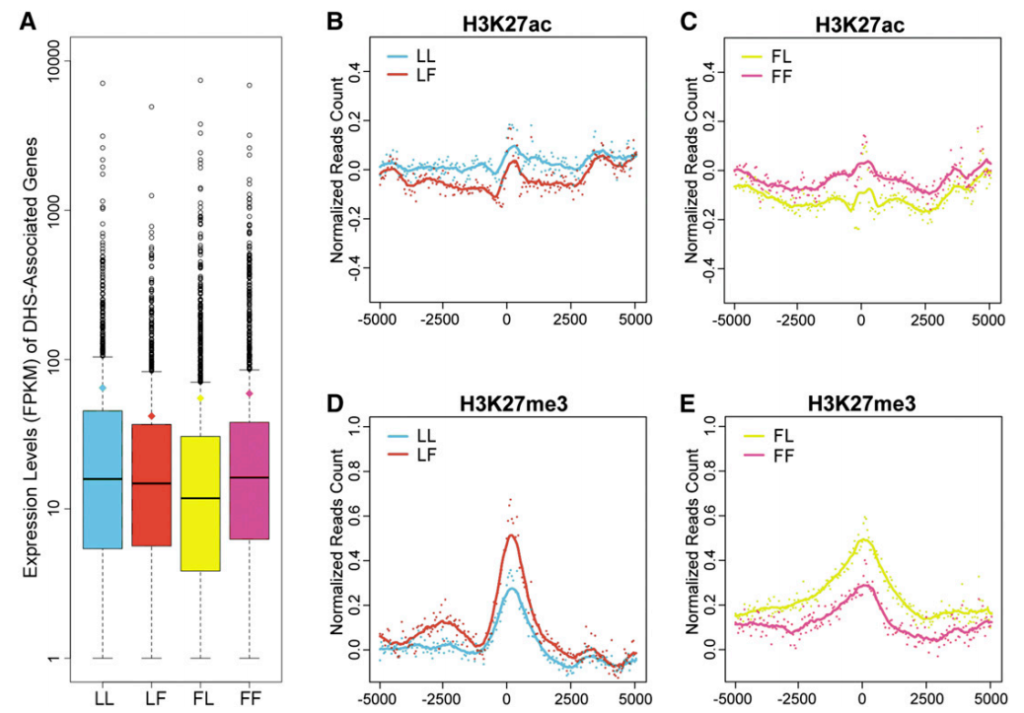
组蛋白修饰在植物增强子及其活性状态尚不完全清楚,但随着研究的不断深入,这方面的内容也在慢慢完善。例如:豌豆PetE和玉米b1的活性增强子分别富含H3/H4ac和H3K9/K14ac。水稻中的基因间NDRs与H4K12ac密切相关,同时也与H3K27me3密切相关。在拟南芥中的一项研究揭示了非活性增强子与H3K27me3具有正相关性,而活性增强子和H3K27ac具有正相关性,前者的相关性比后者更明显(Zhu et al., 2015) 。总之,目前的结果表明,植物活性增强子通常与H3和H4乙酰化有关,而非活性增强子与H3K27me3有关。ChIP-seq和CUT&Tag通常被用于鉴定全基因组范围内组蛋白修饰特征。
可以利用RNA-seq的方法检测eRNA的产生来鉴定增强子活性,但需要高测序深度来检测低丰度的eRNA。通过使用基因表达的帽盖分析(CAGE),仅对RNA的5’端进行测序,可以在较低的测序深度检测eRNA。除了含量低之外,eRNA对降解也很敏感,因此,需要更高灵敏度的方法来检测eRNA。
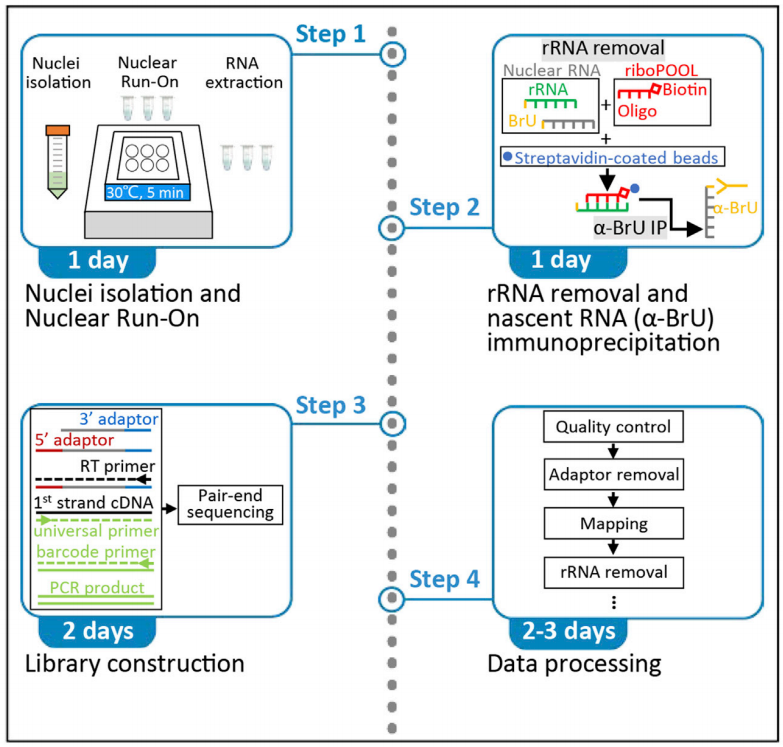
GRO-seq(Genome-wide nuclear run-on assays)能够捕获与活跃转录的RNA聚合酶结合的新生RNA(Nascent RNA),在检测高度不稳定的RNA分子,尤其是发挥关键调控作用的非编码转录本方面具有显著优势,所以GRO-seq是检测eRNA的重要工具(Chen et al., 2022) 。最近开发的GRO-cap在GRO-seq基础上通过特定富集5’加帽的RNAs增加了检测转录起始和捕获RNAs的灵敏度和特异性,这种处理还会降低源于转录后加帽的RNAs的背景信号。
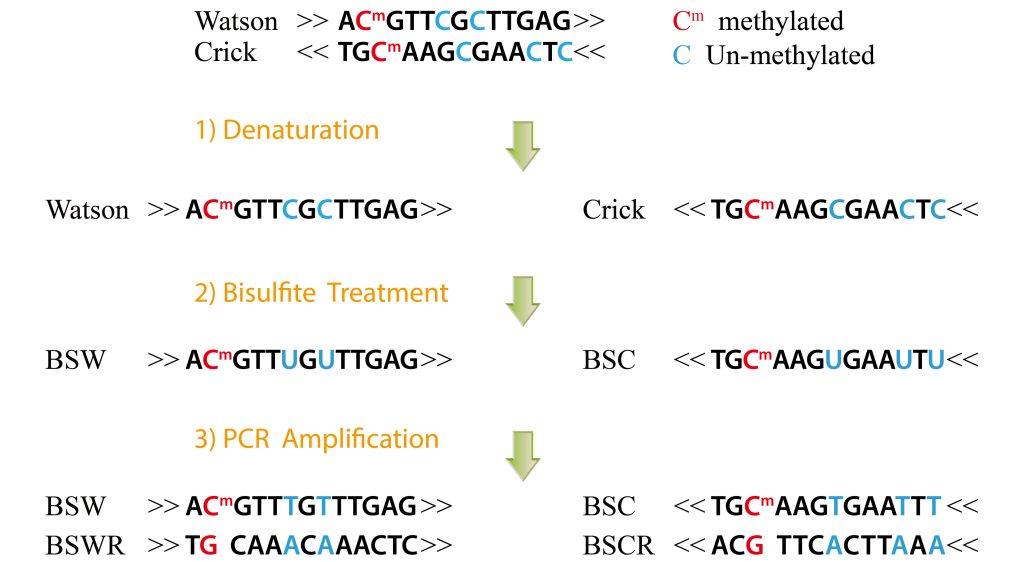
低DNA甲基化水平可能表明存在活性增强子。全基因组DNA甲基化水平可以使用BS(Bisulfite)-seq来检测。用亚硫酸氢盐(Bisulfite)处理DNA可将胞嘧啶残基(C)转化为尿嘧啶(U),但5-甲基胞嘧啶残基(5mC)对其有抗性,并不会发生转变。因此,用亚硫酸氢盐处理过的DNA仅保留甲基化的胞嘧啶。通过上述原理,对基因组DNA进行亚硫酸盐转换,建库和高通量测序,通过对测序Reads中C-T转换进行分析,即可在单碱基分辨率上检测全基因组甲基化修饰的状态。BS-seq首先在拟南芥中实现,然后在其他几种植物中使用。
2019年3月,德国马普研究所Franziska Turck课题组在Nature Plants发表了题为“Targeted DNA methylation represses twoenhancers of FLOWERING LOCUS T in Arabidopsis thaliana“的研究论文。该研究开发了一种通过过表达反向重复序列(IR)介导DNA甲基化RdDM途径的方法,可以鉴定基因的顺式调控元件,并且通过该方法鉴定到拟南芥中FT基因的新型增强子E区,揭示了FT基因的C和E区为转录增强子,其与近端FT启动子组合,在叶韧皮部中响应光周期控制FT的表达(Zicola et al., 2019) 。
染色体构象捕获技术(Chromosome conformation capture,3C)及其衍生技术(如4C、5C、Hi-C、ChIA-PET等)测量不同基因组区域内的染色质相互作用。3C技术的要点是:先用甲醛交联染色质,固定蛋白与DNA,使染色质保持三维结构。再用一种限制性内切酶(HindIII、BglII、SacI、BamH或EcoRI)切割染色质,蛋白周围的非互作基因被切开,使互作DNA与其他非互作DNA分离。这样形成的loop有两种,一种是同一基因间的loop,一种是互作基因间的loop,用PCR的方式区分这两种loop。
大多数3C通常仅能分析几十到几百kb染色质之间的相互作用,通量低,费时费力。4C用于测定一点到多点之间的染色质交互作用。使用反向PCR产生单基因座的全基因组相互作用图,研究已知DNA片段(bait)与全基因组未知DNA片段之间的互作。5C用于测定多点到多点之间的染色质交互作用。基于3C的基本原理,结合连接介导的扩增来增加3C检测的通量,识别两组大量位点之间并行的数百万个相互作用。ChIA-PET配对末端标签测序分析染色质相互作用技术,把染色质免疫沉淀(ChIP)技术、染色质邻近式连接(Chromatin proximity ligation)技术、配对末端标签(Paired-endtag,PET)技术和新一代测序(Next-generation sequencing)技术融为一体,在基因组三维折叠和套环状态下分析基因表达和调控,全基因组范围内分析远程染色质相互作用。而Hi-C技术以整个细胞核为研究对象,利用高通量测序技术,结合生物信息分析方法,研究全基因组范围内整个染色质DNA在空间位置上的关系,通过对染色质内全部DNA相互作用模式进行捕获,获得高分辨率的染色质三维结构信息。
Adrian J, Farrona S, Reimer JJ, et al. 2010. cis-Regulatory elements and chromatin state coordinately control temporal and spatial expression of FLOWERING LOCUS T in Arabidopsis. Plant Cell 22:1425-1440
Andersson, R. & Sandelin, A. 2020. Determinants of enhancer and promoter activities of regulatory elements. Nat. Rev. Genet 21, 71–87.
Chen Y, Zhu J, Xie Y, et al. 2022. Protocol for affordable and efficient profiling of nascent RNAs in bread wheat using GRO-seq. STAR Protoc 3:101657.
Creyghton, M. P. et al. 2010. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proc. Natl. Acad. Sci. U. S. A. 107, 21931–21936 .
Heintzman, N. D. et al. 2007. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nat. Genet 39, 311–318.
Lee, T. I. & Young, R. A. 2013. Transcriptional regulation and its misregulation in disease. Cell 152, 1237–1251.
Lettice LA, Heaney SJ, Purdie LA, et al. 2003. A long-range Shh enhancer regulates expression in the developing limb and fin and is associated with preaxial polydactyly. Hum Mol Genet 12:1725-1735.
Mathelier A, Zhao X, Zhang AW, et al. 2014. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res 42:D142-147.
Motte H, Vanneste S, Beeckman T. 2019. Molecular and Environmental Regulation of Root Development. Annu Rev Plant Biol 70:465-488.
O’Malley RC, Huang SC, Song L, et al. 2016. Cistrome and Epicistrome Features Shape the Regulatory DNA Landscape. Cell 165:1280-1292.
Pennacchio, L. A., Bickmore, W., Dean, A., Nobrega, et al. 2013. G. Enhancers: Five essential questions. Nat. Rev. Genet. 14, 288–295.
Raatz B, Eicker A, Schmitz G, et al. 2011. Specific expression of LATERAL SUPPRESSOR is controlled by an evolutionarily conserved 3′ enhancer. Plant J 68:400-412.
Weber B, Zicola J, Oka R, et al. 2016. Plant Enhancers: A Call for Discovery. Trends Plant Sci 21:974-987.
Xiao, R. et al. 2019. Pervasive Chromatin-RNA Binding Protein Interactions Enable RNA-Based Regulation of Transcription. Cell 178, 107-121.e18 .
Zhu B, Zhang W, Zhang T, et al. 2015. Genome-Wide Prediction and Validation of Intergenic Enhancers in Arabidopsis Using Open Chromatin Signatures. Plant Cell 27:2415-2426.
Zeitlinger, J. 2020. Seven myths of how transcription factors read the cis-regulatory code. Curr. Opin. Syst. Biol 301, 127065.
Zicola J, Liu L, Tanzler P, et al. 2019. Targeted DNA methylation represses two enhancers of FLOWERING LOCUS T in Arabidopsis thaliana. Nat Plants 5:300-307.
Zhang W, Zhang T, Wu Y, et al. 2012. Genome-wide identification of regulatory DNA elements and protein-binding footprints using signatures of open chromatin in Arabidopsis. Plant Cell 24:2719-2731.