孤生作物是建立多样化和气候适应型粮食体系的关键,气候变暖促使研究人员更加关注lablab这样的作物。扁豆(lablab)一种原产于非洲的豆科植物,抗旱能力强可在热带地区种植,是食品和饲料的来源。基因组辅助育种能解锁用于作物改良的遗传变异,为新的绿色革命带来希望。然而,之前用短读测序生成的lablab基因组草图代表性严重不足。为了解决这一问题,近日来自南安普顿大学生物科学学院的Mark A. Chapman联合国际畜牧研究所的Oluwaseyi Shorinola和Chris S. Jones团队在Nature Communications 杂志上在线发表了名为Chromosome-level genome assembly and population genomic resource to accelerate orphan crop lablab breeding的研究论文。该研究基于长读长测序进行染色体水平基因组组装,并对野生和驯化种质重测序发现了lablab的双重驯化起源。作者还对lablab群体中的遗传和表型多样性进行研究。
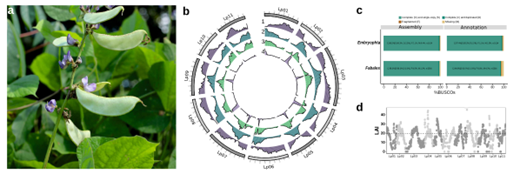
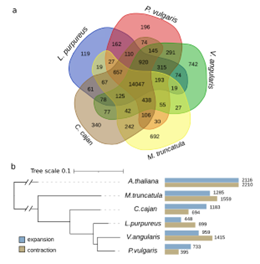
作者用Oxford Nanopore Technology (ONT) MinION平台对lablab(cv. Highworth)的基因组进行测序,生成了28.4 Gbp的数据, reads平均长度为6.1 kb。最初组装成2260个contigs,N50为11.0 Mbp,总组装长度为426.2 Mbp。利用Hi-C数据把contigs聚集到覆盖417.9 Mbp的11个染色体组,N50为38.1 Mbp。接着对lablab基因组进行注释,共有30922个基因,79512个转录本,平均每个基因有2.57 个亚型。还鉴定出168174个TE序列,占基因组28.1%。和其他豆科植物(P.vulgaris、V.angularis、Cajanus cajan、Medicago truncatula)比较进行了基因家族分析。五种豆科植物有14047个共同的orthogroups,并在119个物种特异性orthogroups中鉴定到417个lablab基因。这些特异的基因富集在脂肪酸生物合成、阿拉伯糖代谢等通路。与其他豆科植物和拟南芥相比,lablab中有448个基因家族扩张,899个基因家族收缩。扩张的基因家族富集在木质素和果胶代谢,以及光合作用等通路,收缩的基因家族参与了如酰胺生物合成和代谢过程。
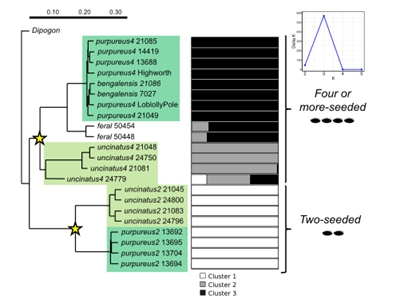
先前的工作表明lablab至少发生两次驯化,分别发生在双种子和四种子时期。为了验证作者用野生和驯化种质进行重测序。分析发现在23个样本中共鉴定出25940192个变异(23.7M SNPs和2.2M indels)。用包含157913个变异的数据集构建了进化树,发现双种子和四种子 lablab在野生和驯化样本之间存在明显分类。作者还研究基于多种证据,证实了先前关于lablab的两个驯化起源的假说。
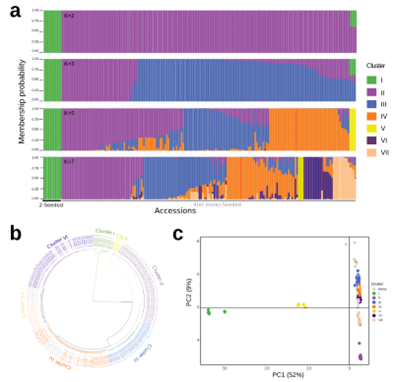
作者收集了来自25个国家和地区的203份lablab种质材料,分析lablab基因组多样性。用STRUCTURE进行群体结构分析和聚类。通过层次聚类和 PCA 检测到相似的聚类和群体分层。第一类和第五类仅包含来自非洲的种质,分别包括所有野生四种子和双种子种质。集群III和VII中的材料主要来自亚洲和澳大利亚。基于历史气象学和形态农艺性状数据,几种种群聚类在表型上有所不同。
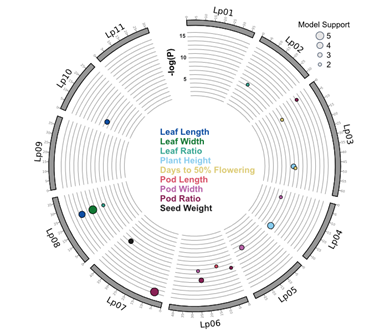
作者将125份lablab种质的表型数据与经过质量过滤的DArTseq标记结合起来,进行全基因组关联分析(GWAS)。在八条染色体上鉴定到18个标记(8个SNP和10个SilicoDArT),这些标记与叶长、叶宽、叶比、株高、开花天数、豆荚长度、荚宽、荚率和千粒重性状关联。相关标记解释了7-24%的表型变异。这些标记可用于标记辅助选择对lablab进行遗传改良。
文章来源:植物生物技术Pbj