首先,CRY2经过蓝光依赖的寡聚反应成为活性四聚体(Sang et al., 2005;Shao et al., 2020)。BIC1和BIC2(隐色素1和2的蓝光抑制剂)蛋白与光激发的CRY2相互作用,负调控CRY2的光寡聚,而在没有光的情况下,活性的CRY2同聚物也可能发生热松弛,成为非活性单体(Wang et al., 2016;Shao et al., 2020)。
其次,CRY2同聚物的活性受到四种相关蛋白激酶PPKs(光调节蛋白激酶1-4)催化的蛋白磷酸化反应的正调控(Shalitin et al., 2002;Wang et al., 2015;Liu et al., 2017)。
最后,光激发和磷酸化的CRY2蛋白在E3泛素连接酶Cul4COP1/SPAs的催化下进行多泛素化,随后被26S蛋白酶体降解(Lin et al., 1998;Yu et al., 2007;Liu et al., 2017)。
与动物CRYs一样,植物CRYs的丰度和整体细胞活性受磷酸化、泛素化和蛋白水解的调节(Wang Q and Lin C, 2020)。然而,目前已知只有cullin 4家族E3泛素连接酶Cul4COP1/SPAs调控植物CRYs(Wang Q and Lin C, 2020)的泛素化和降解,这就提出了高度保守的CRYs在不同进化谱系中是如何被差异调控的问题。
在动物模型中,多种E3泛素连接酶调控CRYs,先前的遗传学研究也表明,植物CRYs中存在多种E3泛素连接酶。然而,到目前为止,只有一种E3连接酶Cul4COP1/SPAs被报道用于植物CRYs。根据先前的研究表明:Cul3LRBs具有调节PIF3泛素化和phyB依赖的红光反应的重要功能,但是尚不清楚Cul3LRBs是否在CRY依赖的信号转导过程中发挥作用。看过本文的同学应该知道,作者通过研究表明Cul3LRBs是拟南芥CRY2的第二个E3连接酶。
以上就是关于这一文献案例的研究背景部分,有了这部分的铺垫大家再去理解单个实验的时候可能会更容易!另外在研究背景部分出现了几个不同的E3泛素连接酶的写法(Cul4COP1/SPAs,Cul3LRBs),不知道大家对这个是否了解,说实话小远一开始不是很了解,所以专门去查了一下文献资料,下面是小远查到的一些资料,希望也能够帮助到你!
E3s是泛素化途径中种类最多的酶(人类有600种E3s),因为它们介导了底物特异性。目前,E3连接酶根据特征域的存在和泛素转移到底物蛋白的机制可分为三种主要类型。这里主要为大家介绍第一类(因为文中涉及E3泛素连接酶的属于第一类),剩下的两类给大家简单展示一下,如果大家感兴趣可以自己去下载对应的参考文献查看噢!
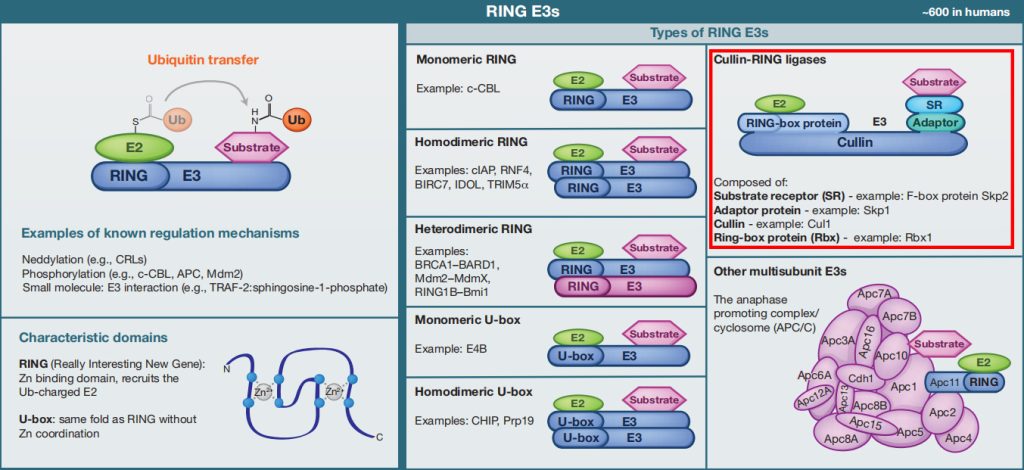
①RING E3s
RING E3s是最丰富的泛素连接酶类型。它们的特征是存在一个被称为RING(Really Interesting New Gene)的锌结合结构域,或存在一个U-box结构域,它采用相同的RING折叠,但不包含锌。RING和U-box结构域负责结合带泛素带电的E2并刺激泛素转移。
RING E3介导泛素直接转移到底物,作为一个支架,使带泛素带电的E2相对于底物蛋白定向。RING E3可以作为单体、同型二聚体,或异型二聚体来发挥作用。同二聚体环通常可以结合两个E2(每个单体各结合一个);但异二聚体环似乎不是这样。类似地,U-box结构域也可以作为单体或同型二聚体工作。
一些RING E3s是由多个亚基组成的,如cullin-RING连接酶(CRLs)。CRLs是一种高度多样化的泛素连接酶,具有几个共同的特征。它们在cullin支架上组装,在其N端结合一个RING-box蛋白,在其C端结合一个适配器蛋白和一个底物受体(负责底物特异性)。
另一个重要的多亚基E3是后期促进复合物/环小体(APC/C),这是一个由19个亚基组成的大型组装体,其中包括一个RING亚基(Apc11)和一个类似于Cullin的亚基(Apc2)。
图1 RING E3s的分类(Morreale F E and Walden H, 2016)。
上面提到的Cul4COP1/SPAs,Cul3LRBs就属于RING E3s这一类里面的,具体的类别小远已经用红色虚线框标出来了,更详细的内容就需要大家自己去动动手指去查阅相关文献了,这里面涉及的内容实在是太多了,小远只能帮你到这儿了!
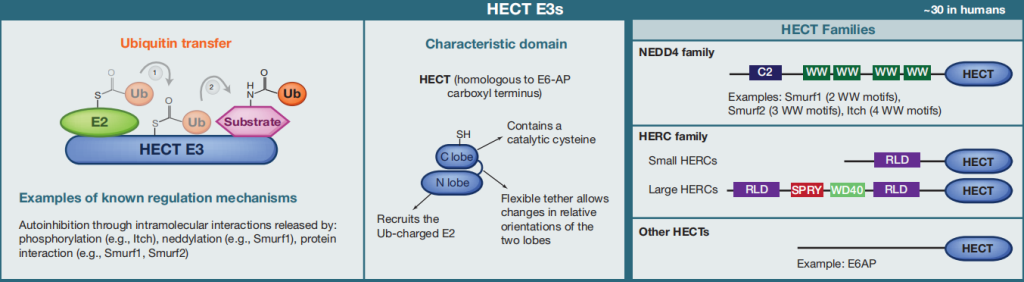
②HECT E3s
图2 HECT E3s的分类(Morreale F E and Walden H, 2016)。
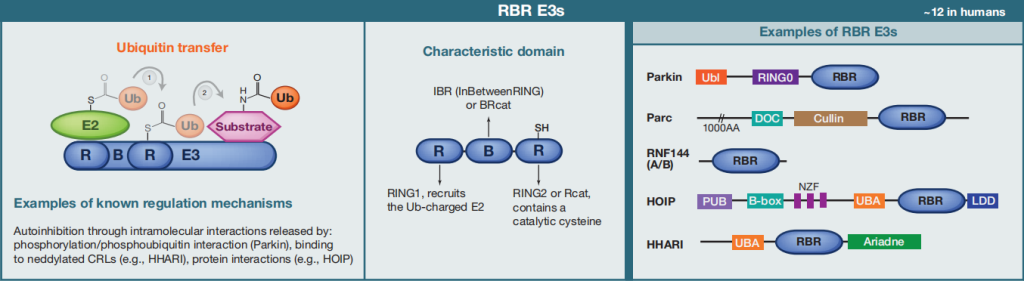
③RBR E3s
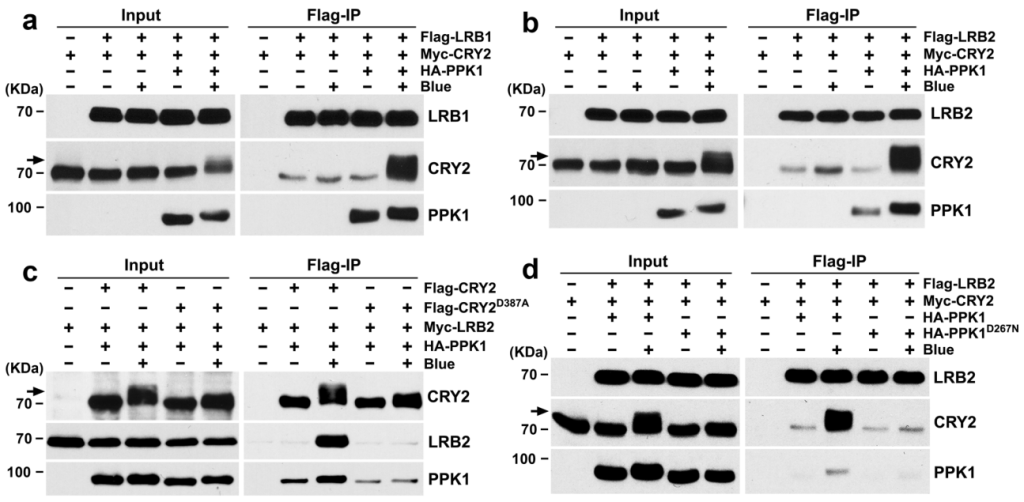
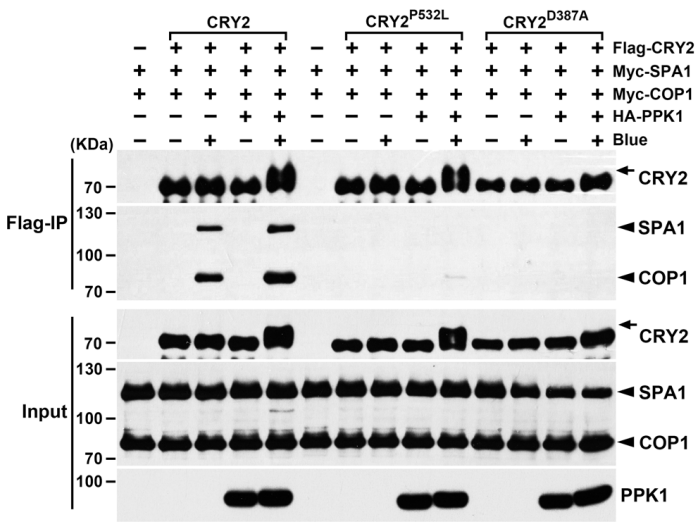
图4 LRBs与HEK293T细胞中磷酸化的CRY2相互作用(Chen et al., 2021)。(a)免疫共沉淀(Co-IP)分析显示LRB1和CRY2在异源HEK293T细胞中存在蓝光和磷酸化依赖的相互作用。用Flag偶联珠子进行免疫沉淀(IP)。IP(LRB1)和Co-IP(CRY2)产物分别用抗Flag抗体和抗CRY2抗体进行检测。用抗HA抗体检测PPA1。(b)Co-IP检测显示,在HEK293T细胞中,LRB2和CRY2存在蓝光和磷酸化依赖的相互作用。(c)Co-IP分析显示LRB2不能与CRY2D387A突变体相互作用。IP(CRY2和CRY2D387A)和Co-IP(LRB2)产物分别用抗CRY2和抗MYC抗体检测。(d)Co-IP检测显示了在HEK293T细胞中,LRB2和CRY2存在磷酸化依赖的相互作用。PPK1D267N:无催化活性的PPK1。
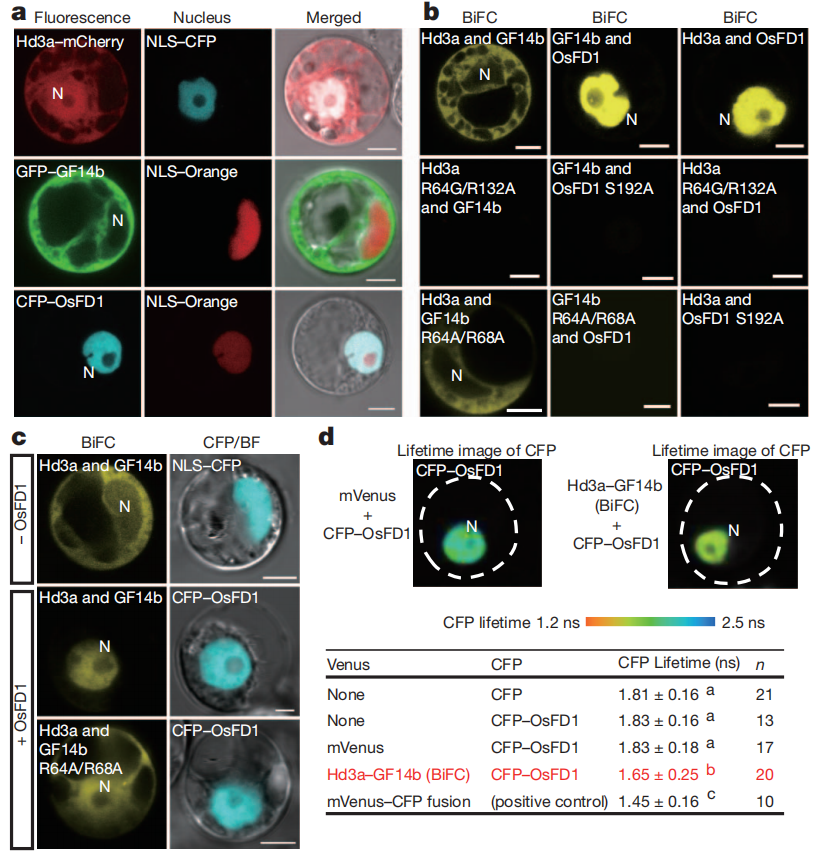
Co-IP除了检测三个蛋白之间的相互作用之外,还可以检测更多蛋白之间的相互作用吗?答案是肯定的,在小远列举的这篇文献案例中就有这样的例子,不过具体的结果就不给大家解读了,就当小远给大家布置的作业,希望大家可以自己把下面这个图搞清楚噢!
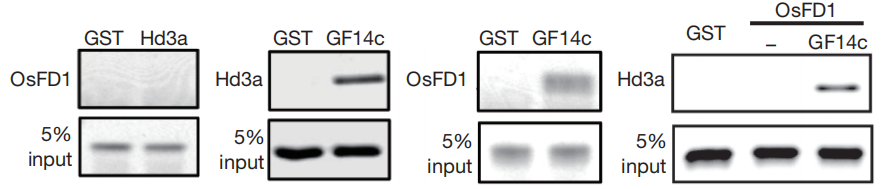
OsFD1:开花时间基因OsFD1编码bZIP转录因子。短日照下,成花素Hd3a与14-3-3蛋白在茎端细胞互作,形成复合物转运至核内,与转录因子OsFD1结合形成三元成花素激活复合物,诱导OsMADS15转录,促进开花。长日照下,成花素RFT1通过14-3-3蛋白与磷酸化的bZIP转录因子OsFD1互作形成三元成花素激活复合物(FAC),正调控长日照下花特征基因OsMADS14/15/18/34表达,从而在长日照下启动水稻成花转变。OsFD1中192位丝氨酸(S192)的磷酸化修饰对FAC形成至关重要,有利于FAC进入核内。
GF14c:水稻14-3-3家族中的一个基因,14-3-3/GF14蛋白在防卫反应和非生物胁迫应答中有重要功能,水稻基因组中至少存在8个编码这类蛋白的基因:GF14a,GF14b,GF14c,GF14d,GF14e,GF14f,GF14g,GF14h。GF14c基因编码的蛋白是一个核定位蛋白,只在幼苗叶片、节间和抽穗期的穗部表达。水稻成花素Hd3a(FT同源物)能与14-3-3蛋白在茎顶端细胞中互作,互作形成的复合物转运到核内,与转录因子OsFD1互作。产生的成花素激活复合物(FAC)能诱导OsMADS15基因,即AP1,促进开花。14-3-3蛋白是成花素的胞内受体,这为操控各种作物和树木的开花提供了新的方法。
上面对这几个基因的介绍相对于将要介绍的文献来说是超前的,在2011年那个年代,成花素在花诱导过程中的确切功能是不清楚的,成花素受体也尚未确定。因此,作者对此进行了探索(虽然现在都已经很清楚了),在探索的过程中,作者为了了解OsFD1、Hd3a、GF14c三者之间的关系,利用GST pull-down的方法进行了验证。
作者通过研究发现两个Hd3a分子似乎通过14-3-3和bZIP蛋白的相互作用被锚定在OsMADS15启动子DNA上。然后,作者研究了这三种蛋白质在水稻原生质体中的亚细胞定位和相互作用。作者在细胞质和细胞核中检测到了mCherry标记的Hd3a的荧光;细胞质中GFP标记的GF14b,细胞核中也有,但比较微弱;细胞核中用CFP标记的OsFD1(图7a)。
通过双分子荧光互补(BiFC)检测Hd3a-GF14b的相互作用,且在GF14b主要定位的细胞质中检测到(图7b)。值得注意的是,GF14b-OsFD1的BiFC信号主要在细胞核中检测到,而单独的GF14b始终主要在细胞质中检测到。BiFC还检测到细胞核中Hd3a和OsFD1之间的关联,如报道的FT-FD的相互作用(Abe et al., 2005;Wigge et al., 2005)。然而,Hd3a R64G/R132A和OsFD1 S192A突变体中Hd3a-OsFD1相互作用被取消,这两个突变体也失去了与GF14b相互作用的能力。
为了评估OsFD1对Hd3a、GF14b和Hd3a-GF14b复合物亚细胞定位的影响,作者将上述实验与共表达CFP-OsFD1相结合。当共表达CFP-OsFD1时,GFP-GF14b、Hd3a-mCherry和Hd3a-GF14b复合物明显集中在细胞核内(7c)。综上所述,这些结果表明Hd3a-GF14b复合物从细胞质转移到细胞核,在细胞核中与OsFD1形成更大的蛋白质复合物。
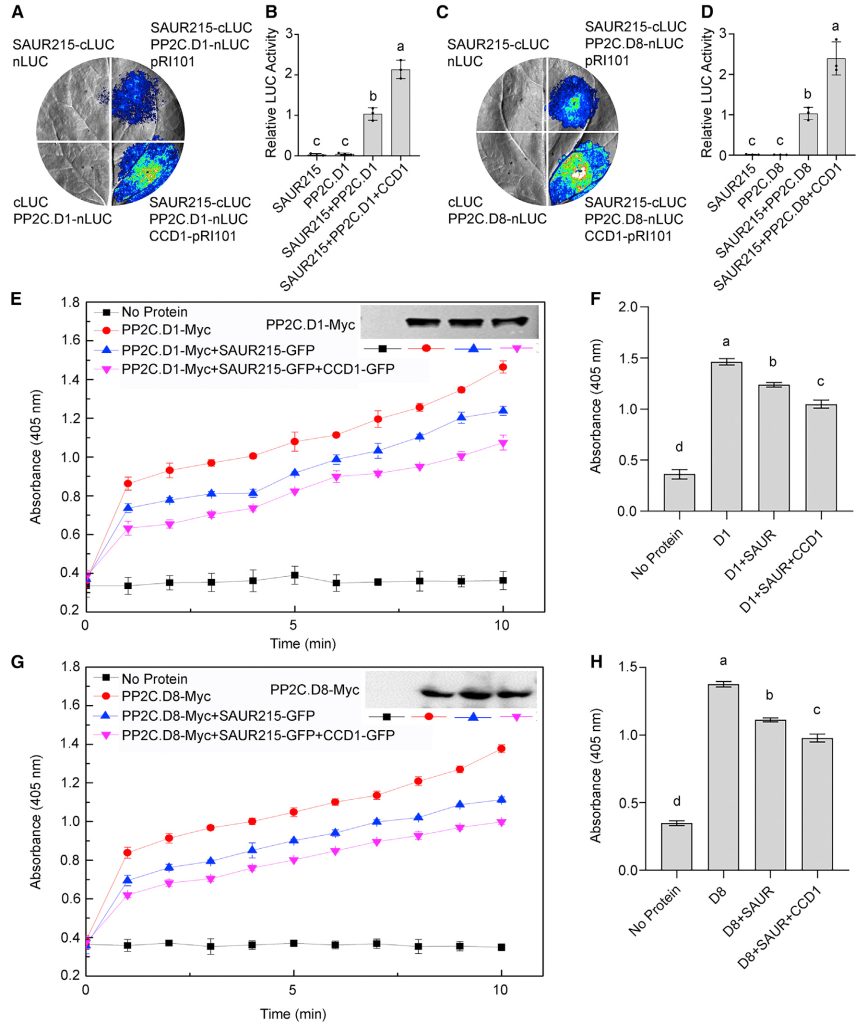
TaSAUR215:早期生长素反应蛋白
TaPP2C.D1/8:2C型蛋白磷酸酶
References:
Abe M, Kobayashi Y, Yamamoto S, et al. FD, a bZIP protein mediating signals from the floral pathway integrator FT at the shoot apex[J]. Science, 2005, 309(5737): 1052-1056.
Cashmore A R. Cryptochromes: enabling plants and animals to determine circadian time[J]. Cell, 2003, 114(5): 537-543.
Chen Y, Hu X, Liu S, et al. Regulation of Arabidopsis photoreceptor CRY2 by two distinct E3 ubiquitin ligases[J]. Nature communications, 2021, 12(1): 2155.
Cui M, Li Y, Li J, et al. Ca 2-dependent TaCCD1 cooperates with TaSAUR215 to enhance plasma membrane H-ATPase activity and alkali stress tolerance by inhibiting PP2C-mediated dephosphorylation of TaHA2 in wheat[J]. 2023.
Guo H, Yang H, Mockler T C, et al. Regulation of flowering time by Arabidopsis photoreceptors[J]. Science, 1998, 279(5355): 1360-1363.
Kwaaitaal M, Keinath N F, Pajonk S, et al. Combined bimolecular fluorescence complementation and Förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells[J]. Plant physiology, 2010, 152(3): 1135-1147.
Lin C, Yang H, Guo H, et al. Enhancement of blue-light sensitivity of Arabidopsis seedlings by a blue light receptor cryptochrome 2[J]. Proceedings of the National Academy of Sciences, 1998, 95(5): 2686-2690.
Liu Q, Wang Q, Deng W, et al. Molecular basis for blue light-dependent phosphorylation of Arabidopsis cryptochrome 2[J]. Nature communications, 2017, 8(1): 15234.
Liu Q, Wang Q, Liu B, et al. The blue light-dependent polyubiquitination and degradation of Arabidopsis cryptochrome2 requires multiple E3 ubiquitin ligases[J]. Plant and Cell Physiology, 2016, 57(10): 2175-2186.
Morreale F E, Walden H. Types of ubiquitin ligases[J]. Cell, 2016, 165(1): 248-248. e1.
Sancar A. Cryptochrome: the second photoactive pigment in the eye and its role in circadian photoreception[J]. Annual review of biochemistry, 2000, 69(1): 31-67.
Sang Y, Li Q H, Rubio V, et al. N-terminal domain–mediated homodimerization is required for photoreceptor activity of arabidopsis cryptochrome 1[J]. The Plant Cell, 2005, 17(5): 1569-1584.
Shalitin D, Yang H, Mockler T C, et al. Regulation of Arabidopsis cryptochrome 2 by blue-light-dependent phosphorylation[J]. Nature, 2002, 417(6890): 763-767.
Shao K, Zhang X, Li X, et al. The oligomeric structures of plant cryptochromes[J]. Nature Structural & Molecular Biology, 2020, 27(5): 480-488.
Shyu Y J, Suarez C D, Hu C D. Visualization of AP-1–NF-κB ternary complexes in living cells by using a BiFC-based FRET[J]. Proceedings of the National Academy of Sciences, 2008, 105(1): 151-156.
Taoka K, Ohki I, Tsuji H, et al. 14-3-3 proteins act as intracellular receptors for rice Hd3a florigen[J]. Nature, 2011, 476(7360): 332-335.
Wang Q, Barshop W D, Bian M, et al. The blue light-dependent phosphorylation of the CCE domain determines the photosensitivity of Arabidopsis CRY2[J]. Molecular plant, 2015, 8(4): 631-643.
Wang Q, Lin C. Mechanisms of cryptochrome-mediated photoresponses in plants[J]. Annual Review of Plant Biology, 2020, 71: 103-129.
Wang Q, Zuo Z, Wang X, et al. Photoactivation and inactivation of Arabidopsis cryptochrome 2[J]. Science, 2016, 354(6310): 343-347.
Yu X, Klejnot J, Zhao X, et al. Arabidopsis cryptochrome 2 completes its posttranslational life cycle in the nucleus[J]. The Plant Cell, 2007, 19(10): 3146-3156.
Wigge P A, Kim M C, Jaeger K E, et al. Integration of spatial and temporal information during floral induction in Arabidopsis[J]. Science, 2005, 309(5737): 1056-1059.