NRT1.1是第一个被克隆的硝酸盐转运蛋白,近几十年来,科学家们对NRT1.1开展了大量的研究,是目前研究的最为清楚的硝酸盐转运体。
目前,研究发现NRT1.1具有以下功能:
1,NRT1.1是双亲和性硝酸盐转运体;
2,NRT1.1是NO3-的信号感受器;
3,NRT1.1显示生长素转运活性;
4,NRT1.1可对非生物胁迫进行应答调控。
科学家们是如何一步一步对这个基因进行研究的呢,和伯小远一起来看看吧。
后来,Tsay等从基因组中首次克隆出CHL1基因,确认CHL1(AtNRT1.1)具有转运硝酸盐的功能,在植物中调控根对NO3–的吸收(图2) (Tsay et al., 1993)。该研究首次成功地鉴定出了植物中的硝酸盐转运基因,开启了解析植物利用硝酸盐分子机制的研究热潮。
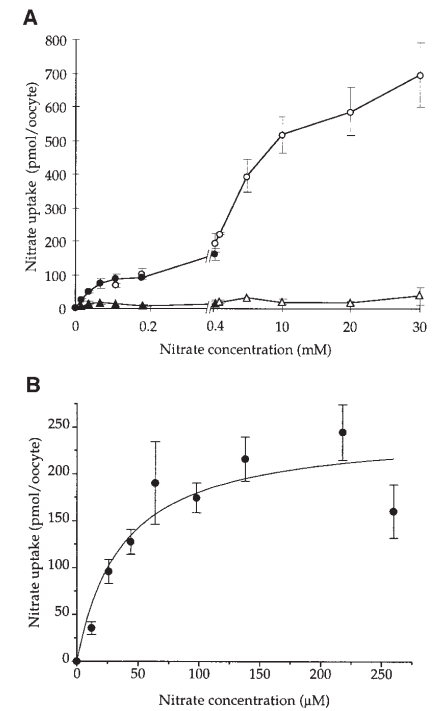
以上结果表明NRT1.1是一个双亲和硝酸盐转运体,即其兼具高、低亲和硝酸盐转运活性。
这两种亲和性是如何转换的呢?
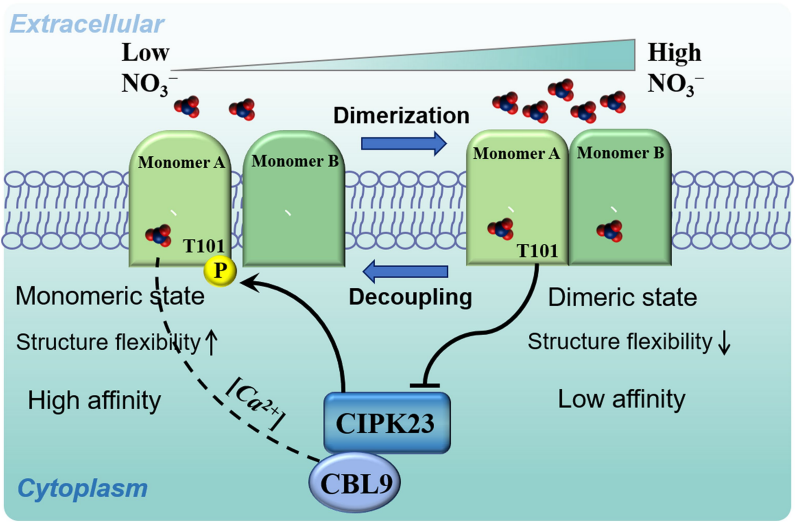
将NRT1.1上第101位苏氨酸突变为丙氨酸(T101A,模拟去磷酸化状态),其表现为低亲和NO3–转运蛋白活性。将第101位苏氨酸突变为天冬氨酸(T101D,模拟磷酸化状态),其表现为高亲和NO3–转运蛋白活性 (Liu and Tsay, 2003)。
除了Thr101这个位点,在低氮条件下,NRT1.1的第492位的脯氨酸在NO3–信号转导中也起着非常重要的作用 (Bouguyon et al., 2015)。
Thr101的磷酸化状态又是怎么被调控的呢?
NRT1.1上Thr101的磷酸化状态受蛋白激酶CIPK23调控。当外界NO3–的浓度低于0.2mM时,能显著诱导蛋白磷酸酶CIPK23的表达,CIPK23与CBL1/CBL9互作使Thr101磷酸化,进而使NRT1.1表现为高亲和NO3–转运蛋白活性。当植物处于高浓度的硝酸盐环境时,上述磷酸化过程被抑制,NRT1.1蛋白表现出低亲和性NO3–转运蛋白活性 (Ho et al., 2009)。
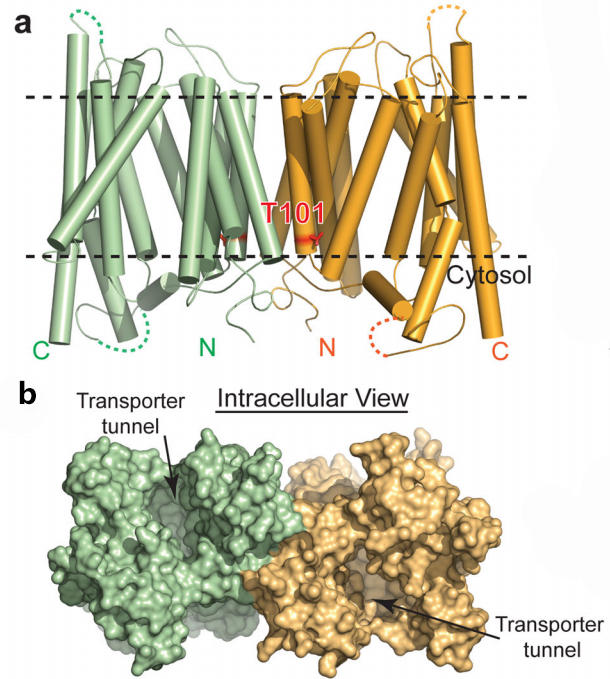
后来,通过对NRT1.1蛋白的结构进行解析 (Parker and Newstead, 2014; Sun et al., 2014),对上述磷酸化调控机制做了更进一步的解释(图4、图5)。
图4 NRT1.1蛋白的二聚体 (Sun et al., 2014)。研究发现,两个相邻的NRT1.1分子以并列的方式并列放置。(a)使用圆柱表示NRT1.1二聚体;(b)NRT1.1二聚体的正交视图。
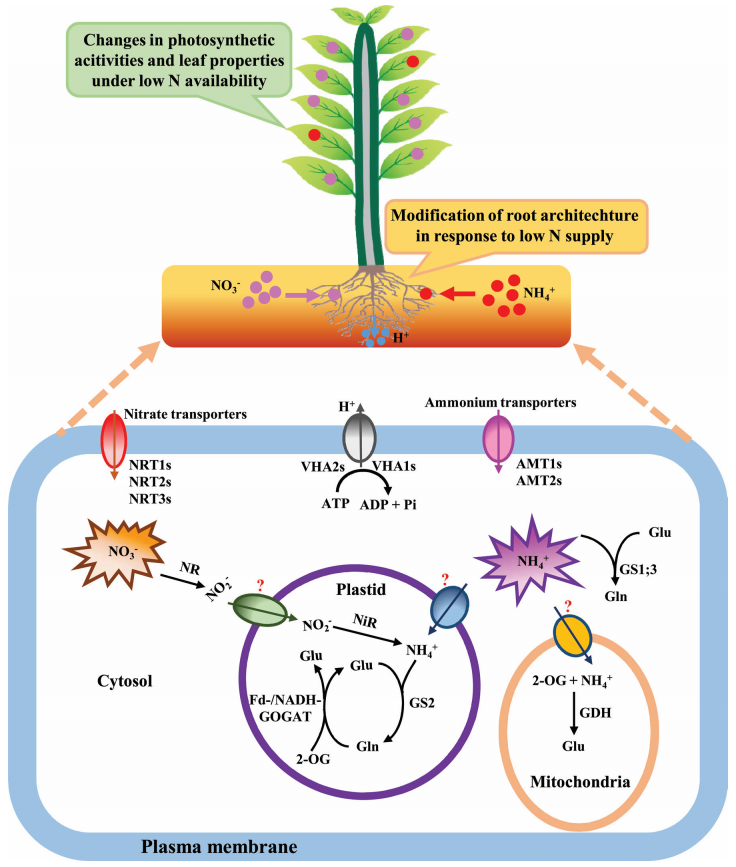
对NRT1.1双亲和的调控能够使植物利用大幅度浓度范围的硝酸盐,使植物能更好的适应外界NO3–浓度的变化,避免外界硝酸盐过多或过少对植物造成的不良影响。
在2009年之前的研究中发现,NRT1.1参与了硝酸盐的信号转导过程,比如,其抑制AtNRT2.1、刺激ANR1调节侧根生长、抑制L-Glu诱导的根系构型调节、刺激休眠种子的萌发等 (Ho et al., 2009),这些缺陷是由于硝酸盐的摄取减少,从而导致硝酸盐与未知传感器的可及性降低,还是NRT1.1本身就作为硝酸盐的信号传感器导致的呢?
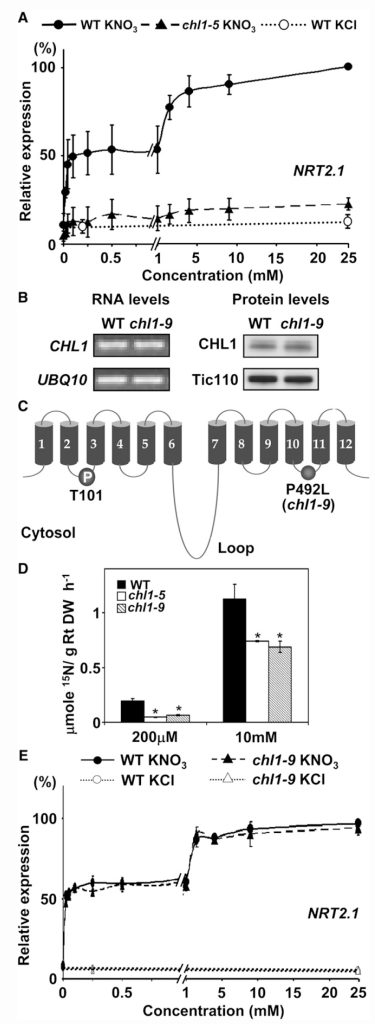
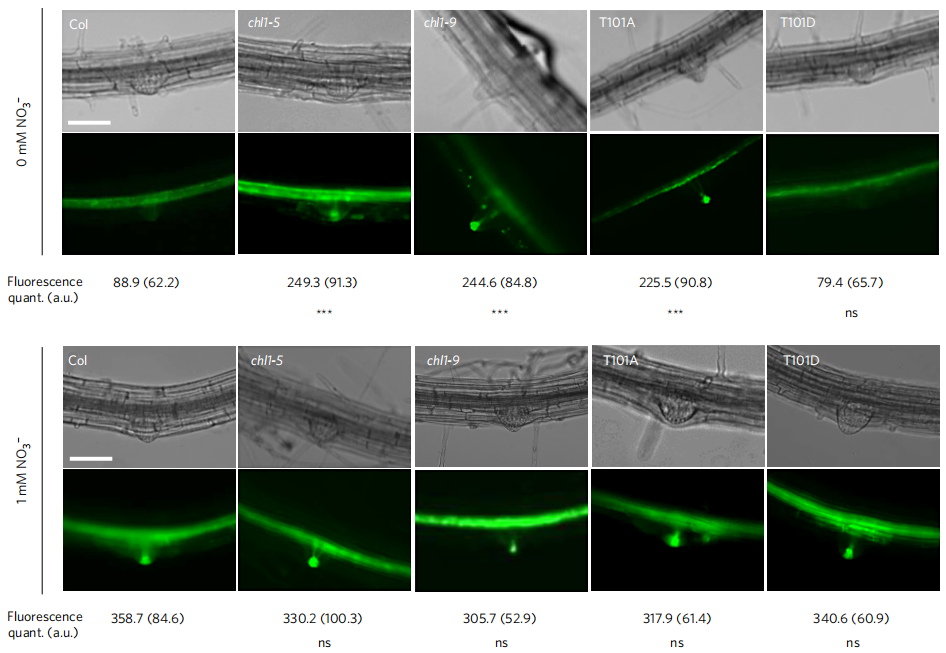
Tsay实验室分析了两种不同的nrt1.1突变体——chl1-9和chl1-5,chl1-9突变体仅丧失了NO3–吸收的功能,chl1-5同时丧失了NO3–吸收和响应的功能,将chl1-9基因组片段转入chl1-5中,响应功能可以恢复,但吸收功能无法恢复。这说明NRT1.1的吸收功能和信号调控功能是相互独立的,其信号功能不需要其必须具备转运活性。在chl1-5中,初级硝酸盐响应(Primary nitrate response, PNR)水平降低到野生型的30%,表明CHL1参与了对硝酸盐的感知(图6) (Ho et al., 2009)。
图6 chl1-9突变体在硝酸盐吸收方面存在缺陷,但可以表现出正常的初级硝酸盐反应 (Ho et al., 2009)。(A)chl1-5突变体的初级硝酸盐反应存在缺陷;(B)chl1-9突变体与野生型植株中CHL1基因的mRNA和CHL1蛋白的表达情况;(C)chl1-9突变体的突变位置,CHL1第492位的脯氨酸突变成了亮氨酸;(D)chl1-9突变体在硝酸盐吸收方面存在缺陷;(E)检测chl1-9突变体的初级硝酸盐反应,结果显示其与野生型无显著差别。
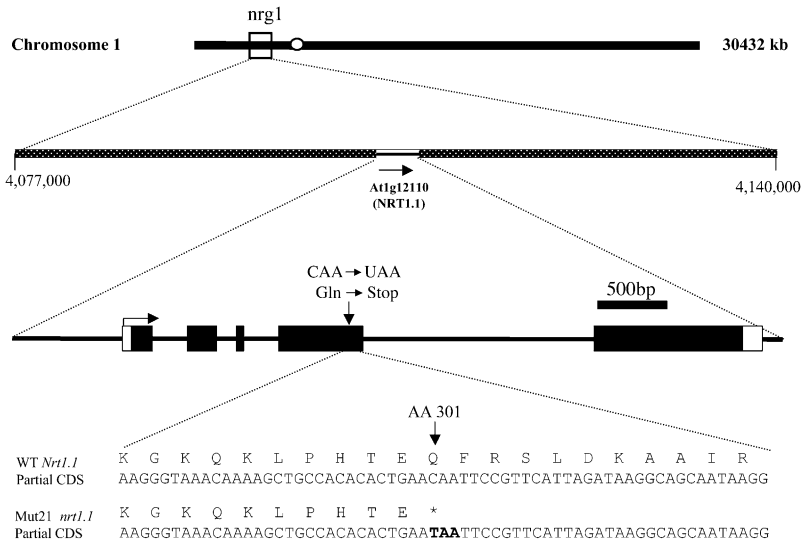
在同一年,Crawford实验室利用正向遗传学方法,找到一个在NO3–响应方面存在缺陷的突变体,通过图位克隆,发现该突变体的突变位点位于NRT1.1基因上(图7) (Wang et al., 2009)。
在植物中除了NRT1.1外,是否还存在其他的硝酸盐感受器呢?Ho等人推测还有其他的硝酸盐感受器,因为在不同生长条件下,chl1突变体的初级硝酸盐响应减少的幅度在50%-85%之间变化 (Ho et al., 2009)。去年,这方面有重要的研究进展,不同于以往认为NRT1.1是主要的硝酸盐感受器,Liu等认为NLP7为主要的硝酸盐感受器 (Liu et al., 2022),大家感兴趣的话可以好好读一下这篇Science期刊文章哈。
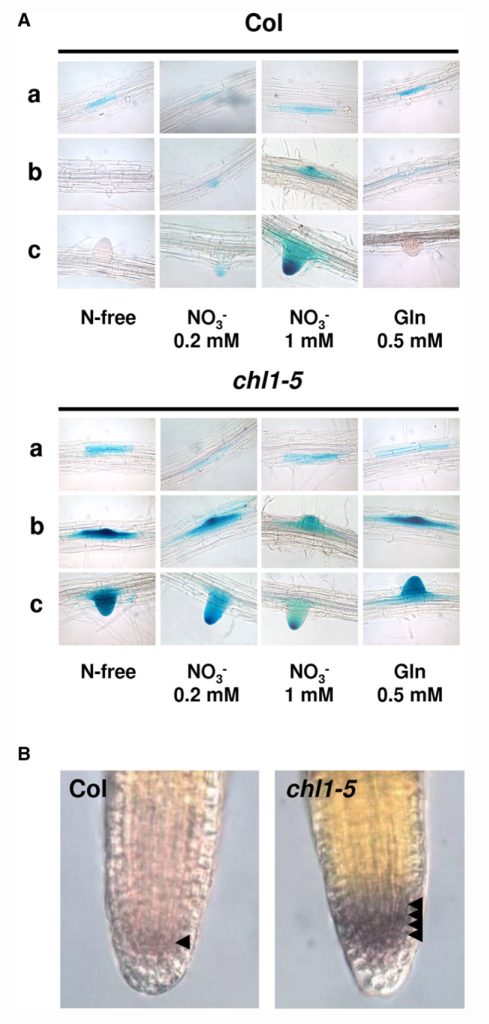
图8 NRT1.1的突变导致硝酸盐依赖的生长素在侧根位置的积累情况 (Krouk et al., 2010)。(A)将DR5::GUS转入野生型和chl1-5中,在不同时期对侧根进行GUS染色:a、侧根原基起始,b、侧根原基出现之前,c、侧根出现;这些植株在图中所示的氮源培养基上培养了8d;(B)在野生型和chl1-5植株中使用免疫定位的方法检测IAA。黑色箭头为IAA信号。
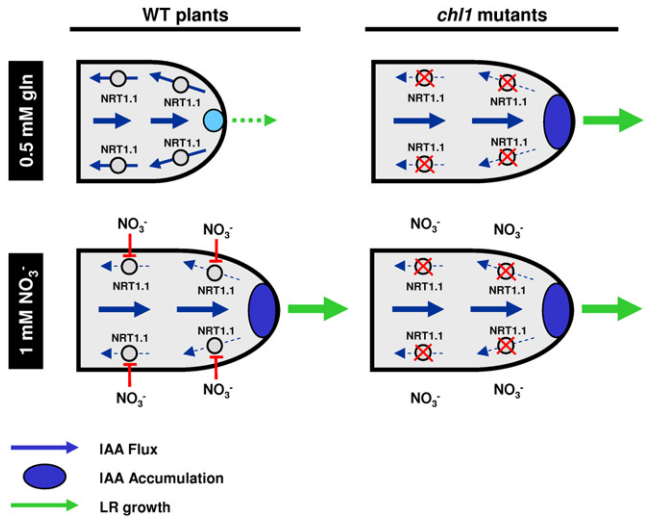
图9 NRT1.1影响侧根生长的模型 (Krouk et al., 2010)。对野生型植株来说,无硝酸盐存在时(以0.5mM谷氨酰胺Gln为氮源),NRT1.1在侧根中基向运输生长素,阻止了生长素在侧根根尖的积累,减缓了侧根的生长和伸长。当硝酸盐存在时,NRT1.1对生长素的运输被抑制,使生长素在侧根根尖积累,加速了侧根的生长。在chl1突变体中,由于NRT1.1的突变导致其无法基向运输生长素,无论外界氮源为何,生长素都会在侧根根尖积累,加速侧根的生长。
对上述现象的进一步研究表明,NRT1.1的T101位点的磷酸化对于其生长素转运活性是必需的(图10) (Bouguyon et al., 2015)。
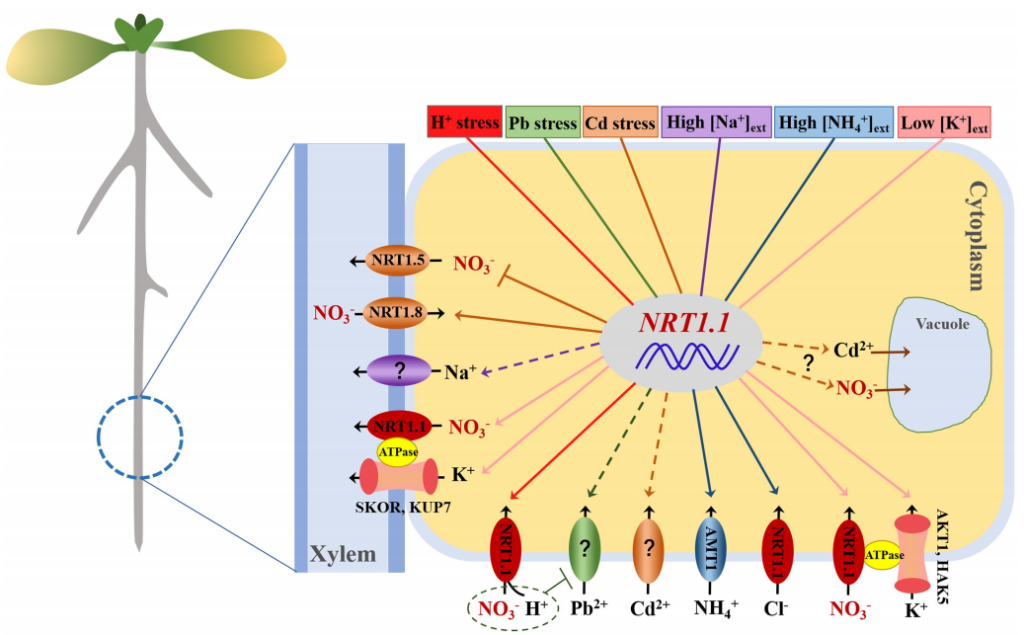
以干旱为例,NRT1.1基因可以控制叶片中气孔的开放程度,在nrt1.1突变体中,保卫细胞中硝酸盐含量降低,无法产生硝酸盐引起的去极化,导致气孔开放程度降低,从而使蒸腾作用减弱,最终使植株抗旱。这表明抑制NRT1.1介导的NO3–运输至保卫细胞中可能会更有利于植物应对干旱胁迫。干旱会促进脱落酸(ABA)的产生,而很多研究发现CIPK23参与ABA反应,所以未来的研究要集中在将ABA与NRT1.1联系起来的分子机制 (Fang et al., 2021)。
除了与抗旱有关,NRT1.1还与H+毒性、Na+积累、重金属胁迫、低K+、高NH4+等非生物胁迫响应有关,内容比较丰富,伯小远找了半天,发现综述“NRT1.1 Dual-Affinity Nitrate Transport/Signalling and its Roles in Plant Abiotic Stress Resistance”里关于硝酸盐非生物胁迫介绍的挺详细的,如果大家感兴趣的话,可以仔细读一下这篇文献喔。
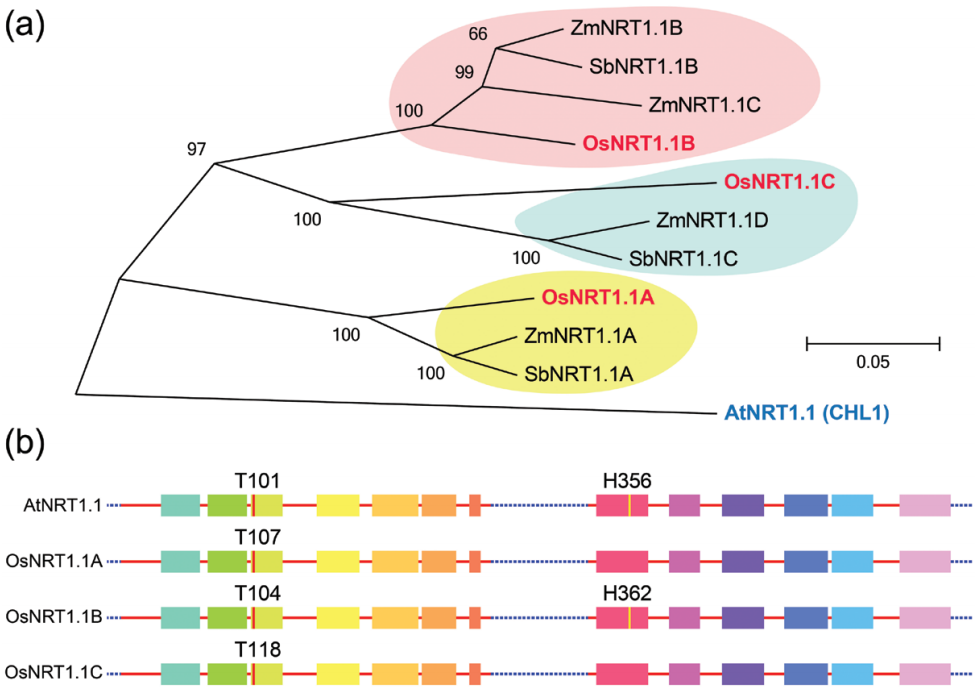
在拟南芥中只有1个NRT1.1基因,而在水稻中存在AtNRT1.1的三个同源基因,分别被命名为OsNRT1.1A、OsNRT1.1B和OsNRT1.1C 。OsNRT1.1A和OsNRT1.1B具有明显的功能差异,质膜定位的OsNRT1.1B是拟南芥AtNRT1.1的功能同源蛋白,也介导硝酸盐信号转导,通过钙依赖性或钙不依赖性途径将硝酸盐信号传导到下游硝酸盐反应,简单来说,它可能负责感知环境中的氮以触发下游的硝酸盐反应,而液泡膜定位的OsNRT1.1A可能感知细胞内的氮状态以微调氮代谢过程(图12) (Wang et al., 2020)。
在水稻中存在粳稻和籼稻两个亚种。对OsNRT1.1B这个基因来说,它在粳稻和籼稻中出现了分化,NRT1.1B基因的第980位碱基在粳稻等位基因NRT1.1B-japonic(粳稻)中是一个C,而在NRT1.1B-indica(籼稻)中为T,从而使NRT1.1B蛋白的第327位氨基酸在粳稻和籼稻中分别为苏氨酸和蛋氨酸。这个SNP位点的差别使籼稻拥有更好的氮利用效率(NUE) (Hu et al., 2015)。将NRT1.1B-indica引入到粳稻品种可以提高粳稻品种的NUE以及产量。
讲到这里,大家再看到文献中不熟悉的NRT1.1基因时,就大致可以明白是怎么回事了,其实就是在不同物种里对NRT1.1基因的命名,如果在该物种中其同源基因比较多,就会用字母或者数字再进行区分。
最后,伯小远斗胆改写了实业家张謇的一句话:天之生人也,与草木无异,若遗留一二有用科学,与草木同生,即不与草木同腐朽。和大家共勉!新的一年又开始啦,祝大家都能为科研做出不菲的贡献,让我们一起加油吧!
Bouguyon E, Brun F, Meynard D, et al. Multiple mechanisms of nitrate sensing by Arabidopsis nitrate transceptor NRT1.1. Nat Plants. 2015;1:15015. Published 2015 Mar 2. doi:10.1038/nplants.2015.15
Crawford NM, Glass ADM. Molecular and physiological aspects of nitrate uptake in plants.Treands in plant science. 1998; 3(10): 389-395. doi.org/10.1016/S1360-1385(98)01311-9
Doddema, H., Hofstra. J., and Feenstra, W. Uptake of nitrate by mutants of Arabidopsis fhaliana, disturbed in uptake or reduction of nitrate. I. Effect of nitrogen source during growth on uptake of nitrate and chlorate. Physiologia Plantarum. 1978; 43, 343-350. doi.org/10.1111/j.1399-3054.1978.tb01592.x
Doddema, H. and Telkamp, G.P. Uptake of Nitrate by Mutants of Arabidopsis thaliana, Disturbed in Uptake or Reduction of Nitrate. Physiologia Plantarum. 1979; 45: 332-338. doi.org/10.1111/j.1399-3054.1979.tb02593.x
Hu B, Wang W, Ou S, et al. Variation in NRT1.1B contributes to nitrate-use divergence between rice subspecies. Nat Genet. 2015;47(7):834-838. doi:10.1038/ng.3337
Huang NC, Chiang CS, Crawford NM, Tsay YF. CHL1 encodes a component of the low-affinity nitrate uptake system in Arabidopsis and shows cell type-specific expression in roots. Plant Cell. 1996;8(12):2183-2191. doi:10.1105/tpc.8.12.2183
Ho CH, Lin SH, Hu HC, Tsay YF. CHL1 functions as a nitrate sensor in plants. Cell. 2009;138(6):1184-1194. doi:10.1016/j.cell.2009.07.004
Lauter FR, Ninnemann O, Bucher M, Riesmeier JW, Frommer WB. Preferential expression of an ammonium transporter and of two putative nitrate transporters in root hairs of tomato. Proc Natl Acad Sci U S A. 1996;93(15):8139-8144. doi:10.1073/pnas.93.15.8139
Liu KH, Huang CY, Tsay YF. CHL1 is a dual-affinity nitrate transporter of Arabidopsis involved in multiple phases of nitrate uptake. Plant Cell. 1999;11(5):865-874. doi:10.1105/tpc.11.5.865
Liu KH, Tsay YF. Switching between the two action modes of the dual-affinity nitrate transporter CHL1 by phosphorylation. EMBO J. 2003;22(5):1005-1013. doi:10.1093/emboj/cdg118
Liu KH, Liu M, Lin Z, et al. NIN-like protein 7 transcription factor is a plant nitrate sensor. Science. 2022;377(6613):1419-1425. doi:10.1126/science.add1104
Luo J, Li H, Liu T, Polle A, Peng C, Luo ZB. Nitrogen metabolism of two contrasting poplar species during acclimation to limiting nitrogen availability. J Exp Bot. 2013;64(14):4207-4224. doi:10.1093/jxb/ert234
Krapp A, David LC, Chardin C, et al. Nitrate transport and signalling in Arabidopsis. J Exp Bot. 2014;65(3):789-798. doi:10.1093/jxb/eru001
Krouk G, Lacombe B, Bielach A, et al. Nitrate-regulated auxin transport by NRT1.1 defines a mechanism for nutrient sensing in plants. Dev Cell. 2010;18(6):927-937. doi:10.1016/j.devcel.2010.05.008
Parker JL, Newstead S. Molecular basis of nitrate uptake by the plant nitrate transporter NRT1.1. Nature. 2014;507(7490):68-72. doi:10.1038/nature13116
Siddiqi MY, Glass AD, Ruth TJ, Rufty TW. Studies of the Uptake of Nitrate in Barley: I. Kinetics of NO(3) Influx. Plant Physiol. 1990;93(4):1426-1432. doi:10.1104/pp.93.4.1426
Sun J, Bankston JR, Payandeh J, Hinds TR, Zagotta WN, Zheng N. Crystal structure of the plant dual-affinity nitrate transporter NRT1.1. Nature. 2014;507(7490):73-77. doi:10.1038/nature13074
Tsay YF, Schroeder JI, Feldmann KA, Crawford NM. The herbicide sensitivity gene CHL1 of Arabidopsis encodes a nitrate-inducible nitrate transporter. Cell. 1993;72(5):705-713. doi:10.1016/0092-8674(93)90399-b
Wang R, Xing X, Wang Y, Tran A, Crawford NM. A genetic screen for nitrate regulatory mutants captures the nitrate transporter gene NRT1.1. Plant Physiol. 2009;151(1):472-478. doi:10.1104/pp.109.140434
Wang W, Hu B, Li A, Chu C. NRT1.1s in plants: functions beyond nitrate transport. J Exp Bot. 2020;71(15):4373-4379. doi:10.1093/jxb/erz554