预测sgRNA的有效性通常是通过网站计算工具进行的。这些计算工具预测与植物基因组编辑的实验结果会存在一些不同(Naim et al., 2020)。因此,使用所选sgRNAs构建的CRISPR/Cas9载体的编辑效率需要在原生质体(Brandt et al., 2020; Shan et al., 2014; Wang et al., 2016)或根癌农杆菌介导的毛状根(Do et al., 2019; Zheng et al., 2020)等实验系统中进行测试。
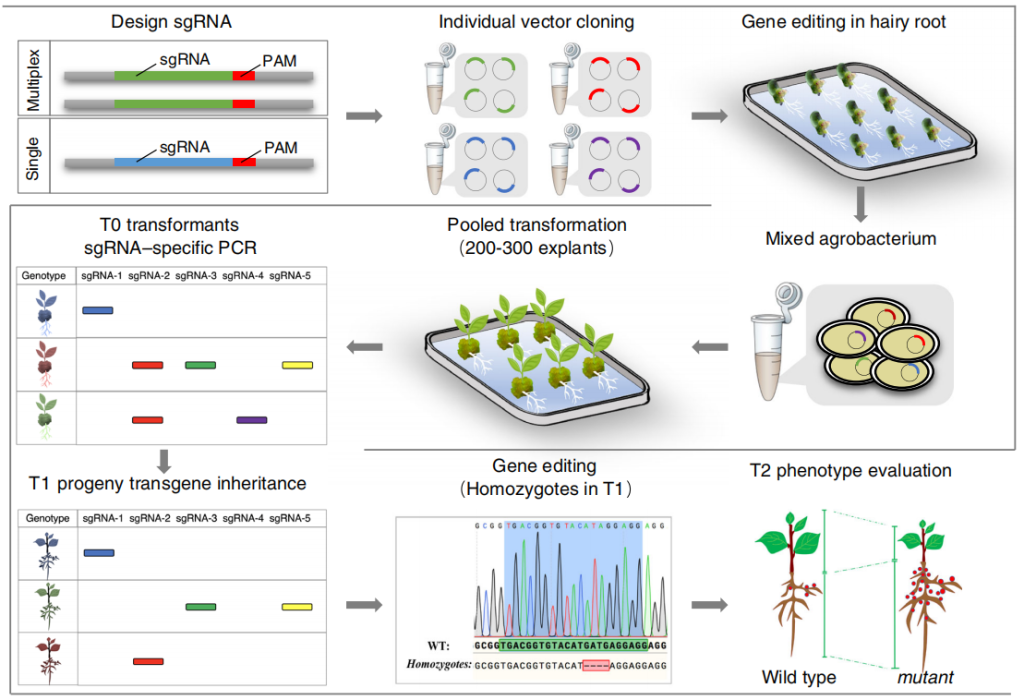
大豆转化效率低下阻碍了CRISPR/Cas9工具在创制大豆突变体上的应用,为了优化这一工具,可以在sgRNA的基因编辑效率上进行优化,根据上面的介绍,对大豆sgRNA基因编辑效率的优化可以通过根癌农杆菌介导的毛状根体系,大豆中毛根体系的又分为两种:下胚轴毛根体系和离体毛根体系,这两种方法都可以用于检测大豆sgRNA的基因编辑效率。
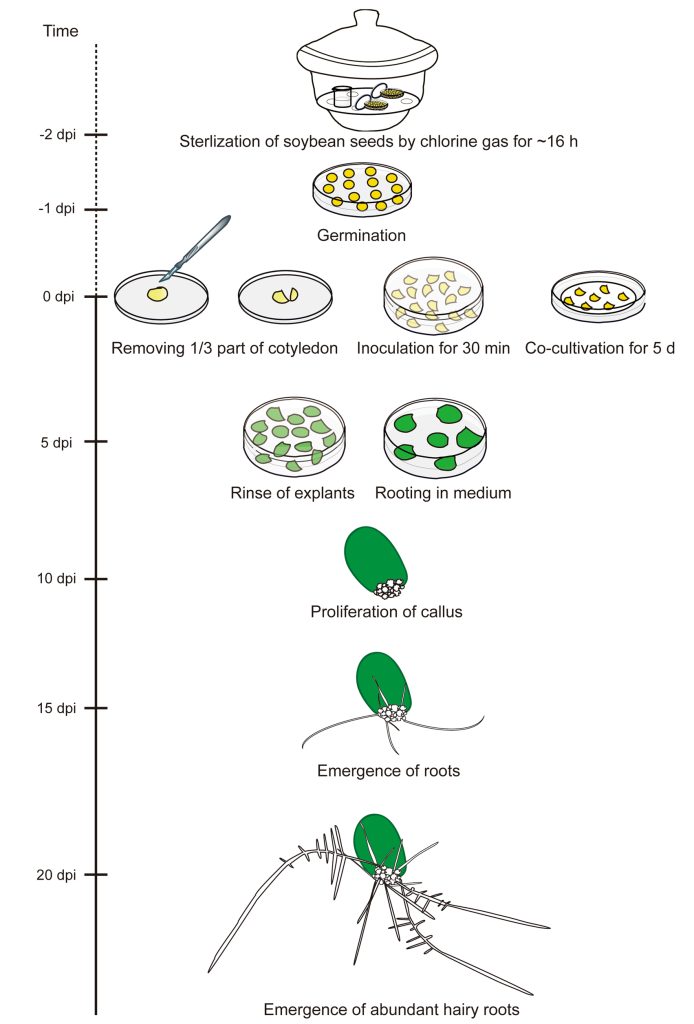
②倒去过氧化氢溶液,用过量的无菌水冲洗种子5-6次。
②将含有次氯酸盐的烧杯放入干燥器中,加入盐酸,然后立即盖上盖子,并用封口膜密封。
③将种子放在氯气中保存14-18h进行灭菌处理。
②用针尖取一滴细菌(图1b),把它放在要感染的部位,然后用针穿过膏体刺入植物(图1c)。确保针穿过下胚轴的中心部分。
③用针把更多的细菌膏塞进伤口。
②将菌悬液吸进注射器,刺入子叶节和下胚轴三次(图1c)。确保针穿过下胚轴的中心部分。
③在伤口中注射1-2滴菌悬液。
从培养皿中挑取转有对应质粒的K599阳性单克隆菌落,转移到5mL含有对应抗生素的YEP液体培养基中。随后将培养基放在28°C,250rpm的摇床中孵育12小时。接下来,将1mL的YEP农杆菌培养液转移到100mL的YEP液体培养基中,在28℃下以250rmp的转速振荡培养,直到达到600nm(OD600)处的光密度为1.2。在接种大豆子叶之前,农杆菌培养液以3500rpm/min的转速离心10min,农杆菌在共培养(CC)培养基中重悬至OD600为0.6。对于CC培养基的配方,其液体CC培养基含有B5(Sigma),1/2 B5,或MS(Sigma),1/2 MS盐(添加0.5 g/L MES),2%蔗糖,1.67mg/L 6-BA,0.25mg/L GA3和40mg/L乙酰丁香酮(AS)(pH=5.4)。
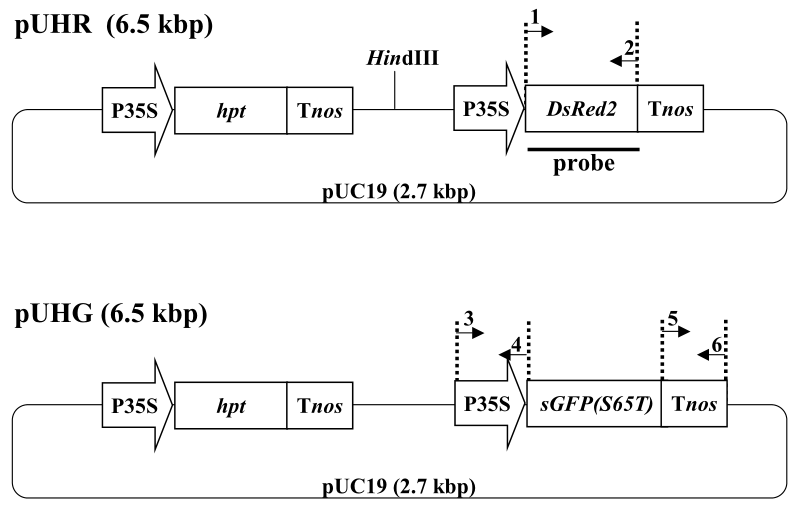
图4 质粒载体结构(Nishizawa et al., 2006)。pUHR和pUHR分别是带有红色荧光蛋白基因DsRed2和绿色荧光蛋白基因sGFP(S65T)的载体。
图6 含35S::AtMyb75转基因豆科植物毛状根中花青素的积累(Fan et al., 2020)。大豆PI377578(a);L. corniculaus(b);L. japonicus(c,d)和M. truncatula R108(e)中过表达的阳性毛状根是紫色或红色的。转基因阳性的根用箭头表示。bar=1cm
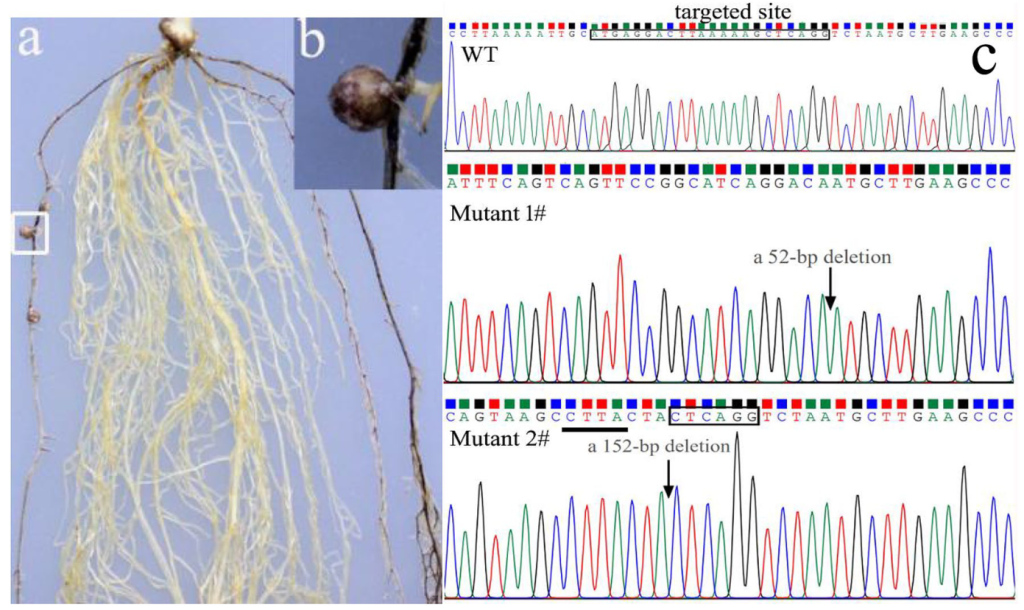
特异性根瘤菌可以与特定的豆科植物(或豆科植物的基因型)建立有效的共生关系,从而形成固氮瘤。例如,大豆PI377578可以防止根瘤菌USDA193结瘤,大豆中的Rfg1负责结瘤限制(未发表的数据)。为了进一步验证AtMyb75作为报告基因的可靠性,在USDA193接种的PI377578中用以AtMyb75作为报告基因的CRISPR/Cas9系统敲除Rfg1。转基因根只含有双等位纯合或杂合的Rfg1突变体才能形成根瘤。如预期的那样,成熟的固氮根瘤在产生花青素的紫色/红色转基因根上成功形成(图7a, b)。在非转基因根上没有形成结节(图7a)。转基因根瘤经PCR分析和测序确认(图7c)。
为了评估毛状根过表达AtMyb75是否能诱导不同大豆品种花青素积累,研究者分析了86个大豆基因型。86个大豆品种中有39个积累了可视化的花青素,发现在紫色下胚轴的大豆品种中过表达AtMyb75,转基因根中会积累花青素,而绿色下胚轴的大豆品种不能。这就需要对更多具有紫色和绿色下胚轴的不同基因型的大豆进行进一步的实验测试。随着花青素合成机制的阐明,有可能通过调控绿色下胚轴大豆中参与花青素合成的其他基因的表达积累花青素,并作为报告基因广泛应用于大豆的所有基因型(Fan et al., 2020)。
研究人员还在紫花品种ZH42对GmW1进行了基因编辑,基因编辑大豆植株T0出现了三种花色,通过分析T1突变体的表型和纯合型,发现得到的T0代白花植株为双等位纯合(图8j)。还发现了花的颜色和茎的颜色之间的对应关系,野生型Ws82的茎为绿色,花为白色,而Ws82中GmW1过表达植株的茎为紫色,花为紫色。在ZH42w1突变体中,茎色由紫色变为绿色,花色由紫色变为白色。紫色花植株的茎是紫色的,而白色花植株的茎是绿色的(图8k,l)。因此无需等待开花,就可以通过幼苗的茎的颜色快速识别转基因植株。
综上,在这个新的报告系统中,颜色强度可以作为过表达转基因植株表达水平的反映。过表达GmW1的植物中发现了浅粉色、粉色或紫色的颜色,GmW1表达水平越高,颜色越深。虽然存在颜色过弱而无法识别的可能性,但最好是选择表达相对较强的转基因植物进行研究,表达过弱的转基因植物应该被淘汰。对于基因编辑载体,将35S::GmW1表达元件整合到CRISPR载体中,由于编辑植株会产生三类花色情况,仅通过花或茎的颜色不能直接确定纯合和杂合。但通过分析花或茎的颜色可以筛选出纯合突变体(Chen et al., 2022)。
但该研究并没有构建GmW1为报告基因的载体进行其他基因过表达和基因编辑的测试,GmW1作为报告基因是否会影响其他基因的过表达或编辑还未可知。而且该研究仅测试了Jack、Ws82和ZH42三种品种,观察花和茎的颜色是否对于大多数大豆品种都适用还需要研究。
抗性基因为报告基因的检测方法
该研究中植株除草剂抗性检测,具体做法如下:首先在野生型大豆上涂抹不同浓度的草丁膦,从而确定大豆的草丁膦适宜鉴定浓度为135mg/L。同时对T0代转化体和野生型植株的一半叶片涂抹135mg/L草丁膦。1周后,转基因植株维持正常状态(图9B),而野生型大豆叶片开始变黄(图9A);一段时间后,野生型植株整株都枯黄,而转基因植株基本没有发生变化(图10)。为了验证转基因表型的稳定性,对T1代的PCR检测阳性植株依然进行草丁膦检测,得到与T0代相似的结果(图9C,D)。为了进一步明确转基因植株的抗除草剂能力,对草丁膦抗性植株的叶片继续喷施草甘膦,部分植株依然维持抗性(图9F)。由此可以初步确定已将EPSPS基因及Bar基因成功的转入大豆植株中(刘志敏等, 2013)。
图9 野生型大豆与转基因大豆除草剂抗性检测(刘志敏等, 2013)。A:WT;B:T0叶片涂抹草丁膦;C:WT;D:T1叶片涂抹草丁膦;E:WT;F:T1叶片喷施草甘膦。
References:
刘志敏等. 农杆菌介导的大豆萌发种子转化技术[J]. 生物技术通报. 2013, 08:83-87.
茆少星. 转基因产品四种快检方法比较与快检方法评价模型构建[D]. 2021, 南京师范大学,MA thesis.
钱雪艳等. 农杆菌介导大豆子叶节转化cry1Iem基因[J]. 中国油料作物学报 2016, 06:757-763.
Bai M, Yuan J, Kuang H, et al. Generation of a multiplex mutagenesis population via pooled CRISPR‐Cas9 in soya bean[J]. Plant Biotechnology Journal, 2020, 18(3): 721-731.
Brandt K M, Gunn H, Moretti N, et al. A streamlined protocol for wheat (Triticum aestivum) protoplast isolation and transformation with CRISPR-Cas ribonucleoprotein complexes[J]. Frontiers in Plant Science, 2020, 11: 769.
Cheng Y, Wang X, Cao L, et al. Highly efficient Agrobacterium rhizogenes-mediated hairy root transformation for gene functional and gene editing analysis in soybean[J]. Plant Methods, 2021, 17(1): 1-12.
Chen L, Yuan S, Cai Y, et al. See the color, see the seed: GmW1 as a visual reporter for transgene and genome editing in soybean[J]. The Crop Journal, 2022, 2214-5141.
Di R, Purcell V, Collins G B, et al. Production of transgenic soybean lines expressing the bean pod mottle virus coat protein precursor gene[J]. Plant Cell Reports, 1996, 15(10): 746-750.
Do P T, Nguyen C X, Bui H T, et al. Demonstration of highly efficient dual gRNA CRISPR/Cas9 editing of the homeologous GmFAD2–1A and GmFAD2–1B genes to yield a high oleic, low linoleic and α-linolenic acid phenotype in soybean[J]. BMC plant biology, 2019, 19(1): 1-14.
Fan Y, Wang X, Li H, et al. Anthocyanin, a novel and user-friendly reporter for convenient, non-destructive, low cost, directly visual selection of transgenic hairy roots in the study of rhizobia-legume symbiosis[J]. Plant Methods, 2020, 16:94.
Kereszt A, Li D, Indrasumunar A, et al. Agrobacterium rhizogenes-mediated transformation of soybean to study root biology[J]. Nature protocols, 2007, 2(4): 948-952.
Naim F, Shand K, Hayashi S, et al. Are the current gRNA ranking prediction algorithms useful for genome editing in plants?[J]. PLoS One, 2020, 15(1): e0227994.
Shan Q, Wang Y, Li J, et al. Genome editing in rice and wheat using the CRISPR/Cas system[J]. Nature protocols, 2014, 9(10): 2395-2410.
Nishizawa K, Kita Y, Kitayama M, et al. A red fluorescent protein, DsRed2, as a visual reporter for transient expression and stable transformation in soybean[J]. Plant Cell Rep, 2006, 25:1355-1361.
Wang W, Akhunova A, Chao S, et al. Optimizing multiplex CRISPR/Cas9-based genome editing for wheat[J]. BioRxiv, 2016: 051342.
Zheng N, Li T, Dittman J D, et al. CRISPR/Cas9-based gene editing using egg cell-specific promoters in Arabidopsis and soybean[J]. Frontiers in plant science, 2020, 11: 800.