了解核内染色体折叠的机制对于确定基因组结构与功能之间的关系至关重要。近年来,诸如高通量染色体构象捕获(Hi-C)等技术的进步使我们能够获得全基因组范围的三维(3D)基因组数据。经过深入研究,人们发现了动物基因组在细胞核中的三维结构,从低分辨率到高分辨率分别为染色体区域,染色质区室,拓扑关联域(TAD),染色质环和染色质丝。在哺乳动物中,基因组有着独特的TAD结构,并且TAD的特征在不同物种中显着保守。顺式作用元件(例如增强子、抑制子和绝缘子)通常位于它们调节的基因启动子的远端,并且这种调控关系表现出强烈的组织特异性。染色质的三维构象可能通过影响这些顺式作用元件和基因之间相互作用来精确的调控基因转录控制。在过去的十年,已经出现了许多技术去检测全基因组或者部分片段的染色质互作。最早的技术路线(Chromatin Conformation Capture简称3C),只能研究一个位点对另外一个位点的相互作用(一对一),而后又发展出了4C(一对全),5C(多对多),Hi-C(全对全),Capture-C(多重一对一)等技术。但在少量细胞中定量检测染色质互作的方法还未曾出现。
近日,斯德哥尔摩卡罗林斯卡大学附属医院Anita Göndör团队在Nature Protocols上发表题为“The Nodewalk assay to quantitate chromatin fiber interactomes in very small cell populations”的方法论文。该文章提出了一种Nodewalk的实验方法,可以仅仅使用300个细胞且高灵敏度的检测全基因组范围的染色质互作,使其具有检测活体组织、肿瘤细胞,转换期细胞的能力。
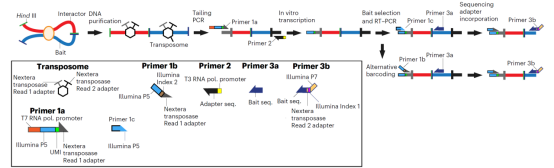
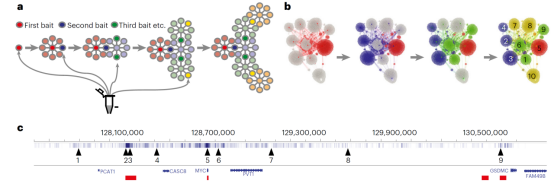
Nodewalk实验时基于最初的3C方法修改而来(图1)。细胞与新鲜制备的1%甲醛(FA)溶液而不是福尔马林交联,为确保获得最佳的和可重复的结果。交联的染色质DNA用一种限制性内切酶(例如Hind III)消化至接近完全(平均达到90%)(图2a),然后有效的进行粘性末端的连接。在去交联和DNA纯化之后,使用Nextera转座酶进行随机的DNA切割,能够消除PCR重复,并连接接头寡核苷酸。而后使用Ampure XP珠子进行纯化,此步骤产生平均大小为500 bp的片段(图2b)。然后通过尾部PCR将T7聚合酶启动子序列与Illumina P5序列和唯一分子标识符(UMI)结合(五个循环,可以根据输入样本的大小增加)(图1)。为了捕获3C产物,通过体外转录将连接的3C DNA转化为3C RNA,进而使3C序列扩增1000 倍,然后使用位于所选区域的限制酶位点附近的引物进行逆转录(图1)。最后,在使用配备有P7序列的相同引物针对已经包含P5序列的cDNA添加Illumina 序列接头后,进行Illumina测序(图1)。该实验的一个重要特点是在连接步骤中包含了交联和消化的果蝇染色质,这使估计背景分子间的连接事件(intermolecular ligation events)的成为可能。由于连接过程中染色质的浓度较低,分子间的连接事的频率通常小于总reads的1%,突出了分子内连接事件的富集。最后,从少量的3C DNA以线性方式生成大量的RNA中间体,使评估染色质纤维相互作用成为可能(图3a到c)。这就使得Nodewalk具有高分辨率和无与伦比的灵敏度,可以全面和定量地检测非常小细胞群(300个细胞)中的染色质互作。
图3:Nodewalk的可视化概要图
文章来源:植物生物技术Pbj