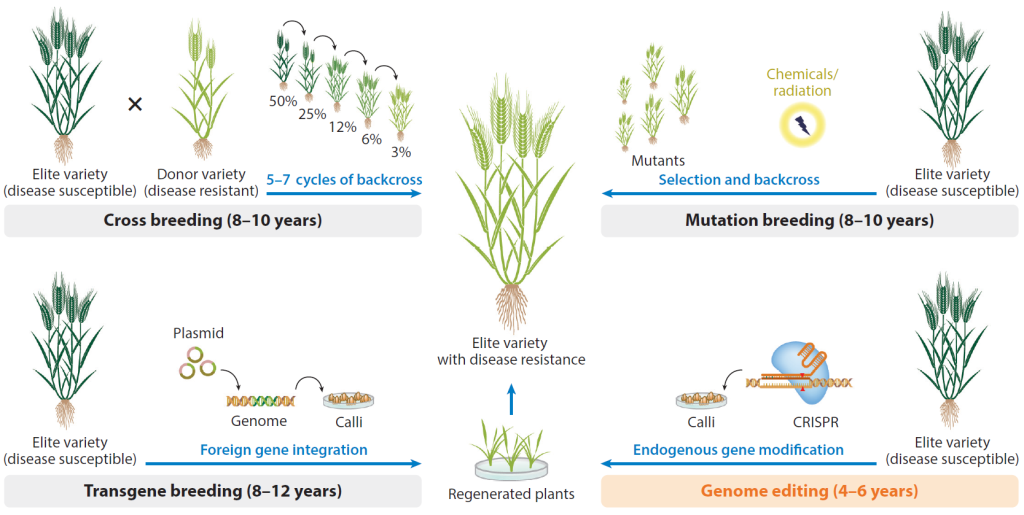
CRISPR/Cas9系统是目前使用最广泛的基因编辑技术,但该系统发挥作用都需要将携带有Cas9-sgRNA以及转基因筛选标记的表达元件通过农杆菌介导等方式导入到受体植物材料中,会将表达元件整合到其基因组上,而非整合元件还可能在植物细胞中传递,造成诸如脱靶、染色体损伤等较为严重的副作用。这影响了该技术的实际应用,同时可能引发的生物安全问题也是限制其商业化的主要因素。尽管插入的外源基因可以通过自交或者回交的手段,从编辑后代植株染色体中分离去除,但该过程费时费力,对于一些倍性复杂、育种周期长或无性繁殖的植物更是如此。因此,建立高效的非转基因(DNA-free)植物基因组编辑技术对于推动基因编辑作物的商业化具有重要的意义。
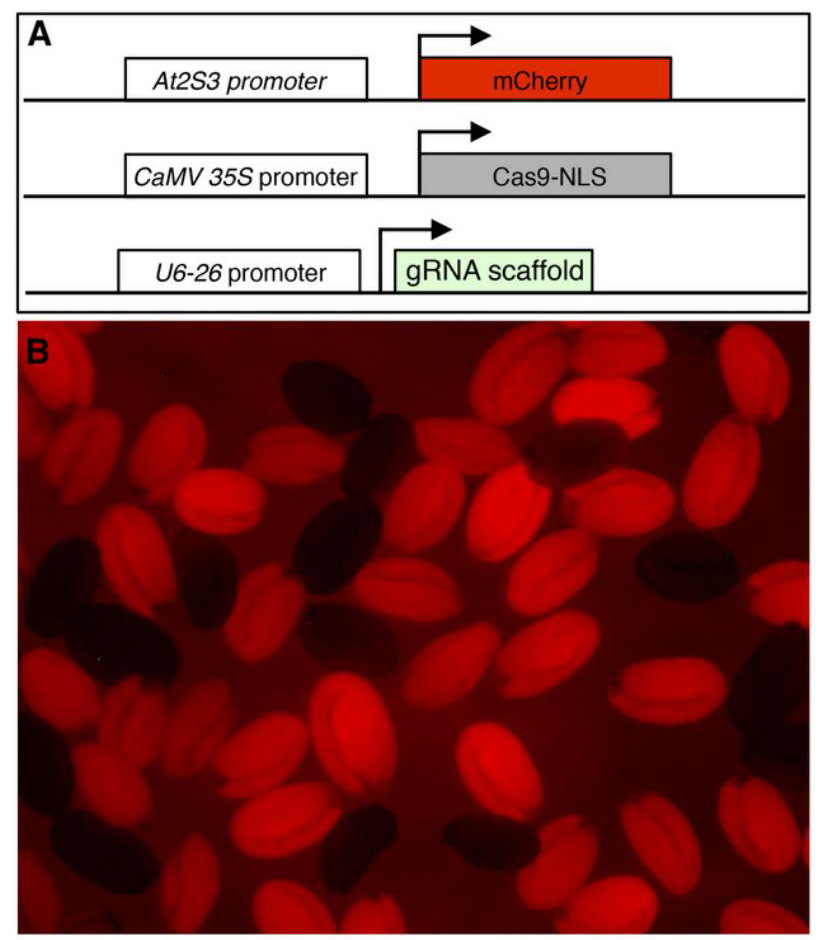
为了从基因编辑作物的子代中筛选出DNA-free的编辑作物,赵云德教授团队构建了带有mcherry荧光蛋白标签的CRISPR/Cas9载体,这样不含有CRISPR/Cas9表达元件的种子就不产生红色荧光,可以利用显微镜对DNA-free的T1代种子进行视觉识别(图3)。这种荧光标记的方法节省了植物生长、基因组DNA提取和基因分型的时间,适用于建立高通量和自动化筛选系统(Gao et al., 2016)。
图3 (A)包含mCherry荧光蛋白表达元件的CRISPR/Cas9载体的示意图。(B)在显微镜下看到的T1代种子,其中DNA-free的种子不产生红色荧光(Gao et al., 2016)。
案例二:
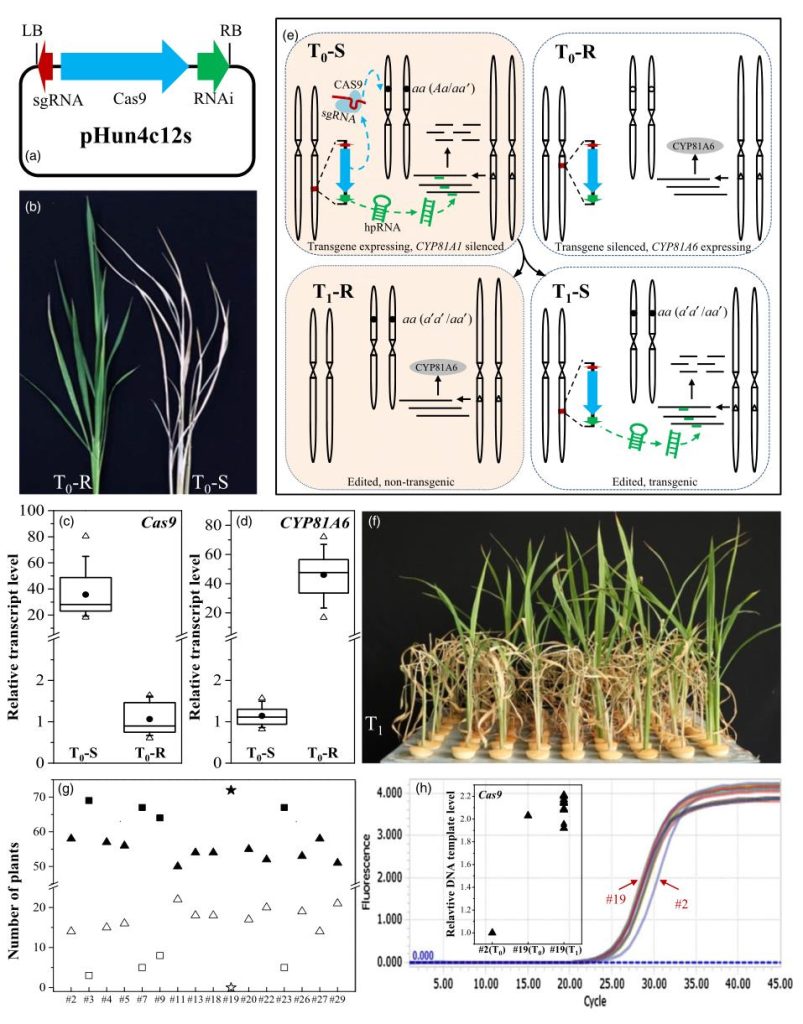
在水稻中,CYP81A6能够编码一种P450细胞色素蛋白,使其对苯达松除草剂具有抗性。而沉默CYP81A6会使水稻植株对苯达松敏感。浙江大学舒庆尧教授团队开发了一种基于RNA干扰(RNAi)的策略,称为CRISPR-S。研究人员将CYP81A6-hpRNAi表达元件添加到CRISPR/Cas9结构中,可获得对苯达松敏感的转基因植物。研究人员向T1植物喷施苯达松后,转基因T1幼苗开始脱水或死亡。相比之下,DNA-free的T1幼苗能够正常生长(图4f)(Lu et al., 2017)。
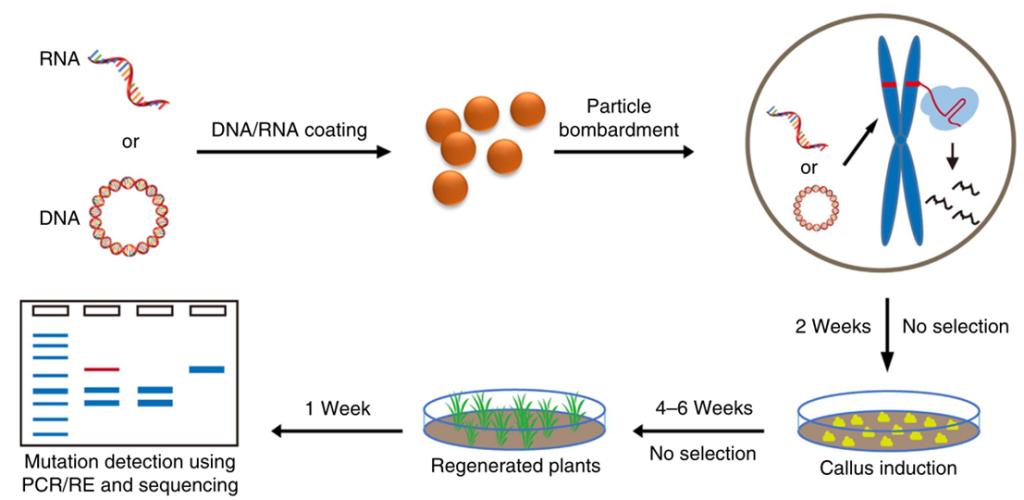
由于环状质粒或RNA,不易被整合进植物基因组中,中科院高彩霞团队和邱金龙团队将CRISPR/Cas9质粒DNA(TECCDNA)或其转录的RNA(TECCRNA)通过基因枪法直接转入小麦胚中(图6)(Zhang et al., 2016)。在瞬时表达和发挥作用后,质粒DNA或RNA会被细胞内源核酸酶分解,从而实现DNA-free的基因编辑,尤其是TECCRNA,在整个基因组编辑过程中不涉及外源DNA。研究人员对该系统在小麦基因组中的定向编辑效果进行了测试。通过对4个小麦品种的7个内源基因(共9个靶位点)靶向编辑发现,TECCDNA在小麦T0转基因植株中编辑效率为1.0%-9.5%,DNA-free基因编辑植株的比例为43.8%-86.8%。TECCRNA的基因编辑效率仅为1.1%,但基因编辑的植株均不含转基因。对突变体植物中32个潜在脱靶位点进行分析,发现通过瞬时表达CRISPR/Cas9 DNA或RNA获得的突变体中均未发生脱靶效应,证明这两种方法具有更高的特异性。此外,该方法简单、成本低,无需进行繁重的抗性筛选。
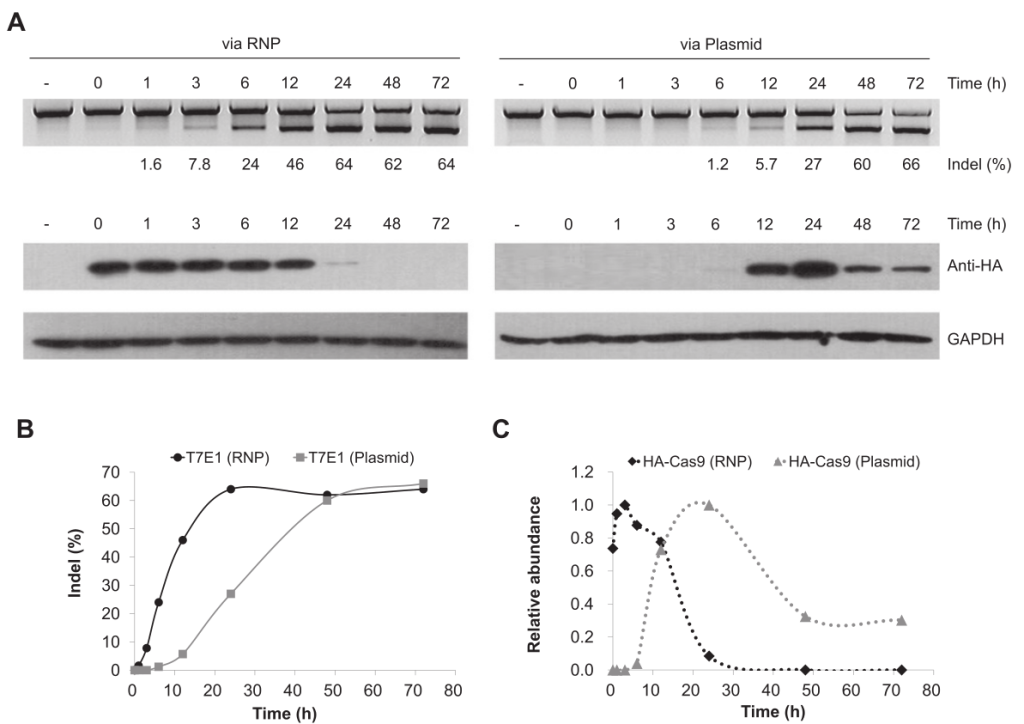
图7 RNPs或质粒转染的基因组编辑的T7E1分析和Western blotting(Kim et al., 2014)。T7E1分析表明,RNPs进入细胞后就会作用于染色体上的DNA靶标,并且在电转染后一天内基因突变水平就达到了平台期。相比之下,通过质粒转染的基因编辑需要三天时间才能达到相同的突变水平。Western blotting分析显示,Cas9蛋白会在细胞中迅速降解,转染后24小时,几乎检测不到Cas9。相比之下,质粒转染方式的Cas9蛋白在细胞内持续表达了几天。
RNPs没有天然的细胞穿透活性,需要辅助递送进生物体中。在动物细胞中,RNPs转染细胞的方法主要是阳离子脂质载体运输法和电转染法。而植物中有PEG-Ca2+法、基因枪法等。RNPs递送的受体也可以是原生质体、胚胎细胞或合子。不同递送方法、不同受体形式或物种会导致不同的基因编辑效率。
案例一:
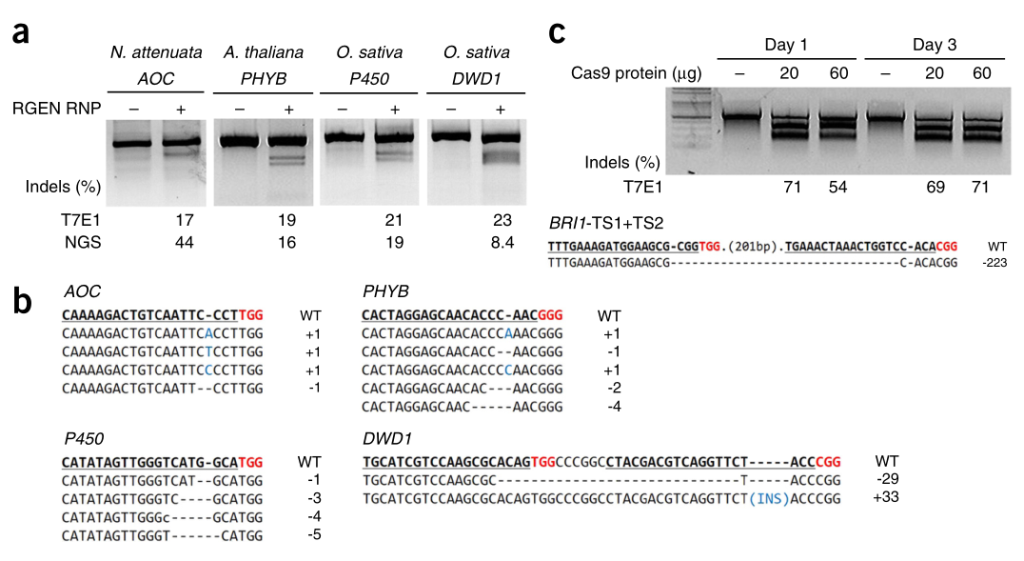
Woo等人将RNPs通过PEG-Ca2+介导的方式直接递送至拟南芥、烟草、莴苣和水稻的植物原生质体中(图8),实现了植物基因组编辑,并以高达46%的效率获得了DNA-free基因编辑植株(Woo et al., 2015)。
图8 在烟草、拟南芥和水稻植物原生质体中RGEN RNPs编辑系统的编辑情况(Woo et al., 2015)。(a)T7E1分析和靶向深度测序测定的基因突变效率。(b)植物细胞中RGEN RNPs诱导的突变DNA序列。(c)拟南芥原生质体BRI1基因被编辑的时间过程分析。
案例二:
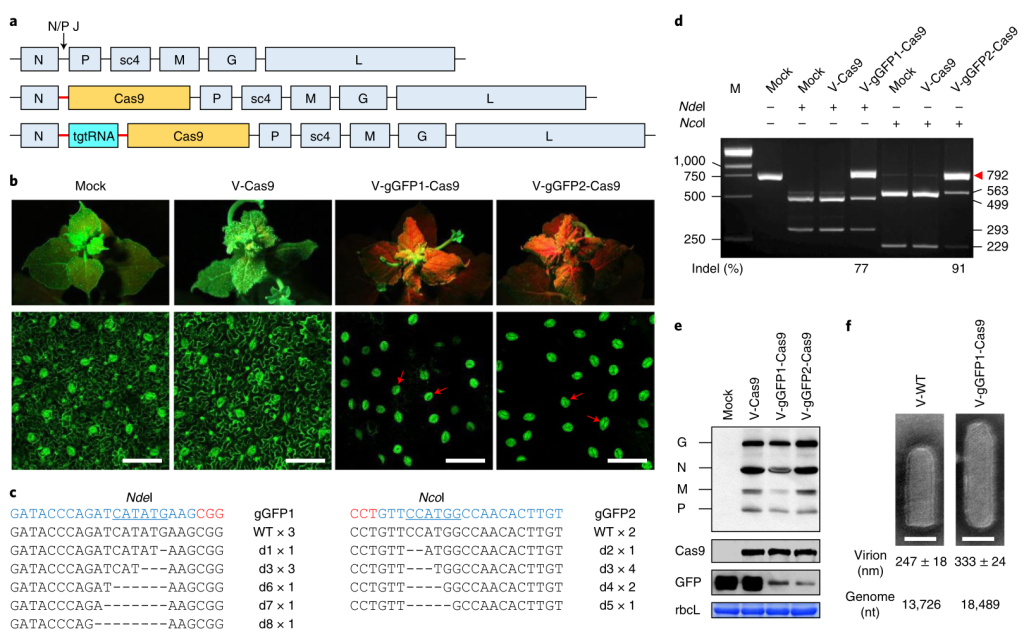
日本理化研究所(RIKEN)的Erika Toda等人以体外受精的合子为RNPs递送的受体来进行基因编辑,这在被子植物中是首次报道。研究人员首先利用电融合的方式使获得的水稻精细胞和卵细胞完成受精,在融合1小时内通过PEG-Ca2+介导的方式,将预先组装好的Cas9-sgRNA RNPs递送至合子胚中(图9)。通过组织培养并在没有筛选剂的情况下可获得全程DNA-free且经过基因编辑的目的植株,获得理想株系的效率在14-64%之间(Toda et al., 2019)。
2020年6月,美国农业部提出了对基因编辑技术的支持性指南,认为无外源基因导入的基因编辑作物无需监管,采用类似政策的国家有加拿大、巴西、阿根廷、澳大利亚、智利、哥伦比亚、以色列等。而欧盟对转基因作物采取了较为保守的态度,倾向于将基因编辑作物等同于转基因作物,但这一裁定,在科学界和行业内引起了轩然大波,大家纷纷表示这将阻碍欧洲基因编辑研究和产业化发展,要求欧盟放宽基因编辑产品的产业化。2022年1月24日,我国农业农村部制定公布了《农业用基因编辑植物安全评价指南(试行)》,其中对于未引入外源基因的基因编辑植物的安全监管,科研人员需要依据在环境安全和食用安全方面可能产生的风险申报安全评价。
虽然每个国家都在根据自己的国情、研发进展等对基因编辑作物制定不同的监管政策,但是大多还是倾向于采取产品导向的监管政策,即根据产品的具体情况来决定是否采取监管措施。DNA-free植物基因组编辑技术由于不会引入外源基因,在国际和国内得到了越来越多的认可。获得更加高效、适用性广、投入低的DNA-free基因组编辑技术,有助于加速作物育种进程、降低监管成本、推动基因编辑作物的产业化应用,我们任重而道远。
References:
Chen K, Wang Y, Zhang R, Zhang H, Gao C. 2019. CRISPR/Cas Genome Editing and Precision Plant Breeding in Agriculture. Annu Rev Plant Biol 70:667-697.
Gao C. 2021. Genome engineering for crop improvement and future agriculture. Cell 184:1621-1635.
Gao X, Chen J, Dai X, Zhang D, Zhao Y. 2016. An Effective Strategy for Reliably Isolating Heritable and Cas9-Free Arabidopsis Mutants Generated by CRISPR/Cas9-Mediated Genome Editing. Plant Physiol 171:1794-1800.
He Y, Zhao Y. 2019. Technological breakthroughs in generating transgene-free and genetically stable CRISPR-edited plants. aBIOTECH 1:88-96.
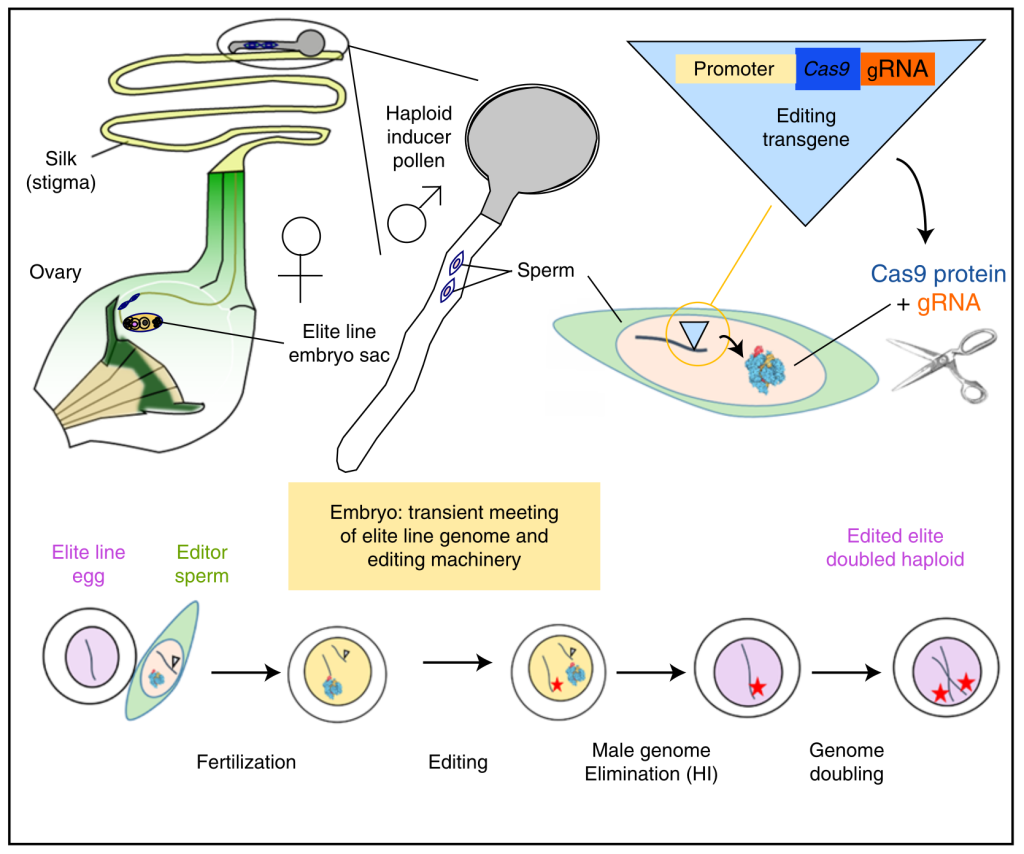
Kelliher T, Dakota Starr, Xiujuan Su, Guozhu Tang, Zhongying Chen. 2019. One-step genome editing of elite crop germplasm during haploid induction. Nat Biotechnol 37:287-292.
Kim S, Kim D, Cho SW, Kim J, Kim JS. 2014. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res 24:1012-1019.
Lu HP, Liu SM, Xu SL, Chen WY, Zhou X, Tan YY, Huang JZ, Shu QY. 2017. CRISPR-S: an active interference element for a rapid and inexpensive selection of genome-edited, transgene-free rice plants. Plant Biotechnol J 15:1371-1373.
Ma X, Zhang X, Liu H, Li Z. 2020. Highly efficient DNA-free plant genome editing using virally delivered CRISPR-Cas9. Nat Plants 6:773-779.
Manghwar H, Li B, Ding X, Hussain A, Lindsey K, Zhang X, Jin S. 2020. CRISPR/Cas Systems in Genome Editing: Methodologies and Tools for sgRNA Design, Off-Target Evaluation, and Strategies to Mitigate Off-Target Effects. Adv Sci (Weinh) 7:1902312.
Toda E, Koiso N, Takebayashi A, Ichikawa M, Kiba T, Osakabe K, Osakabe Y, Sakakibara H, Kato N, Okamoto T. 2019. An efficient DNA- and selectable-marker-free genome-editing system using zygotes in rice. Nat Plants 5:363-368.
Woo JW, Kim J, Kwon SI, Corvalan C, Cho SW, Kim H, Kim SG, Kim ST, Choe S, Kim JS. 2015. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins. Nat Biotechnol 33:1162-1164.
Zhang Y, Liang Z, Zong Y, Wang Y, Liu J, Chen K, Qiu JL, Gao C. 2016. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA. Nat Commun 7:12617.