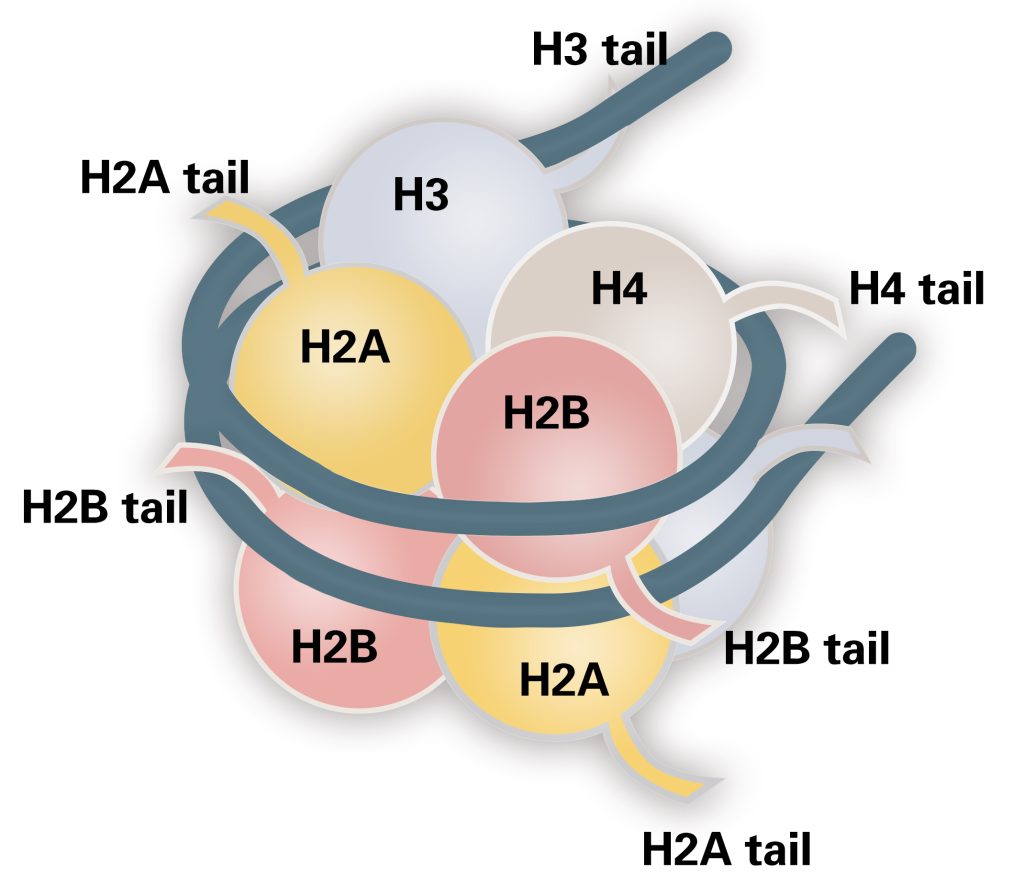
图1 组蛋白结构。
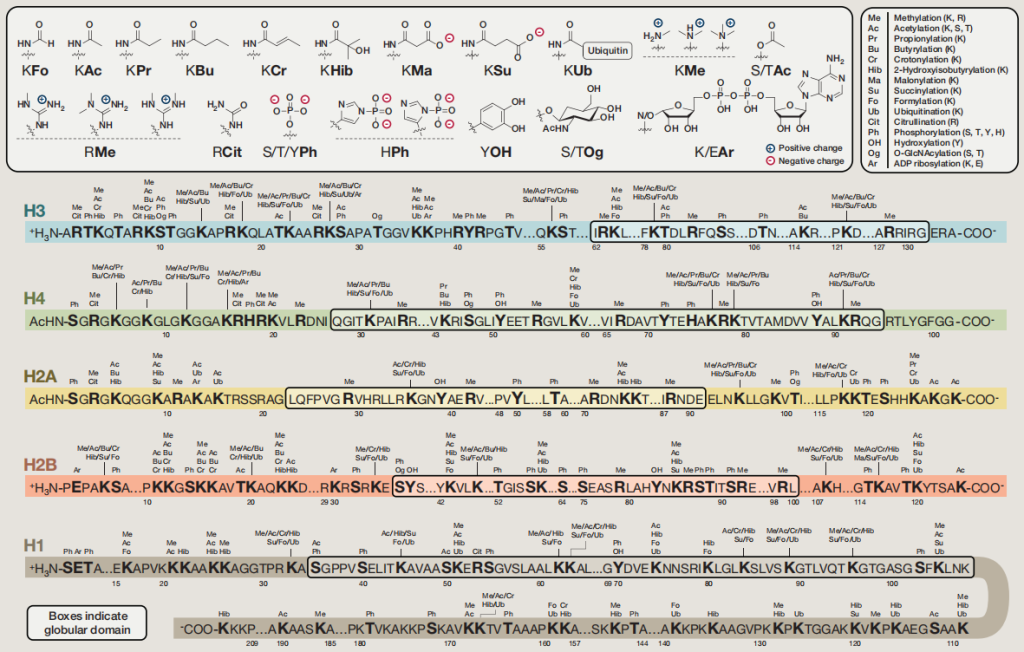
常见的组蛋白修饰包括甲基化、乙酰化、磷酸化、泛素化等,图2展示了各个组蛋白中的修饰情况(Huang et al., 2014)。组蛋白发生修饰后会使染色质的构象等发生变化,从而影响了DNA与组蛋白之间的相互作用,进而调控基因的转录表达。
由于组蛋白修饰的类型众多,因此在描述组蛋白修饰时一般按照“组蛋白名称 + 氨基酸名称 + 氨基酸位置 + 修饰类型”的规则进行描述,例如:H3K4me1和H3K27ac,其具体的含义在前面的文章中已经介绍过了,这里就不再解释了。
下面我们以组蛋白乙酰化和甲基化为例,浅谈一下ChIP-seq技术在这方面的应用,通过上面的介绍,大家也看到了组蛋白的修饰类型非常多,想要通过几篇文献就了解清楚是不太可能的,所以大家除了看小远给大家列举的文献之外还需要自己去下功夫哦!
在真核细胞中,组蛋白受到大量的翻译后修饰,其依次或组合作用会影响染色质的结构和基因组功能。在“H3K36ac Is an Evolutionary Conserved Plant Histone Modification That Marks Active Genes”一文中,作者发现组蛋白H3的lys36位点乙酰化(H3K36ac)是植物中一个新的染色质修饰位点。H3K36ac修饰在种子植物中是进化保守的,包括裸子植物挪威云杉(Picea abies)、被子植物水稻(Oryza sativa)、烟草(Nicotiana tabacum)和拟南芥(Arabidopsis thaliana)。在拟南芥中,H3K36ac在常染色质中富集,而在异染色质中不富集。全基因组染色质免疫沉淀测序实验(ChIP-seq)显示,H3K36ac峰位于基因的5’端,主要位于转录起始位点远端的两个核小体上,与基因长度无关。
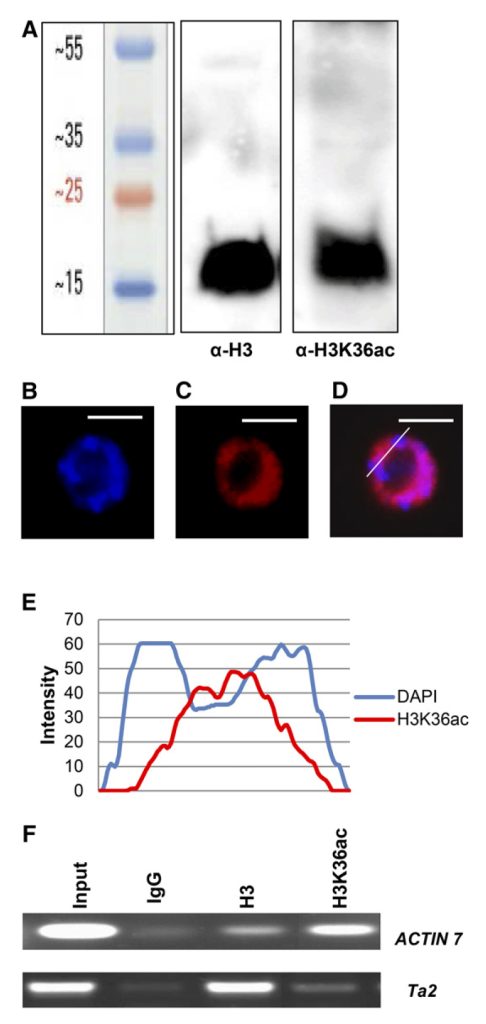
对于上面的这些结果,具体解析如下:作者首先利用质谱鉴定了lys36是新的乙酰化位点,接着为了验证和扩展质谱结果,作者利用PAGE分离拟南芥花序组蛋白提取物,并用抗H3K36ac的抗体进行探针检测。该抗体之前使用修饰和未修饰的多肽进行了斑点印迹和蛋白质免疫印迹的特异性测试,并通过ChIP-seq进行了验证(Morris et al., 2007;Egelhofer et al., 2011)。该抗体与11个不同的乙酰化组蛋白肽(H2AK5ac,H3K14ac,H3K18ac,H3K27ac,H3K4ac,H3K9ac,H4K12ac,H4K16ac,H4K5/8/12/16ac,H4K5ac和H4K8ac)以及许多未修饰或甲基化的组蛋白肽均无交叉反应。拟南芥组蛋白提取物的阳性信号(图3A)证实了植物中H3K36ac修饰的质谱鉴定结果。
利用抗H3K36ac抗体对拟南芥根尖细胞核进行免疫染色,以确定H3K36ac在拟南芥细胞核中的分布。H3K36ac在核仁和4′,6-二氨基-苯乙烯醇(DAPI)致密的染色体中心中没有荧光信号,染色体中心主要由着丝粒和着丝粒间重复序列组成。相比之下,H3K36ac染色可见于正染色区域(图3B-E),表明大多数H3K36ac与较少浓缩、基因丰富的区域以及可能的活性基因相关。为了验证这一预测,使用抗H3K36ac和抗H3的抗体进行甲醛交联ChIP(X-ChIP)。事实上,H3K36ac在转录的常染色质ACTIN7基因中含量较高,而在沉默的异染色质Ta2转座子中含量较低(图3F)。Ta2处H3信号的增加与早期报道一致,可能与异染色质中更致密的染色质结构有关(Rehrauer et al., 2010; Shu et al., 2012)。这些结果表明,H3K36ac存在于常染色质的活性基因上,而不存在于异染色质上。
图3 拟南芥中H3K36ac的检测(Mahrez et al., 2016)。(A)从拟南芥花序中提取的组蛋白在15%的SDS-PAGE凝胶上分离,然后使用抗H3和抗H3K36ac的抗体进行蛋白免疫印迹。(B,C)H3K36ac在根尖细胞核中的定位。利用DAPI(B)对拟南芥根尖间期细胞核进行染色,并用抗H3K36ac的抗体(C)进行免疫荧光分析。(D)DAPI和H3K36ac信号的叠加。图中的白线表示使用ImageJ软件进行分析通过细胞核的截面。(E)DAPI(蓝色)和H3K36ac(红色)荧光强度的定量线谱。(F)X-ChIP检测采用IgG、抗H3和抗H3K36ac的抗体,而引物是针对活性的ACTIN7基因和沉默的Ta2转座因子。
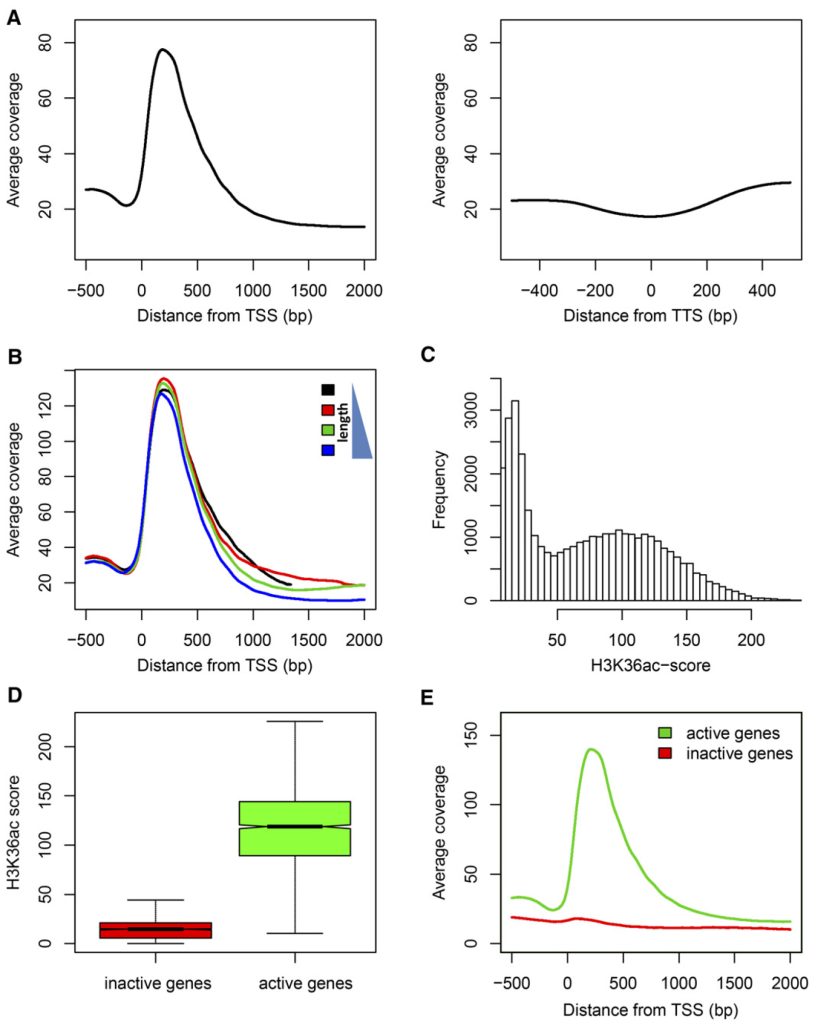
接着,作者进行了天然状态下的ChIP-seq实验,以确定H3K36ac在单核细胞分辨率下的全基因组定位。具体结果如下:
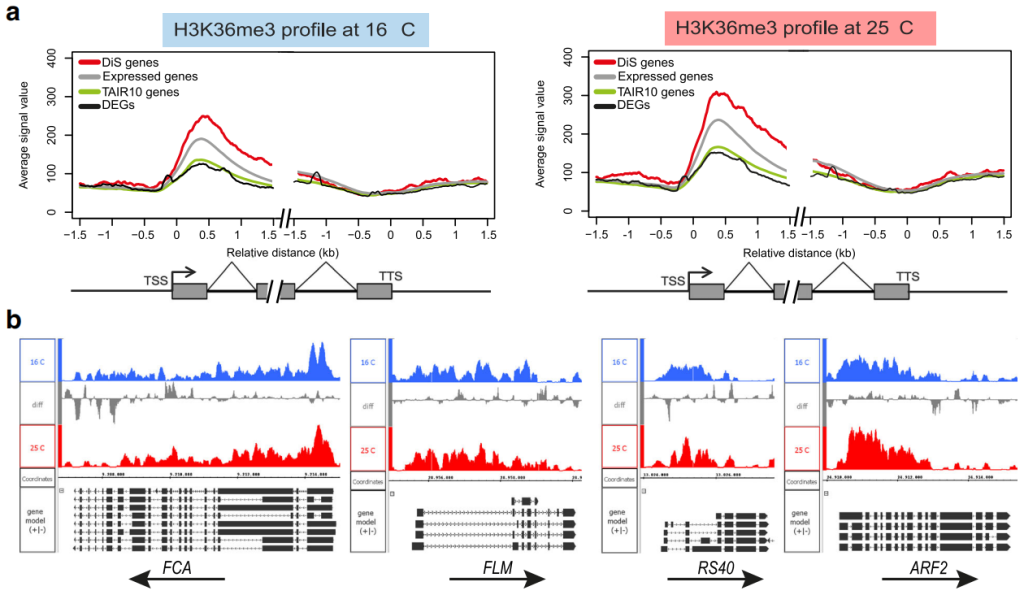
全球变暖严重影响了植物的开花时间和繁殖成功率。前信使RNA(pre-messenger RNA, pre-mRNA)的选择性剪接是植物环境温度调控反应的重要机制,但其具体的调控机制尚不清楚。在拟南芥中,温度升高通过改变关键调控基因的剪接,促进植物形态的变化以及从营养期到生殖期的转变。在“Histone H3 lysine 36 methylation affects temperature-induced alternative splicing and flowering in plants”一文中,作者研究了一种特定的组蛋白修饰是否会影响环境温度诱导的选择性剪接和开花时间。
作者采用全基因组方法,进行RNA测序(RNA-seq)分析和不同环境温度下植物的H3组蛋白36号赖氨酸三甲基化(H3K36me3)的染色质免疫沉淀测序(ChIP-seq)。对这些数据集的分析和比较表明,温度诱导的差异剪接基因在H3K36me3中富集。此外,作者发现H3K36me3沉积的减少会导致温度诱导的选择性剪接的改变。
最后的结果表明,组蛋白标记H3K36me3在剪接调控和植物对环境温度波动的可塑性方面发挥了关键作用。作者的发现为作物育种开辟了新的前景,从而更好地应对气候变化导致的环境变化。
宿主植物对病原体的识别导致快速的转录重排和防御反应的激活。许多防御调节因子的表达在此过程中被诱导,但它们在转录上如何被调控的机制在很大程度上是未知的。在“ChIP-seq reveals broad roles of SARD1 and CBP60g in regulating plant immunity”一文中,作者通过染色质免疫沉淀测序发现,转录因子SARD1和CBP60g与大量编码植物免疫关键调控因子的基因启动子区结合。具体结果如下:
在自身启动子下表达SARD1-HA融合蛋白的sard1 cpb60g突变株中,病原体诱导的ICS1表达恢复到与cbp60g单突变株相似的水平,表明SARD1-HA的功能与野生型SARD1蛋白类似。为了鉴定SARD1的靶向基因,作者利用抗HA的抗体在自身启动子下表达SARD1-HA融合蛋白的转基因植物上进行ChIP实验。免疫沉淀的DNA用Illumina测序。对ChIP测序(ChIP-seq)数据的分析显示了×20的基因组覆盖深度。
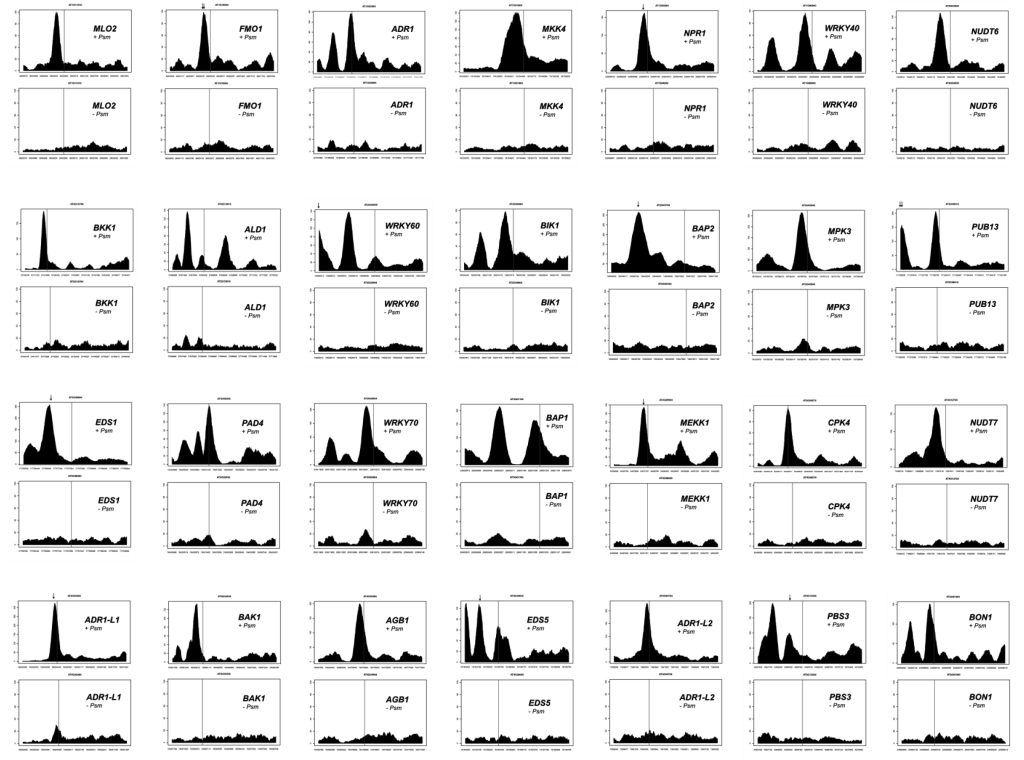
绘制基因组上每个位置的序列覆盖范围,以确定拟南芥基因组中的峰。基因区域的峰分析表明,大部分序列峰位于翻译起始位点上游的1.5kb区域,包括5′-UTRs和启动子区域。去除阴性对照中序列峰相似的基因后,在84个基因的内含子、60个基因的3′-UTRs和1902个基因翻译起始位点上游1.5kb区域中发现了高度≥90的峰。作者的分析重点是在翻译起始位点上游1.5kb区域包含高度为90或更高的峰的组,因为它包含许多编码已知的植物防御调控基因,这些基因被病原体感染强烈诱导(表1)。这些已知的防御调控因子的启动子和编码区序列reads的分布见图6。
图6 补充数据1(本文中没有展示,感兴趣的同学可以自己去下载补充数据1)中基因启动子和编码区序列reads的分布。纵轴表示序列读取的次数。横轴表示染色体上的位置。翻译起始位点的位置用读取行标记。靠近序列峰的GAAATTT基序的位置用箭头表示。+Psm,用P.s.m.浸润处理的SARD1-HA转基因植株的序列分布。ES4326,-Psm,未经处理的SARD1-HA转基因植物的序列分布。
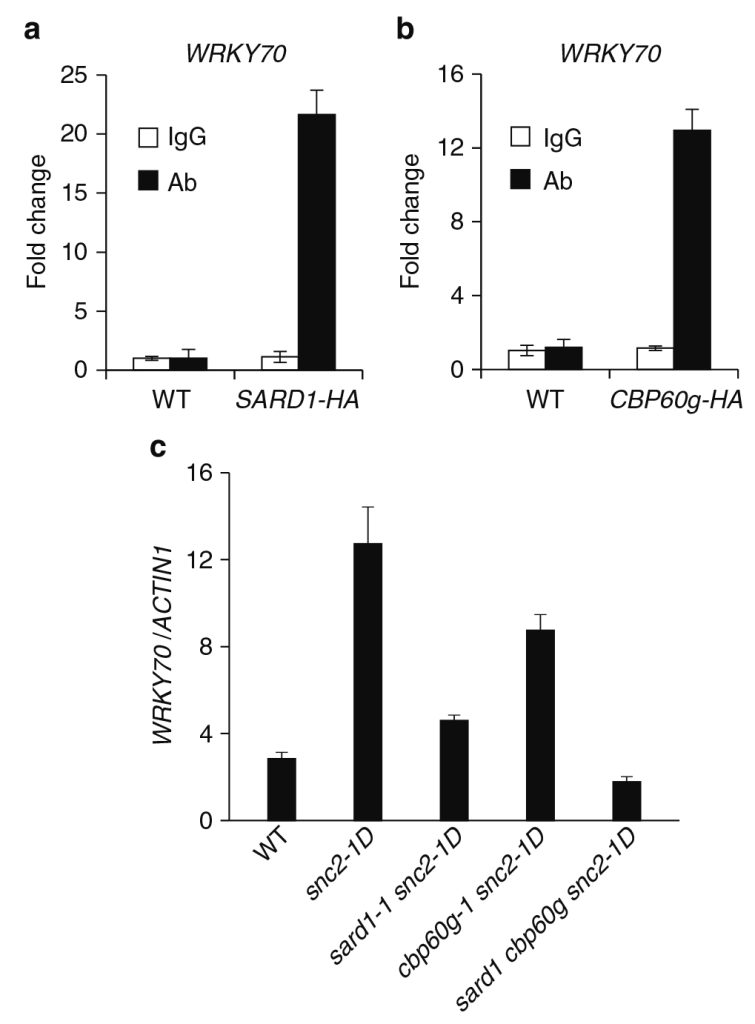
ChIP-seq鉴定的SARD1的候选靶基因之一是WRKY70(表1),它能够调节snc2-1D中水杨酸(Salicylic acid,SA)独立的防御反应(Zhang et al., 2010)。对抗HA抗体免疫沉淀的DNA进行定量PCR分析,证实WRKY70是SARD1的结合靶点(图7a)。在自身启动子表达CBP60g-HA融合蛋白的sard1cpb60g突变体植株中,病原体诱导的ICS1表达恢复到与sard1单突变体相似的水平,表明CBP60g-HA的功能与野生型蛋白相似。为了确定WRKY70是否也是CBP60g的结合靶点,作者使用抗HA的抗体在在其自身启动子下表达CBP60g-HA融合蛋白的转基因植物上进行了ChIP-PCR实验。如图7b所示,CBP60g也靶向WRKY70的启动子区域。
作者的研究为核小体占用和基序序列的共同作用提供了一个全面的评估,它们可能是控制植物基因表达的顺式元件。另外,作者的发现为进一步了解染色质状态对植物转录调控通路的影响铺平了道路。
References:
Egelhofer T A, Minoda A, Klugman S, et al. An assessment of histone-modification antibody quality[J]. Nature structural & molecular biology, 2011, 18(1): 91-93.
Huang H, Sabari B R, Garcia B A, et al. SnapShot: histone modifications[J]. Cell, 2014, 159(2): 458-458.
Kimura H. Histone modifications for human epigenome analysis[J]. Journal of human genetics, 2013, 58(7): 439-445.
Liu M J, Seddon A E, Tsai Z T Y, et al. Determinants of nucleosome positioning and their influence on plant gene expression[J]. Genome research, 2015, 25(8): 1182-1195.
Mahrez W, Arellano M S T, Moreno-Romero J, et al. H3K36ac is an evolutionary conserved plant histone modification that marks active genes[J]. Plant physiology, 2016, 170(3): 1566-1577.
Morris S A, Rao B, Garcia B A, et al. Identification of histone H3 lysine 36 acetylation as a highly conserved histone modification[J]. Journal of Biological Chemistry, 2007, 282(10): 7632-7640.
Pajoro A, Severing E, Angenent G C, et al. Histone H3 lysine 36 methylation affects temperature-induced alternative splicing and flowering in plants[J]. Genome biology, 2017, 18(1): 1-12.
Rehrauer H, Aquino C, Gruissem W, et al. AGRONOMICS1: a new resource for Arabidopsis transcriptome profiling[J]. Plant physiology, 2010, 152(2): 487-499.
Shu H, Wildhaber T, Siretskiy A, et al. Distinct modes of DNA accessibility in plant chromatin[J]. Nature Communications, 2012, 3(1): 1-9.
Sun T, Zhang Y, Li Y, et al. ChIP-seq reveals broad roles of SARD1 and CBP60g in regulating plant immunity[J]. Nature communications, 2015, 6(1): 1-12.
Torres-Perez J V, Irfan J, Febrianto M R, et al. Histone post-translational modifications as potential therapeutic targets for pain management[J]. Trends in Pharmacological Sciences, 2021, 42(11): 897-911.
Zhang Y, Yang Y, Fang B, et al. Arabidopsis snc2-1D activates receptor-like protein-mediated immunity transduced through WRKY70[J]. The Plant Cell, 2010, 22(9): 3153-3163.
| 官网链接:plant.biorun.com |