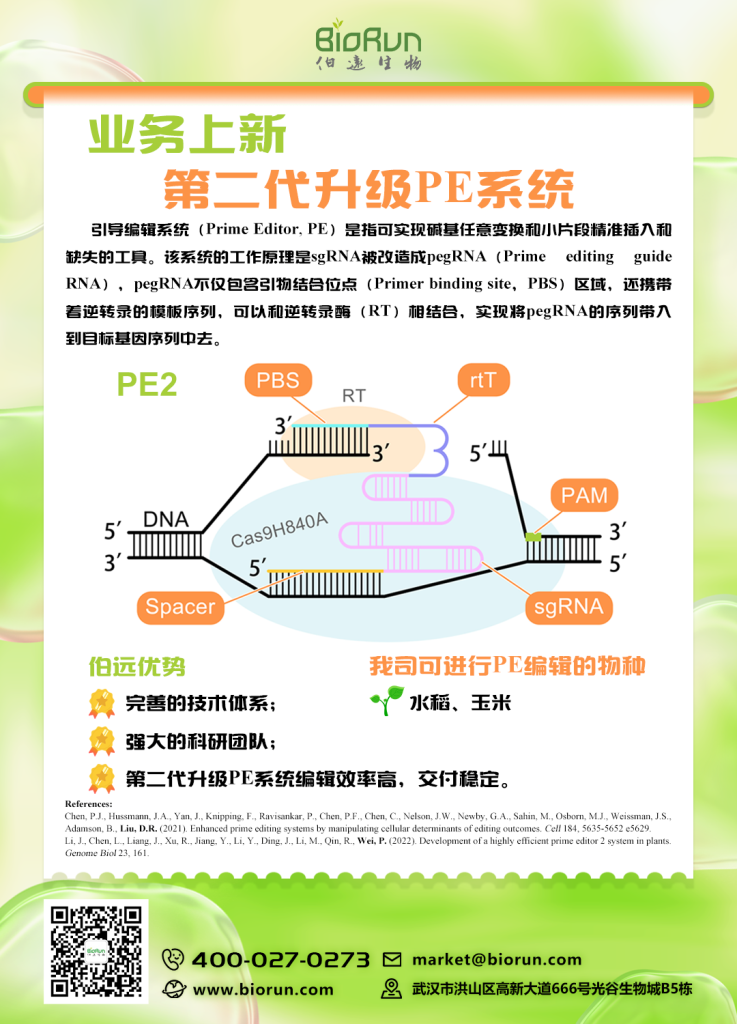
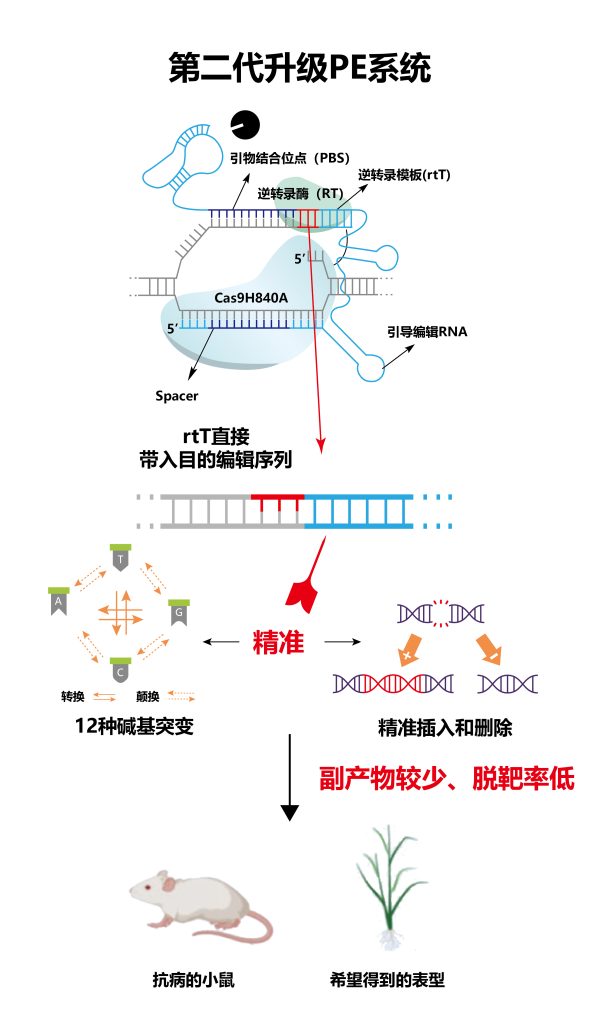
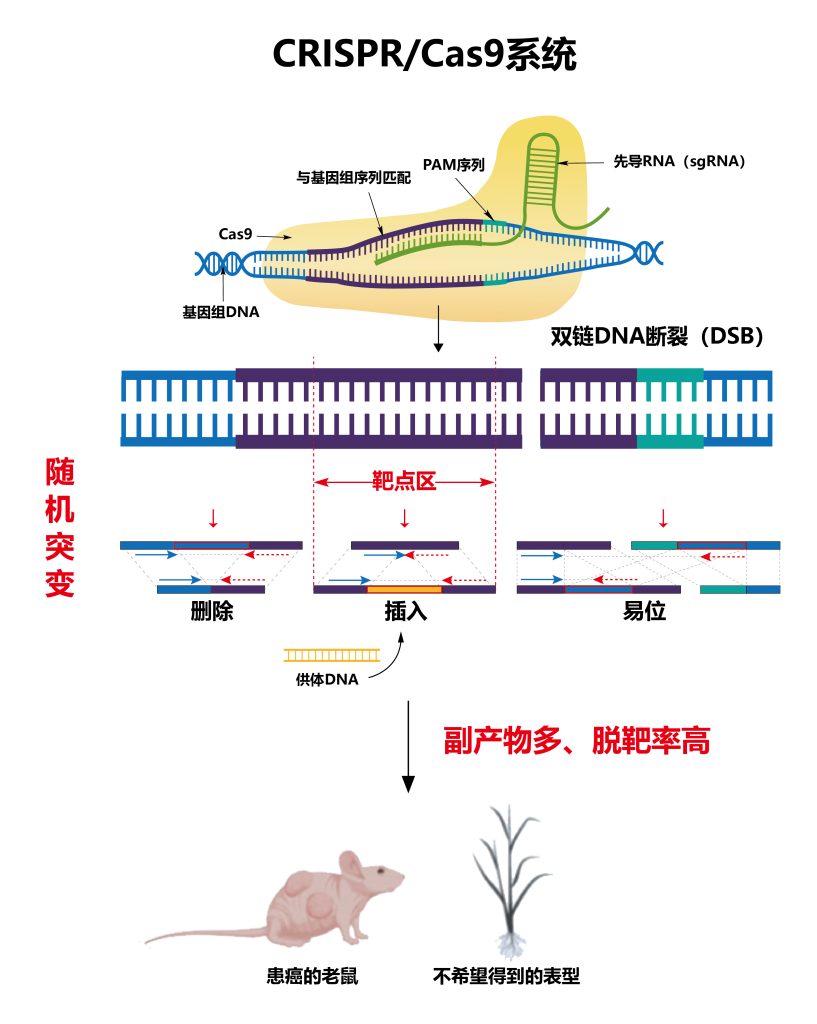
在本文开始之前先给大家直观展示一下第二代升级PE与传统CRISPR/Cas9的区别:
图1 第二代升级PE系统能够进行12种点突变以及精准插入和删除片段,产生的副产物较少、脱靶率低。
1.基因编辑时代的传奇序幕
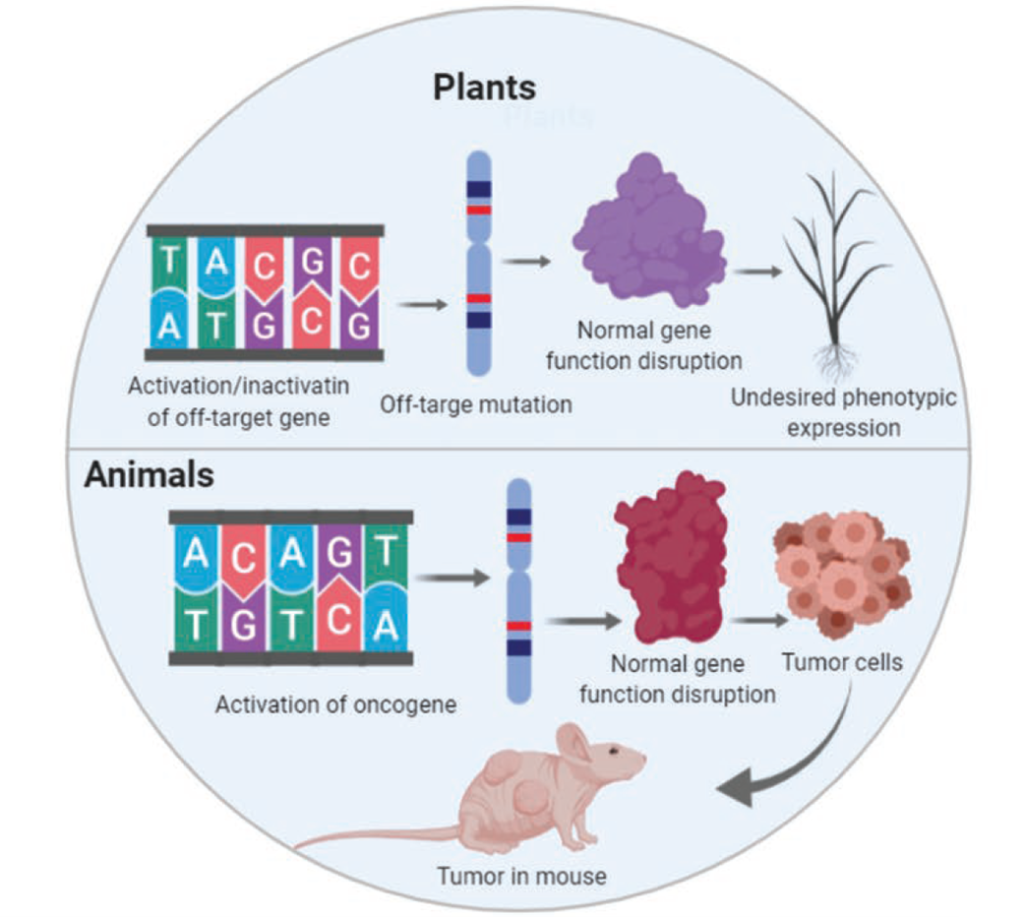
2.第一代引导编辑(Prime editing,PE)系统带来革新
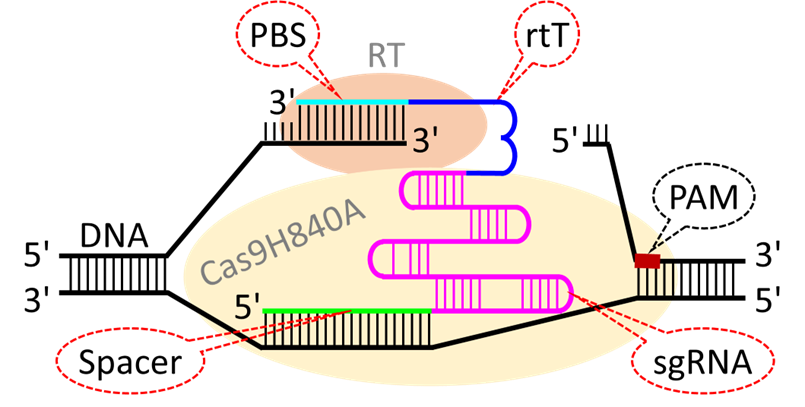
3.第二代升级PE系统诞生的风雨历程
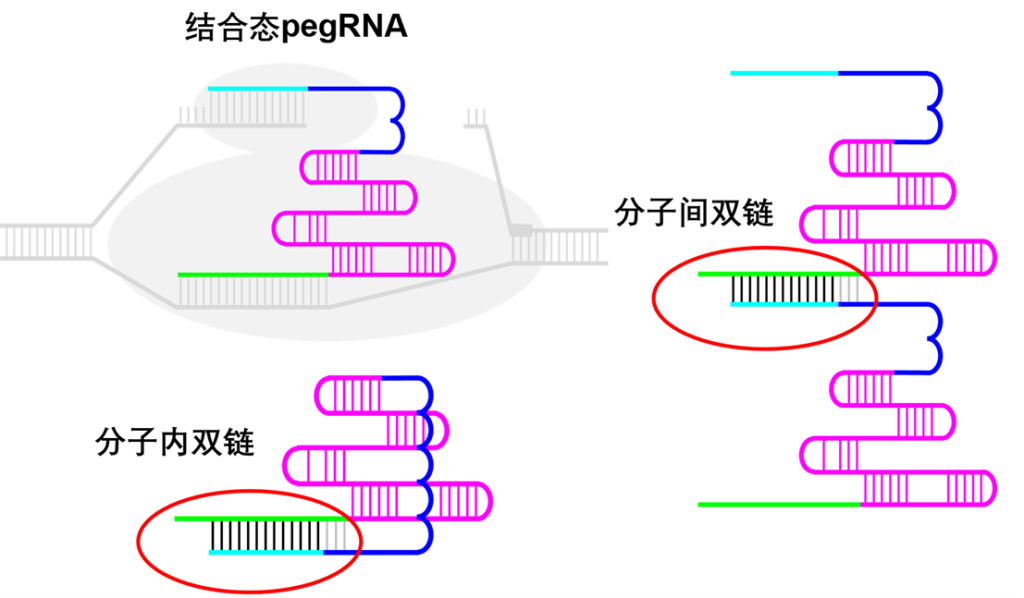
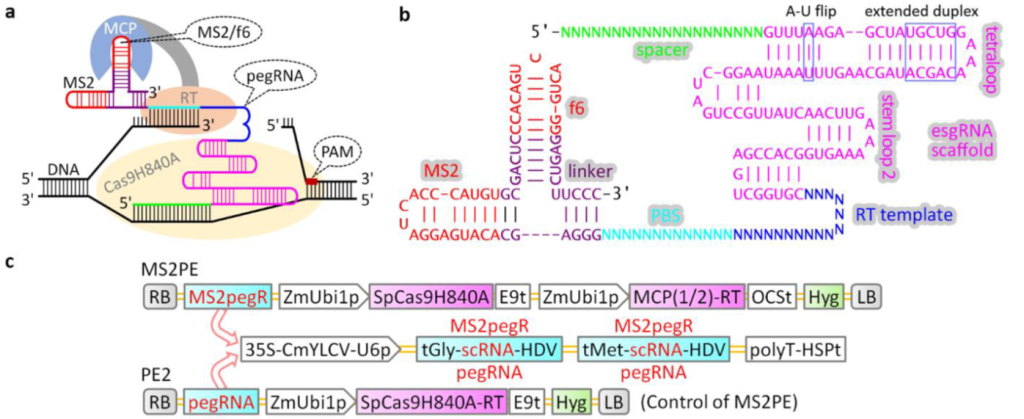 图6 添加MS2 RNA适配体的PE(MS2PE)。(a)MS2PE示意图;(b)MS2pegR的二级结构;(c)MS2PE的序列结构(Chai et al., 2021)。
图6 添加MS2 RNA适配体的PE(MS2PE)。(a)MS2PE示意图;(b)MS2pegR的二级结构;(c)MS2PE的序列结构(Chai et al., 2021)。
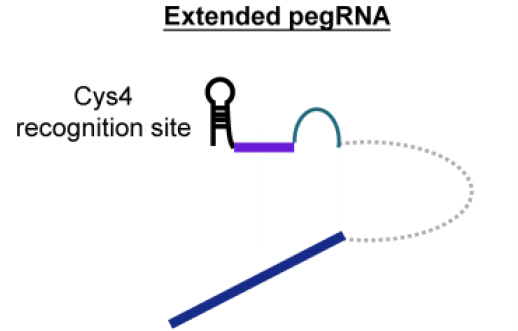
而西北农林科技大学王小龙团队及上海科技大学黄行许团队将20nt Csy4识别位点融合到pegRNA的3’端,该位点天然存在于I-F型CRISPR-Cas系统中,通过Csy4处理会形成一个发夹,被称为extended pegRNA(图7),在诱导Cas9介导的插入缺失方面优于原本的pegRNA(Liu et al., 2021),但也可能会导致更多随机的插入和缺失,并且Csy4的添加可能会阻碍PE系统进入细胞。
图7 Extended pegRNA的二级结构(Liu et al., 2021)。
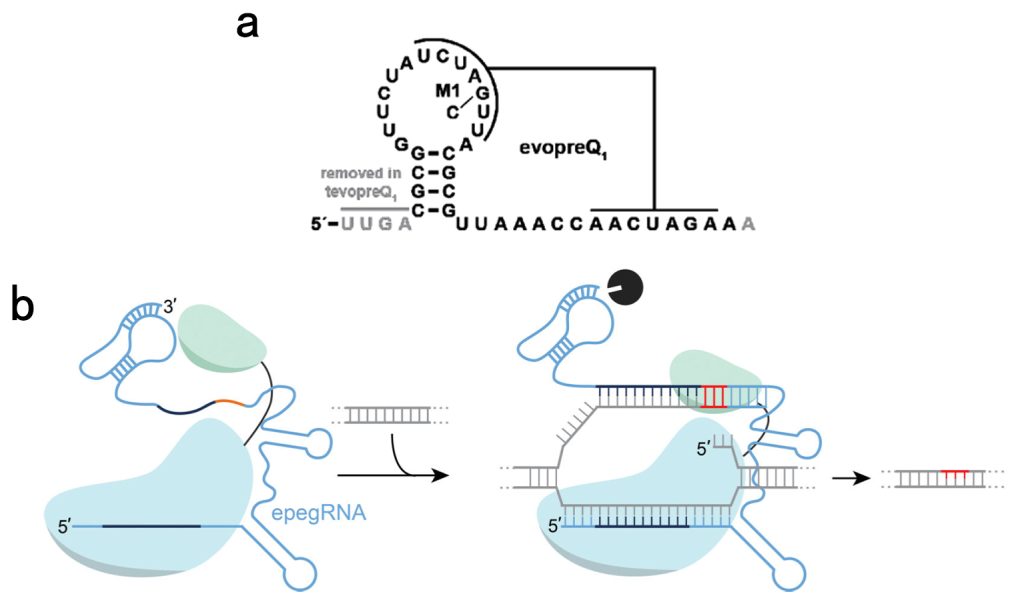
同样,David Liu团队设计了包含结构化RNA(evopreQ1)的工程化pegRNA(engineered pegRNA, epegRNA),保护3’延伸不被外切核酸酶降解,同时保证不形成影响编辑效率双链结构(图8)。epegRNA在HeLa、U2OS和K562细胞以及原代人成纤维细胞中将PE的编辑效率提高3到4倍,而不会增加脱靶率(Nelson et al., 2022)。
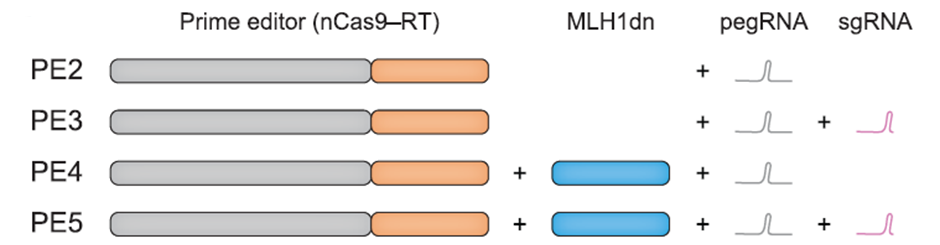
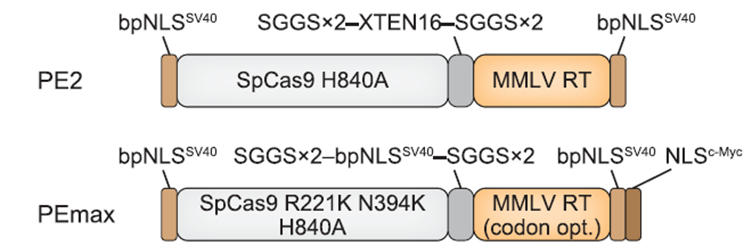
4.第二代PE系统在植物基因编辑上的应用

图11 ePPE示意图(Zong et al., 2022)。
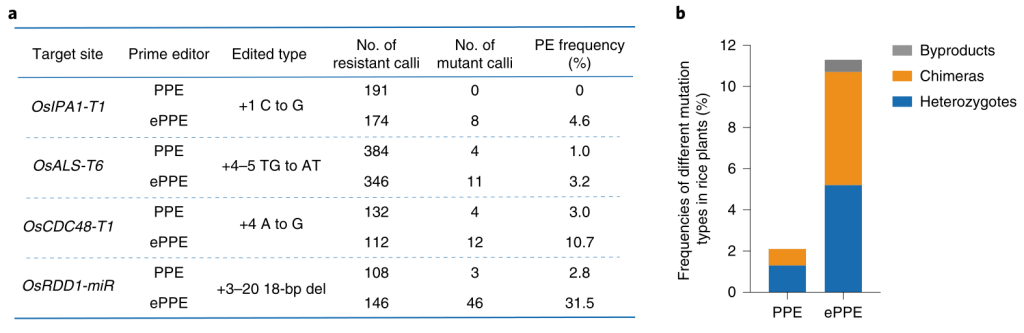
这种第二代PE系统ePPE与改造前的PPE相比,提高了各个内源位点的碱基替换、缺失和插入的效率,平均提高了5.8倍,同时,也没有观察到副产品或脱靶编辑的显著增加。使用ePPE生成耐受磺酰脲类和咪唑啉酮除草剂的水稻植株,编辑效率为11.3%,而使用PPE的编辑效率为2.1%(Zong et al., 2022)。
图12 ePPE在植物中的基因编辑情况。(a)PE和ePPE在四个靶位点对抗性水稻愈伤组织的编辑效率的比较;(b)使用PPE和ePPE得到的OsALS-T6 T0突变体中不同突变类型的效率(Zong et al., 2022)。
案例二:
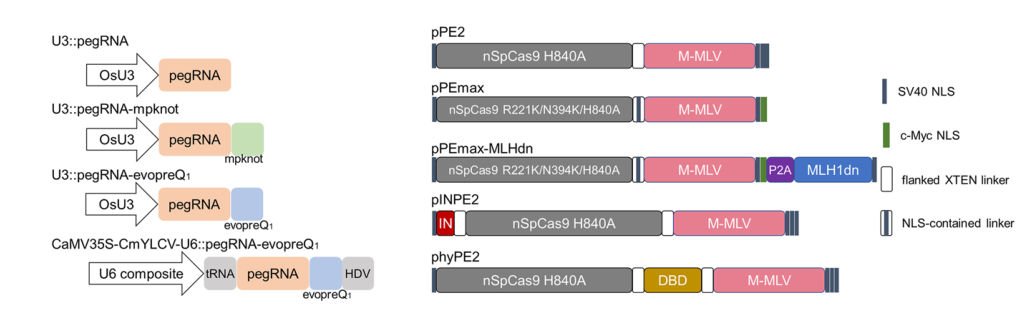
安徽农科院魏鹏程团队对提高水稻中PE系统编辑效率的各种策略进行了测试,通过可行策略的叠加开发出enpPE2系统,这一系统采用了蛋白酶优化后的PEmax,并结合受复合启动子控制的epegRNA(pegRNA-evopreQ1)。为了更有效地生成精确的编辑,通过用U6复合启动子代替OSU3启动子(图13)。测试结果表明,enpPE2可以在转基因植物中引入精确的突变,在T0水稻植株中,enpPE2的编辑效率为64.58%到77.08%之间(Li et al., 2022) 。
图13 PE系统的改良组合策略(Zou et al., 2022)。
案例三:
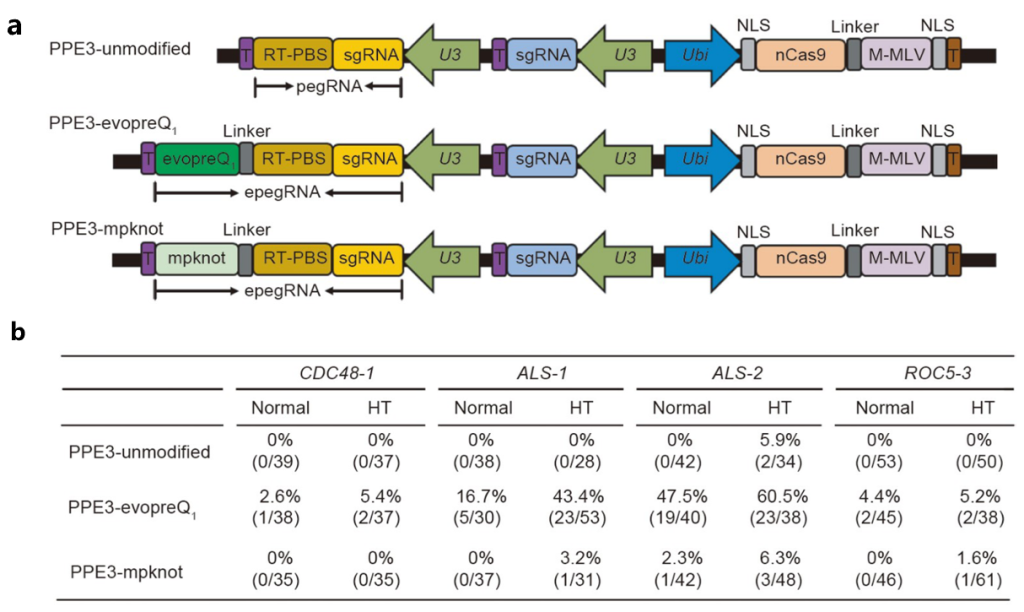
中科院黄勇团队和王春团队在pegRNA的3’末端分别添加了两个不同的结构化 RNA motif(evopreQ1和mpknot)以获得两个不同的epegRNA,并将它们与 nCas9-M-MLV以及niking sgRNA结合分别获得了PPE3-evopreQ1和PPE3-mpknot 系统(图14a)。当使用PPE3-evopreQ1系统结合高温处理时,在所有八个目标位点都获得了精确编辑的植物。此外,目标位点ALS-1和ALS-2的精确编辑效率分别达到了43.4%和60.5%(图14b)(Zou et al., 2022)。
References:
Anzalone AV, et al. 2019. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature 576:149-157.
Chai Y, Jiang Y, Wang J, Qiao D, Zhang Y, Xin C, Zhou Y, Wang X-C, Chen Q-J. 2021. MS2 RNA aptamer enhances prime editing in rice.
Chen PJ, et al. 2021. Enhanced prime editing systems by manipulating cellular determinants of editing outcomes. Cell 184:5635-5652 e5629.
Liu Y, Yang G, Huang S, Li X, Wang X, Li G, Chi T, Chen Y, Huang X, Wang X. 2021. Enhancing prime editing by Csy4-mediated processing of pegRNA. Cell Res 31:1134-1136.
Manghwar H, Li B, Ding X, Hussain A, Lindsey K, Zhang X, Jin S. 2020. CRISPR/Cas Systems in Genome Editing: Methodologies and Tools for sgRNA Design, Off-Target Evaluation, and Strategies to Mitigate Off-Target Effects. Adv Sci (Weinh) 7:1902312.
Nelson JW, et al. 2022. Engineered pegRNAs improve prime editing efficiency. Nat Biotechnol 40:402-410.
Zong Y, et al. 2022. An engineered prime editor with enhanced editing efficiency in plants. Nat Biotechnol.
Li J, Chen L, Liang J, Xu R, Jiang Y, Li Y, Ding J, Li M, Qin R, Wei P. 2022. Development of a highly efficient prime editor 2 system in plants. Genome Biol 23:161.
Zou J, Meng X, Liu Q, Shang M, Wang K, Li J, Yu H, Wang C. 2022. Improving the efficiency of prime editing with epegRNAs and high-temperature treatment in rice. Sci China Life Sci.
| 官网链接:plant.biorun.com |