在前几期的推文酵母杂交那些事儿(一)中小远给大家介绍了酵母单杂、双杂、三杂相关的实验原理,当时由于篇幅有限,有些内容还未讲完,所以,今天我们就接着上次没讲完的内容继续往后面讲,先跟大家剧透一下,今天的内容主要是拓展酵母双杂,大家肯定有疑问,上一次已经拓展的很多了,为啥还要拓展,小远猜想是因为酵母双杂是开启酵母杂交实验的开端,而每一种方法都存在其缺点与局限性,所以科学家们一直在探索如何改进,因此就诞生了很多关于酵母双杂的改良版本,下面就跟着伯小远一起去看看吧!
另一种蛋白融合到泛素N半端的突变体(NubG,即泛素分子被截断后的N端,并且将其中的第13位异亮氨酸突变为甘氨酸,故记为NubG)上,这种突变体对Cub缺乏内在的亲和力,从而可以避免发生自发重组。如果这两个蛋白相互作用,Cub和NubG被迫靠近并重新结合形成完整的泛素,然后被细胞泛素特异性蛋白酶(UBPs)识别。UBPs将融合在Cub上的多肽链裂解,从膜上释放LexA-VP16转录因子。然后,LexA-VP16移动到细胞核,在那里它可以激活整合在酵母报告菌株基因组中的LexA应答报告基因HIS3、ADE2和lacZ。蛋白质相互作用的检测是通过测量激活的报告基因的表达来实现的。因此,细胞质Y2H系统将发生在细胞核外的相互作用转化为一个确定的转录反应,导致在标准的β-半乳糖苷酶试验中,酵母菌落在选择培养基上生长或表现为蓝色菌落。
图1 cytoY2H系统原理图(Möckli et al., 2007)。
cytoY2H系统可以应用于传统酵母双杂交系统中难以研究的蛋白质,包括转录因子,如p53和NF-κB复合体成员。利用cytoY2H系统进行cDNA文库筛选,鉴定了未鉴定的酵母蛋白Uri1p(Uri1p是一种细胞质非常规预折叠蛋白,其分子功能目前尚未明确。之前无法在传统的Y2H体系中使用Uri1p,很可能是因为它的酸性区域较大。)的几个新的互作伙伴(Möckli et al., 2007)。cytoY2H系统通过提供一种方便的方法来筛选转录活性蛋白,是对蛋白质相互作用的检测方法的一种拓展!
既然这里提到了分裂泛素系统,那小远就再啰嗦一下,给大家讲讲膜体系酵母双杂的一些注意事项,在酵母杂交那些事儿(一)中小远只是对膜体系酵母双杂的原理进行了介绍,要是真的进行应用,相对于经典的酵母双杂系统,其过程会相对麻烦一点,不仅仅是研究的蛋白定位在细胞膜上那么简单,想要具体了解就请继续往下看!
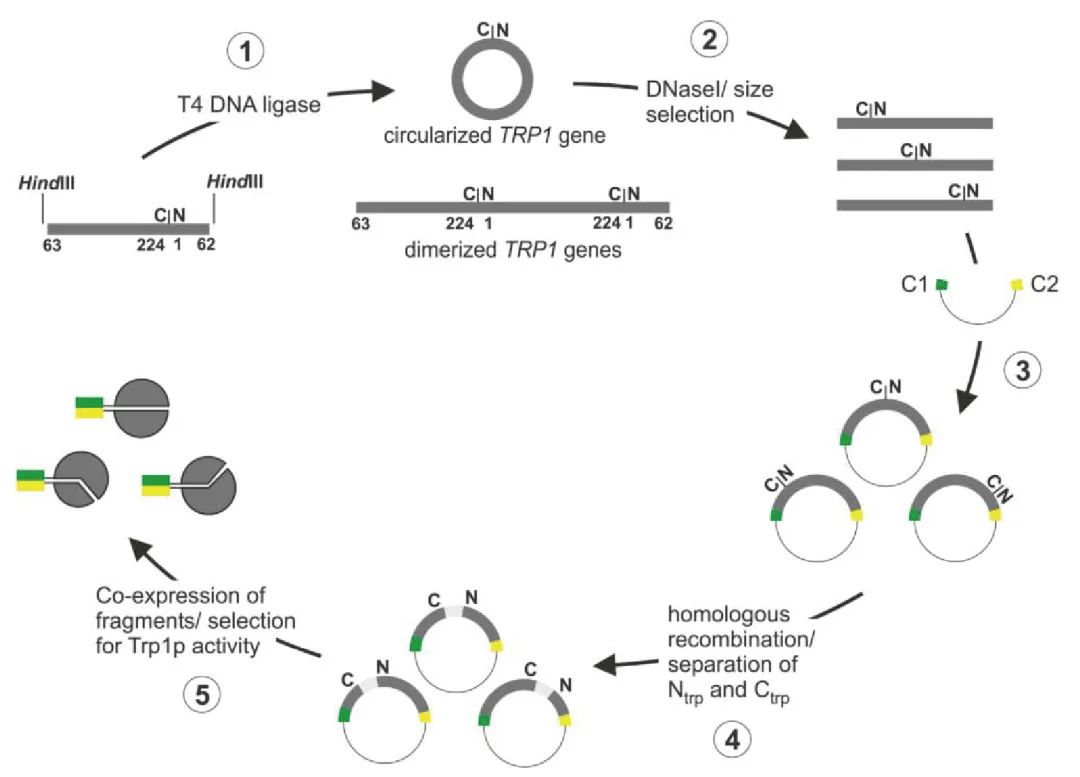 图2 分裂色氨酸蛋白生成的组合方法(Tafelmeyer et al., 2004) 。作为一个起点,使用一个TRP1基因的重排拷贝(灰色),在这个拷贝中,原始的TRP1的N端和C端由一个包含唯一AvrII位点的短连接子连接起来。(1)将线性片段与T4 DNA连接酶孵育,使基因成环,同时形成二聚体和高级寡聚体。(2)用DNaseI处理连接混合物,随机切割出线性分子,并分离出与TRP1大小相对应的片段。(3)分离的片段被克隆到一个酵母表达载体中,该载体包含两个多肽,它们结合成一个反向平行的螺旋状结构(绿色[C1]和黄色[C2]盒子)。需要注意的是,由于平末端克隆步骤,大多数克隆将携带TRP1片段,这些片段含有一个或两个形成螺旋的多肽,或以错误的方向插入质粒。(4)利用酵母细胞中的同源重组将一个终止子序列和PGAL1启动子(浅灰色框)插入Trp1p的原始N端和C端之间。(5)通过对这两个片段的共表达和对酵母细胞色氨酸缺陷互补的选择,分离出功能性的色氨酸分裂对。
图2 分裂色氨酸蛋白生成的组合方法(Tafelmeyer et al., 2004) 。作为一个起点,使用一个TRP1基因的重排拷贝(灰色),在这个拷贝中,原始的TRP1的N端和C端由一个包含唯一AvrII位点的短连接子连接起来。(1)将线性片段与T4 DNA连接酶孵育,使基因成环,同时形成二聚体和高级寡聚体。(2)用DNaseI处理连接混合物,随机切割出线性分子,并分离出与TRP1大小相对应的片段。(3)分离的片段被克隆到一个酵母表达载体中,该载体包含两个多肽,它们结合成一个反向平行的螺旋状结构(绿色[C1]和黄色[C2]盒子)。需要注意的是,由于平末端克隆步骤,大多数克隆将携带TRP1片段,这些片段含有一个或两个形成螺旋的多肽,或以错误的方向插入质粒。(4)利用酵母细胞中的同源重组将一个终止子序列和PGAL1启动子(浅灰色框)插入Trp1p的原始N端和C端之间。(5)通过对这两个片段的共表达和对酵母细胞色氨酸缺陷互补的选择,分离出功能性的色氨酸分裂对。
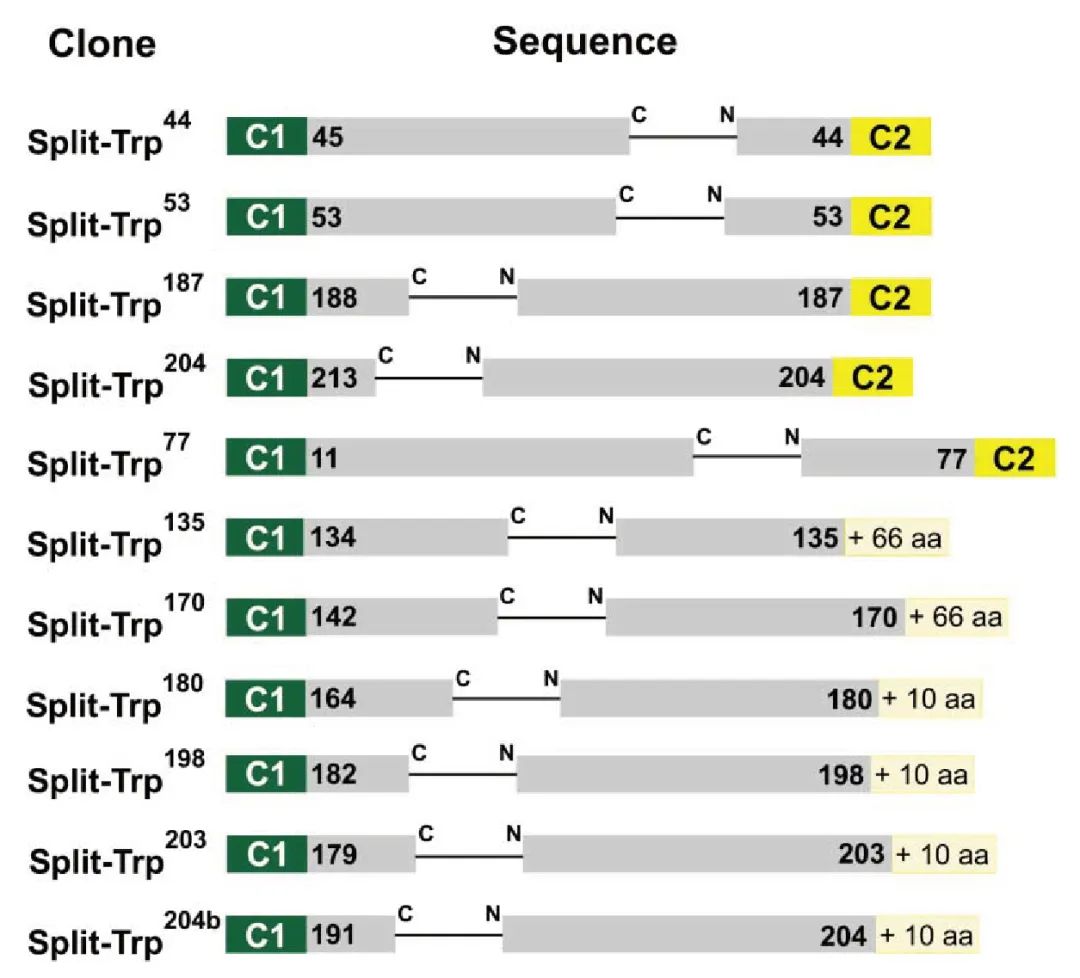
图3 酵母中可补充色氨酸缺陷的选择性分裂色氨酸蛋白对(Tafelmeyer et al., 2004) 。克隆以每个N端片段的最后一个残基命名。灰色矩形代表Trp1p对应的N端和C端片段。C1和C2是形成反平行螺旋[22]的两个多肽。由于阅读框的移位,11个克隆中有6个克隆的C2被10个或66个氨基酸的多肽取代。11个分离克隆中有6个具有重叠的TRP1序列。这就要求它们是由TRP1寡聚基因的2倍酶解产生的。
说明:图2图3介绍的是寻找有功能的色氨酸分裂对的方法,小远是第一次遇见这样的方法,虽然这个文献发表出来也有很多年了,但要不是这次写文章,可能也不会看见,不知道大家之前有没有遇到过呢!为了帮助大家理解,这里的C1和C2是可以互作的两个多肽,加了这个前提大家理解起来是不是就更容易啦。通过这种方式筛选出可用的色氨酸对之后,如图3所示,然后在细实线处切开,分成两段,即CTrp和NTrp,然后将C1和C2分别替换成我们要研究的目的蛋白,也就是分别将需要研究的两个蛋白构建至色氨酸的N端以及C端,然后共转酵母,在对应的培养基上进行筛选,如果可以在缺乏色氨酸的培养基上生长,那就证明两个蛋白存在互作!
上面介绍的两种方法都是经典酵母双杂系统的改进版,其实关于酵母双杂还有一些其它的方法,遇到的概率一般不是很大,所以,这里就不在做过多的介绍了,大家有兴趣可以自己去搜集文献查看哦!或者以后遇到了,小远觉得有必要也会再给大家讲的!
针对经典酵母双杂的局限性,科学家们探索出了不同的方法进行解决,那么针对工作量巨大的经典酵母双杂筛库,科学家们又做了哪些改进呢?
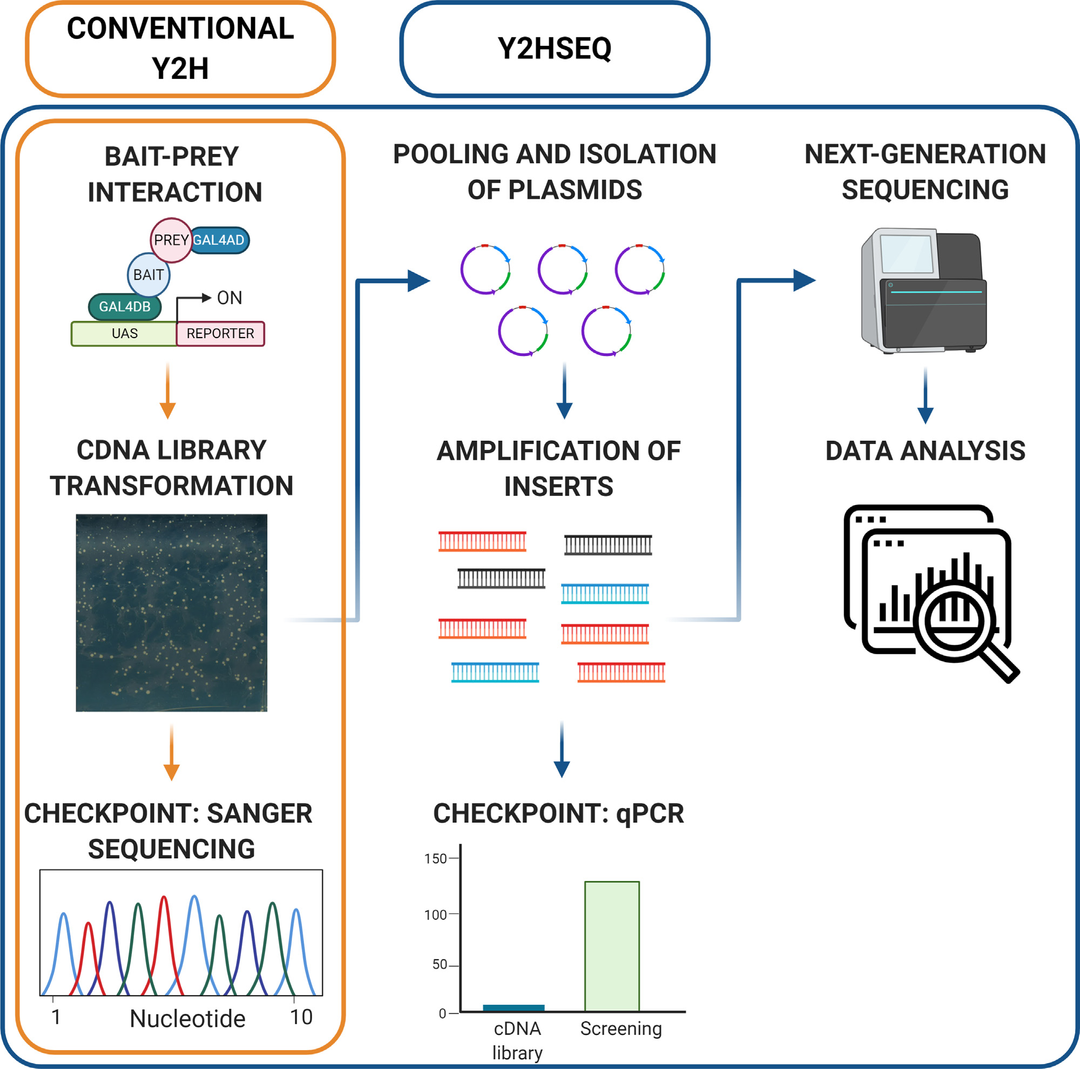
图4 Y2H-seq流程图(Erffelinck et al., 2018)。
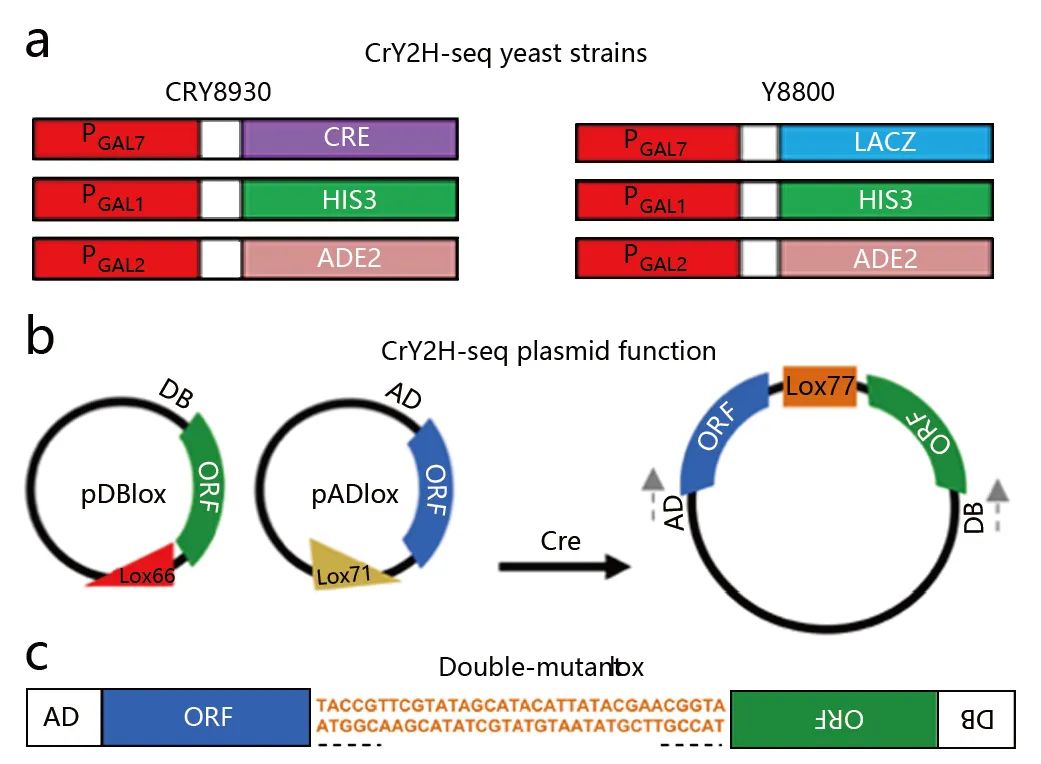 图5 CrY2H-seq菌株及质粒设计。(a)CrY2H-seq使用酵母株CRY8930和Y8800。(b)CrY2H-seq诱饵和猎物质粒pDBlox和pADlox在ORF插入的3’端侧翼含有lox突变位点(分别为lox66和lox71)。在质粒Cre/lox-重组后,使用DNA激活结构域(AD)和DNA结合结构域(BD)特异性引物,通过PCR扩增可恢复融合ORF产物,如图灰色箭头所示。(c)AD和DB引物的代表性PCR扩增子显示融合的ORF。突变的lox位点有下划线标记。
图5 CrY2H-seq菌株及质粒设计。(a)CrY2H-seq使用酵母株CRY8930和Y8800。(b)CrY2H-seq诱饵和猎物质粒pDBlox和pADlox在ORF插入的3’端侧翼含有lox突变位点(分别为lox66和lox71)。在质粒Cre/lox-重组后,使用DNA激活结构域(AD)和DNA结合结构域(BD)特异性引物,通过PCR扩增可恢复融合ORF产物,如图灰色箭头所示。(c)AD和DB引物的代表性PCR扩增子显示融合的ORF。突变的lox位点有下划线标记。
Fashena S J, Serebriiskii I, Golemis E A. The continued evolution of two-hybrid screening approaches in yeast: how to outwit different preys with different baits[J]. Gene, 2000, 250(1-2): 1-14.
Hastie A R, Pruitt S C. Yeast two-hybrid interaction partner screening through in vivo Cre-mediated Binary Interaction Tag generation[J]. Nucleic acids research, 2007, 35(21): e141-e141.
Hirst M, Ho C, Sabourin L, et al. A two-hybrid system for transactivator bait proteins[J]. Proceedings of the National Academy of Sciences, 2001, 98(15): 8726-8731.
Johnsson N, Varshavsky A. Split ubiquitin as a sensor of protein interactions in vivo[J]. Proceedings of the National Academy of Sciences, 1994, 91(22): 10340-10344.
Kim H, Yan Q, Von Heijne G, et al. Determination of the membrane topology of Ost4p and its subunit interactions in the oligosaccharyltransferase complex in Saccharomyces cerevisiae[J]. Proceedings of the National Academy of Sciences, 2003, 100(13): 7460-7464.
Laser H, Bongards C, Schüller J, et al. A new screen for protein interactions reveals that the Saccharomyces cerevisiae high mobility group proteins Nhp6A/B are involved in the regulation of the GAL1 promoter[J]. Proceedings of the National Academy of Sciences, 2000, 97(25): 13732-13737.
Lee T, Yang S, Kim E, et al. AraNet v2: an improved database of co-functional gene networks for the study of Arabidopsis thaliana and 27 other nonmodel plant species[J]. Nucleic acids research, 2015, 43(D1): D996-D1002.
Möckli N, Deplazes A, Hassa P O, et al. Yeast split-ubiquitin-based cytosolic screening system to detect interactions between transcriptionally active proteins[J]. Biotechniques, 2007, 42(6): 725-730.
Stagljar I, Korostensky C, Johnsson N, et al. A genetic system based on split-ubiquitin for the analysis of interactions between membrane proteins in vivo[J]. Proceedings of the National Academy of Sciences, 1998, 95(9): 5187-5192.
Stark C, Breitkreutz B J, Reguly T, et al. BioGRID: a general repository for interaction datasets[J]. Nucleic acids research, 2006, 34(suppl_1): D535-D539.
Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10: protein–protein interaction networks, integrated over the tree of life[J]. Nucleic acids research, 2015, 43(D1): D447-D452.
Tafelmeyer P, Johnsson N, Johnsson K. Transforming a (β/α) 8-barrel enzyme into a split-protein sensor through directed evolution[J]. Chemistry & biology, 2004, 11(5): 681-689.
Venkatesan K, Rual J F, Vazquez A, et al. An empirical framework for binary interactome mapping[J]. Nature methods, 2009, 6(1): 83-90.
Weimann M, Grossmann A, Woodsmith J, et al. A Y2H-seq approach defines the human protein methyltransferase interactome[J]. Nature methods, 2013, 10(4): 339-342.
Yachie N, Petsalaki E, Mellor J C, et al. Pooled‐matrix protein interaction screens using Barcode Fusion Genetics[J]. Molecular systems biology, 2016, 12(4): 863.
Yu H, Tardivo L, Tam S, et al. Next-generation sequencing to generate interactome datasets[J]. Nature methods, 2011, 8(6): 478-480.
官网链接:plant.biorun.com