伯小远经常碰见老师研究的物种没有成熟的遗传转化体系,这意味着过表达基因和敲除基因都做不了,连这种最基础的实验都做不了,那如何研究基因功能呢,是不是就真的没办法解决了呢,下面跟着伯小远来看看吧。
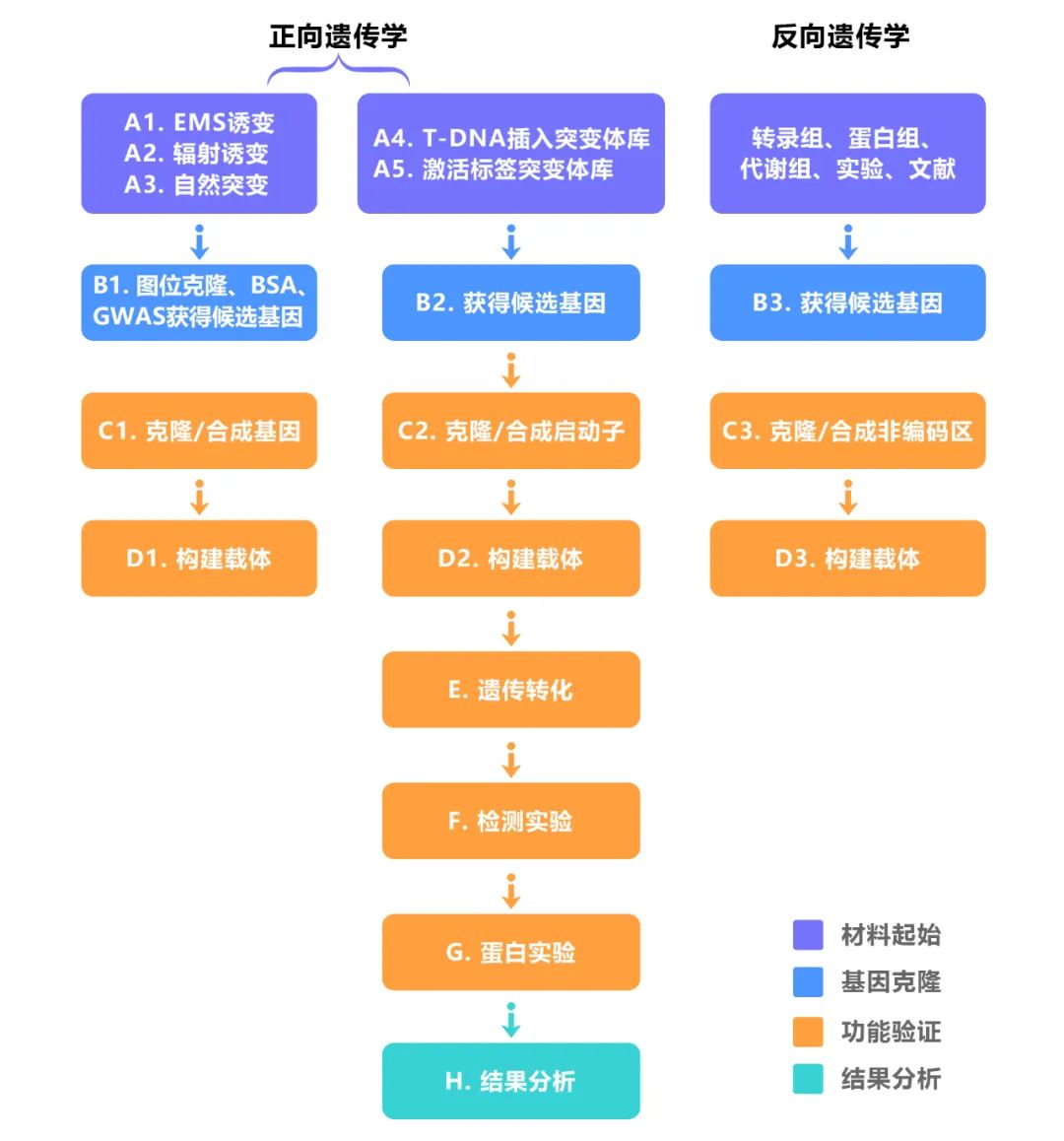
图1 基因功能研究套路,图片来源:伯远生物。
A1-A3:EMS诱变、辐射诱变、自然突变或其他方式获得差异性状材料,一般不知道突变在哪里,需要极辛苦地去鉴定。
A4-A5:T-DNA插入突变体库、激活标签突变体库,不知道突变在哪里,但可以轻松鉴定。现在有些实验室已经创制了基因编辑突变体库,它相比标签突变体库的优点是,能够更直接的知道突变基因的位置和突变情况。
B:一般在获得候选基因的序列后,会在本物种中对同源基因进行序列比对,以及在不同物种中对该基因及其同源基因甚至蛋白序列进行序列比对、进化关系分析等。
C1:克隆模板:一般是cDNA,少数情况会选择以基因组为模板。
C2:克隆模板:基因组。
C3:克隆模板:基因组,这里的非编码序列是指miRNA、lncRNA、circRNA。
注:C中如果不知道完整的基因组序列可用DNA walking技术,如果不知道完整的CDS序列,可试试3’RACE、5’RACE技术。
D1:构建载体:按载体功能分类,构建过表达载体/互补载体、发夹干扰载体、amiRNA载体、基因编辑载体等;按启动子功能分类,除了常用的组成型表达载体,也会根据实验目的构建诱导型启动子、组织特异性启动子载体。
D2:构建启动子分析载体:研究基因的时空表达,即目的基因的启动子+报告基因;启动子活性分析/启动子和转录因子是否相互作用:使用双荧光素酶报告基因系统检测。
D3:构建载体:过表达miRNA/lncRNA/circRNA前体、构建STTM载体以干扰miRNA。
注:D中所涉及的载体构建技术:传统的双酶切-连接-转化、Gateway克隆技术、Golden Gate克隆技术、Gibson同源重组技术,现在一般使用后两种构建技术。
E:植物转基因技术,一般使用农杆菌介导的遗传转化体系,优点是效率高、T-DNA整合效果好、拷贝数低。
F:对转基因植株的检测:
1、确定转基因阳性植株:PCR(由于实验室气溶胶污染易出现假阳性,建立标准PCR实验室可避免出现这种情况),也可以使用试纸条快速鉴定真核抗性基因;
2、荧光定量PCR(qRT-PCR),检测基因的相对表达量即mRNA的相对表达量的,用来分析基因表达丰度和性状之间的相关性;注意qRT-PCR还可以用于检测miRNA/lncRNA、拷贝数鉴定、绝对定量等;
3、Western blot,用来分析蛋白表达量及其与性状之间的相关性;
4、TAIL PCR技术分析转基因插入位点在基因组的位置;
5、Southern blot技术用于分析转基因拷贝数(也可以使用qRT-PCR进行确定,但Southern是金标准)。
G1:体外表达纯化,可以用大肠杆菌或酵母,然后通过蛋白标签进行亲和纯化。对于编码基因可以进行体外表达,纯化到目标蛋白后可以做一些体外活性实验,如果是一个酶可以做催化活性验证,如果是转录因子可以进一步分析其识别的DNA元件,如果是信号蛋白可以进一步分析其相互作用蛋白。
G2:亚细胞定位实验:先通过生物信息学预测基因的定位区域,然后通过与Marker蛋白共定位确定其亚细胞定位的位置。
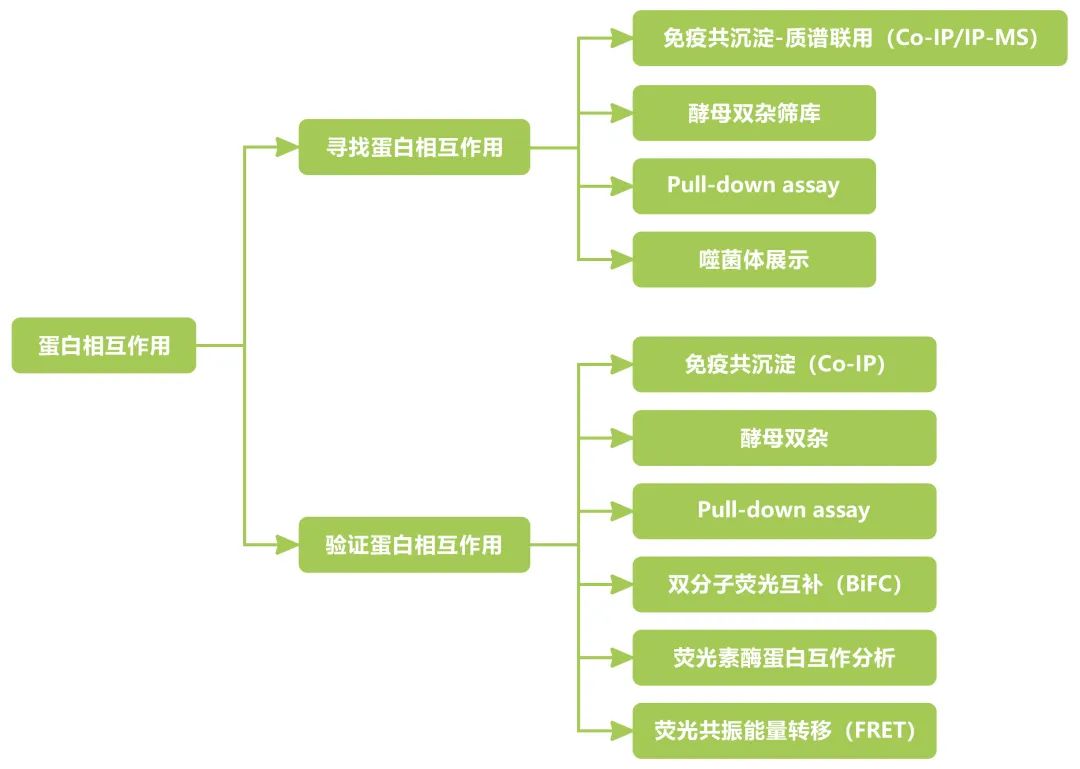
G3:筛选蛋白-蛋白相互作用:酵母双杂筛库、IP-MS、Pull down-MS等;
检测蛋白-蛋白相互作用:酵母双杂、Co-IP、GST Pull-down、BiFC、荧光素酶蛋白互作分析等。
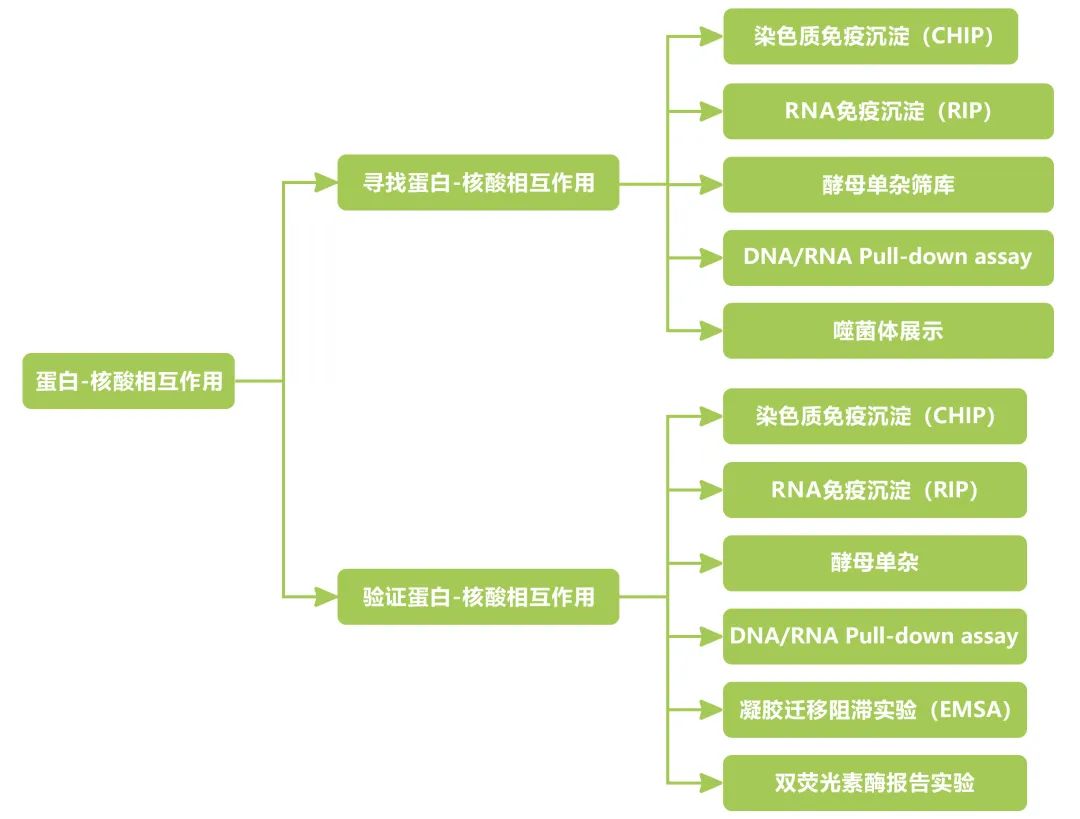
G4:筛选蛋白-核酸相互作用:酵母单杂筛库(用启动子筛选转录因子)、CHIP-seq(用转录因子找启动子)、DNA/RNA Pull-down、RIP等;
检测蛋白-核酸相互作用:酵母单杂、EMSA、CHIP、双荧光素酶报告基因系统、DNA/RNA Pull-down、RIP等。
H:可将上述实验结果再结合转录组、蛋白组、代谢组等数据一起分析,这些结果原则上具有一致性和统一性,能够相互解释和印证。由蛋白实验筛选出的相互作用蛋白、核酸等可重复以上研究路线,通过表型或检测数据更进一步将信号通路范围拓宽,最终推断出信号通路图。
注:伯小远在这里只总结了较常见的实验思路,还有许多实验大家可以根据不同的实验目的再自行补充总结喔!
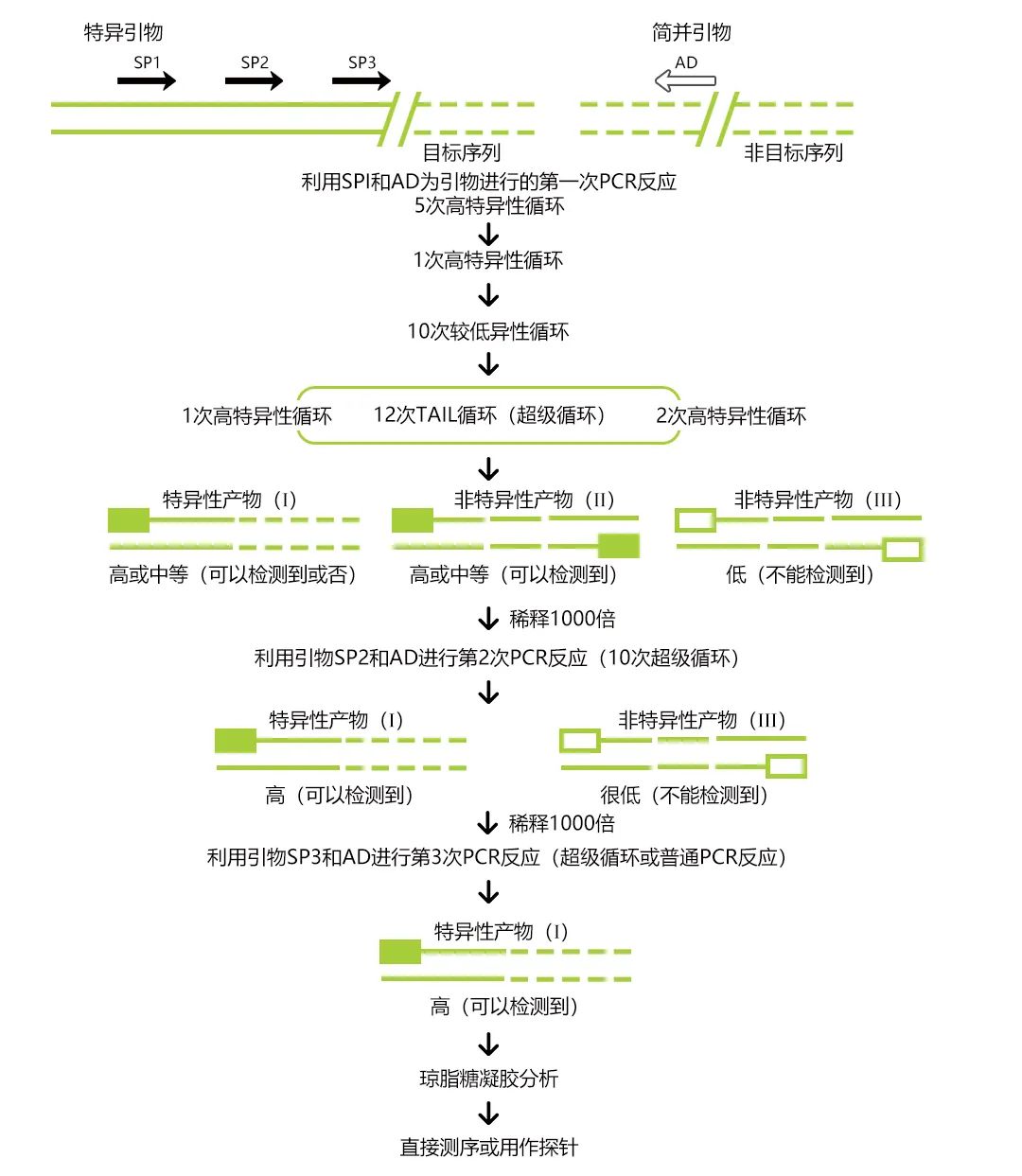 图2 染色体步移的实验原理。基于PCR技术的染色体步移技术先后有十几种方法,可以克隆已知序列的侧翼未知序列,目前最常使用的是TAI LPCR(Thermal Asymmetric Interlaced PCR,热不对称交错PCR)技术,利用特异性套嵌引物,结合简并引物钓取侧翼序列。其基本原理是利用目标序列旁的已知序列设计3个嵌套的特异引物(SP1,SP2,SP3),用它们分别和1个具有低Tm值的短的随机简并引物(AD)相结合,根据引物的长短和特异性的差异设计不对称的温度循环,通过分级反应来扩增特异产物。
图2 染色体步移的实验原理。基于PCR技术的染色体步移技术先后有十几种方法,可以克隆已知序列的侧翼未知序列,目前最常使用的是TAI LPCR(Thermal Asymmetric Interlaced PCR,热不对称交错PCR)技术,利用特异性套嵌引物,结合简并引物钓取侧翼序列。其基本原理是利用目标序列旁的已知序列设计3个嵌套的特异引物(SP1,SP2,SP3),用它们分别和1个具有低Tm值的短的随机简并引物(AD)相结合,根据引物的长短和特异性的差异设计不对称的温度循环,通过分级反应来扩增特异产物。
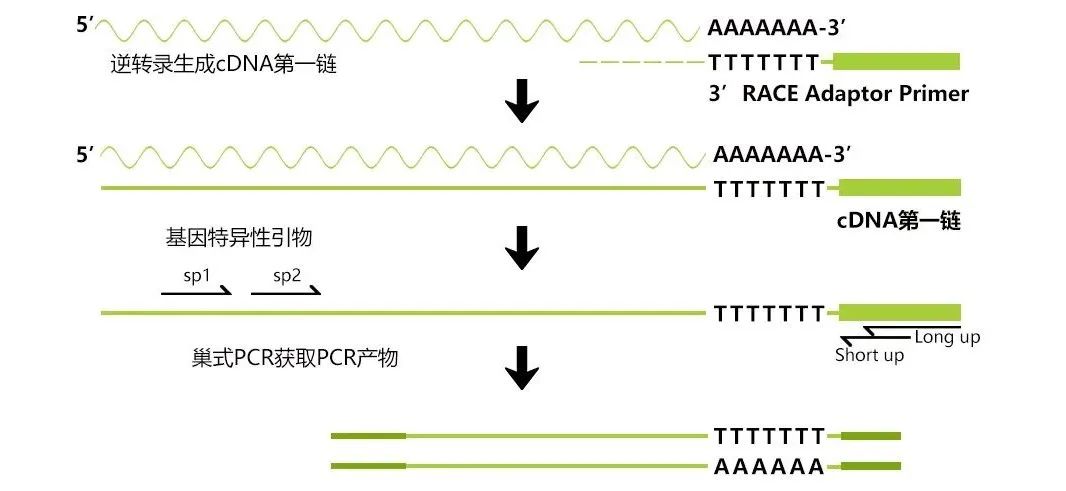
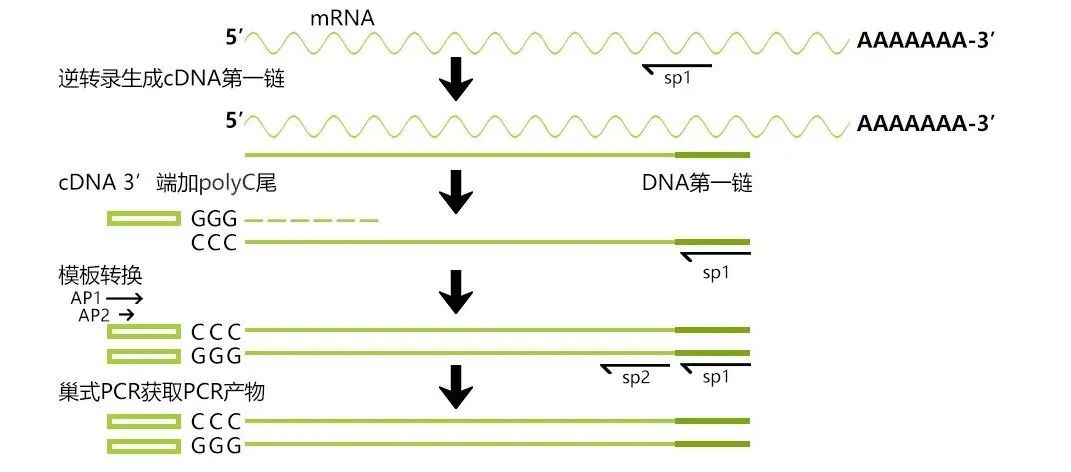
图4 5’RACE原理图。
4.1.1 基因的时空表达模式
qRT-PCR技术的介绍可参考文章“qPCR技术,你需要知道这些事!”
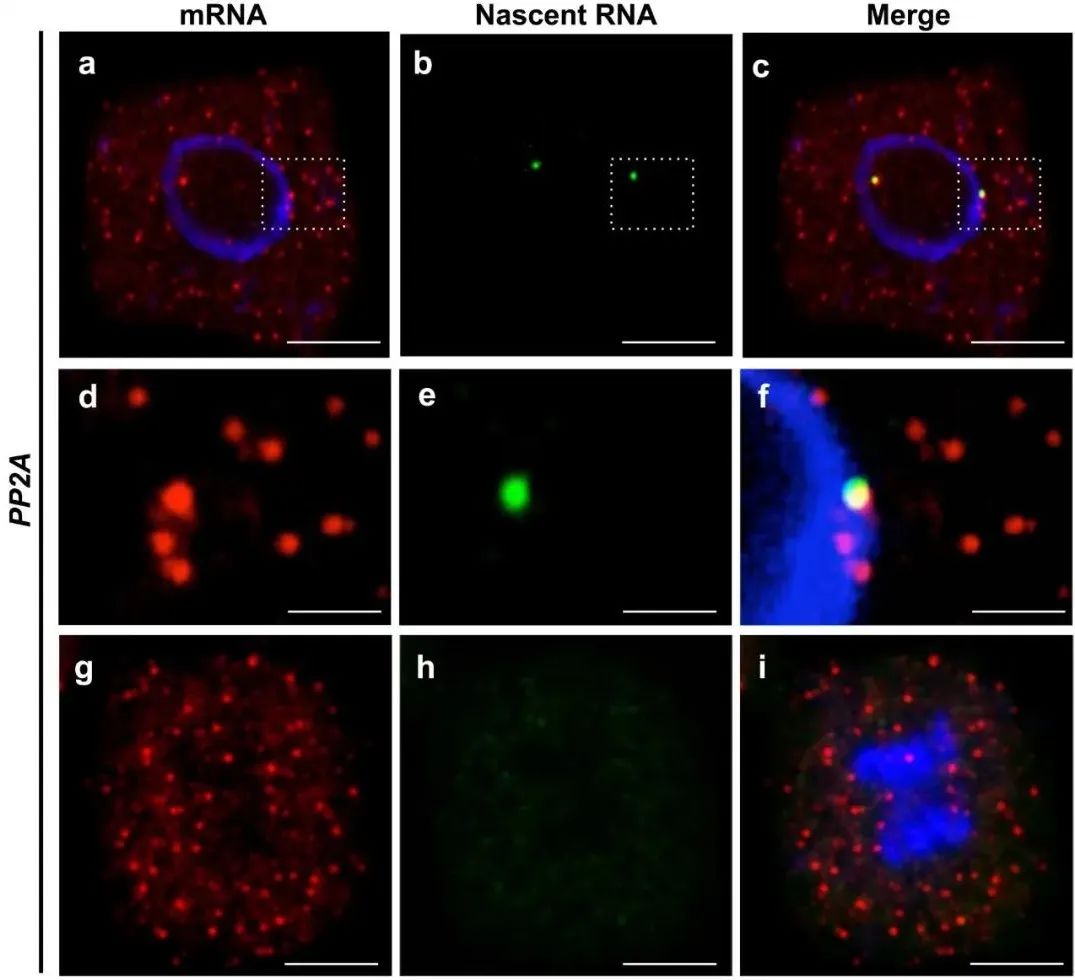
4.1.1.2 在mRNA水平上通过原位杂交进行研究。
4.1.1.3 在蛋白水平上通过Western blot方法进行研究。
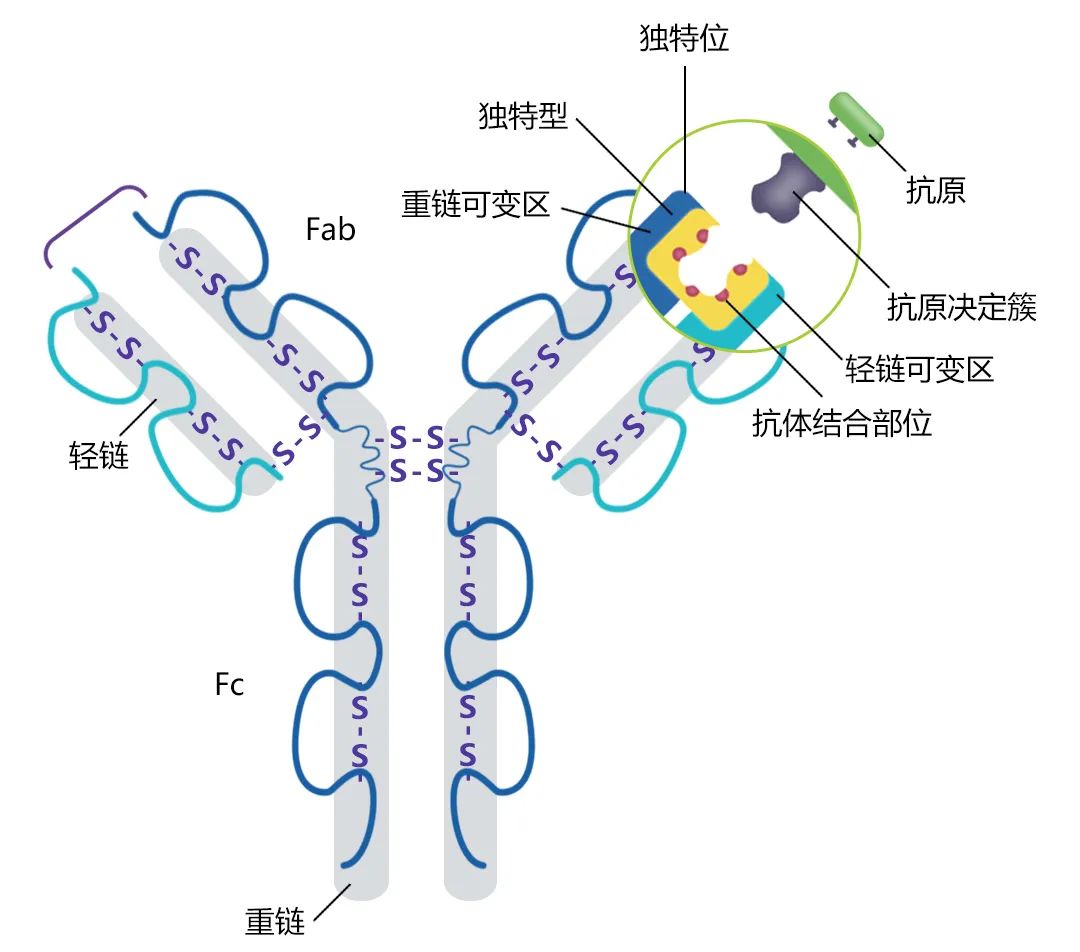 图7 抗原和抗体结合的原理示意图。抗体以Y型单元的形式存在,由四条多肽链组成,每个Y含有两个相同的重链拷贝和两个相同的轻链拷贝。Y型单元由两个可变的抗原特异性Fab臂和与免疫细胞Fc受体结合的恒定Fc尾,Fab臂对实际的抗原结合至关重要。
图7 抗原和抗体结合的原理示意图。抗体以Y型单元的形式存在,由四条多肽链组成,每个Y含有两个相同的重链拷贝和两个相同的轻链拷贝。Y型单元由两个可变的抗原特异性Fab臂和与免疫细胞Fc受体结合的恒定Fc尾,Fab臂对实际的抗原结合至关重要。
大家在实验室中应该经常听到师兄师姐们提到一抗或者二抗,那它们又是什么呢?
一抗是一种免疫球蛋白,能特异性结合目的蛋白或重点研究的其他生物分子,目的是对其进行纯化、检测和测量。使用小鼠、大鼠、兔子、山羊和其他动物作为寄主,可将一抗培育成多克隆或单克隆抗体,并且可直接购买或定制。
二抗可结合针对某一特定物种(如兔子)的所有一抗,即抗体的抗体,属于通用型抗体。二抗上会耦联标记,例如酶(辣根过氧化酶HRP和碱性磷酸酶AP或其衍生物)、荧光基团(FITC、Rhodamine、Texas Red、PE、Rhodamine、Dylight)、生物素、金颗粒等,这些标记可以很容易被检测出来,这样就可以指示出目的蛋白。
如果一抗自带可以被检测出的耦联标记,则不需要二抗,但这样会导致生产成本很高,所以商业化的二抗就设计为可识别所有的一抗,这样可以免去对每一个一抗进行标记。
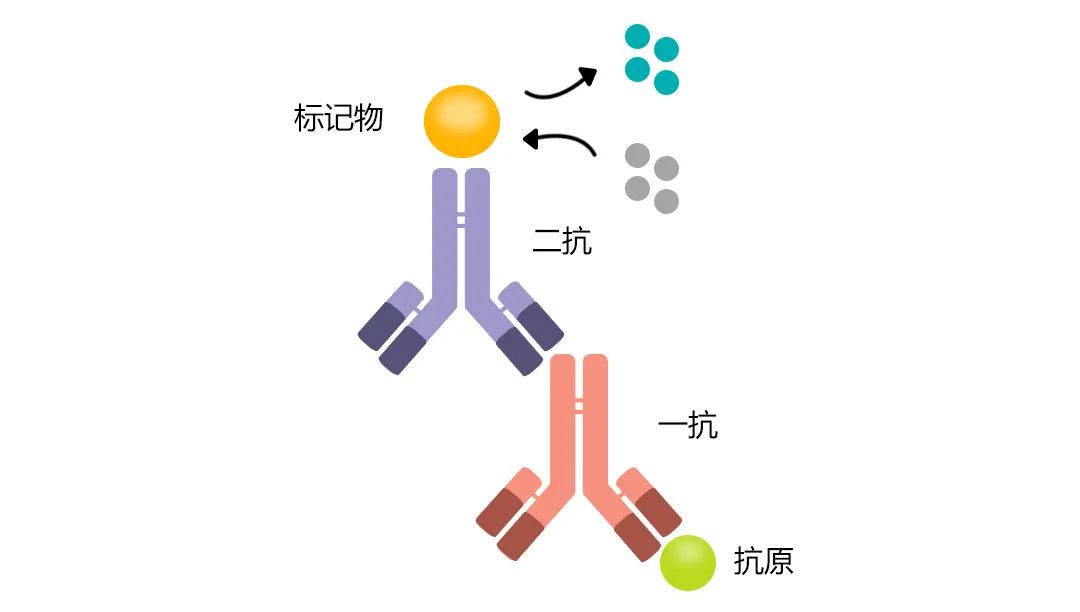
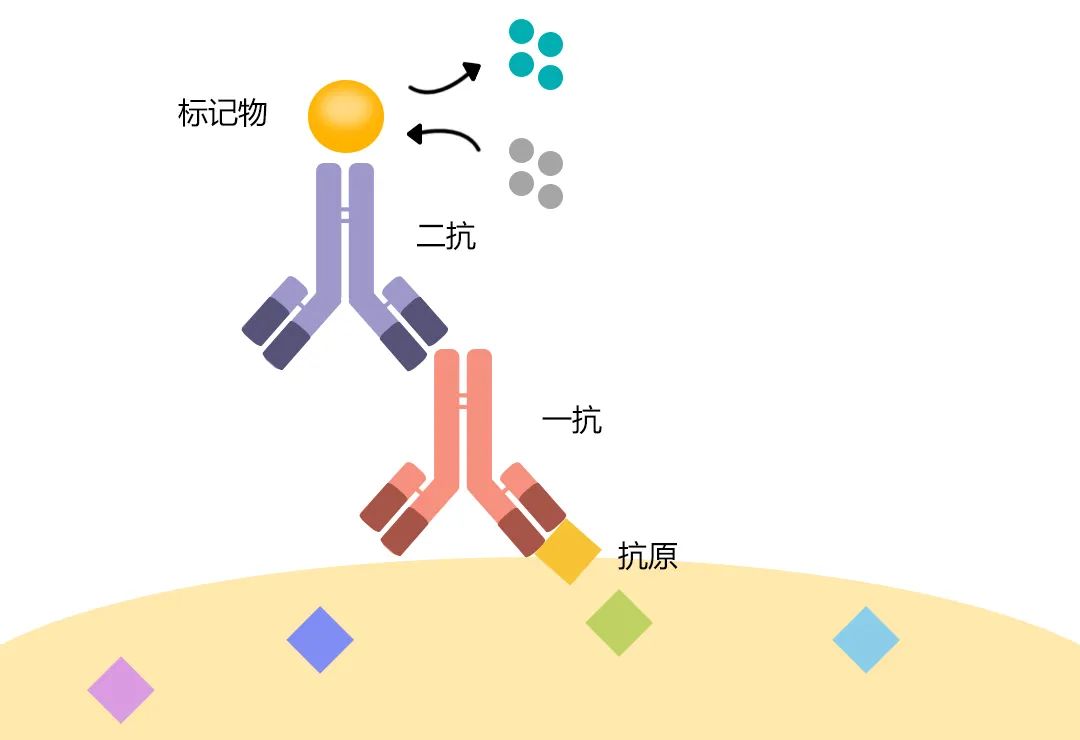
图8 目的蛋白、一抗、二抗结合示意图。一抗用来特异性识别目的蛋白,二抗用来放大信号并显色。
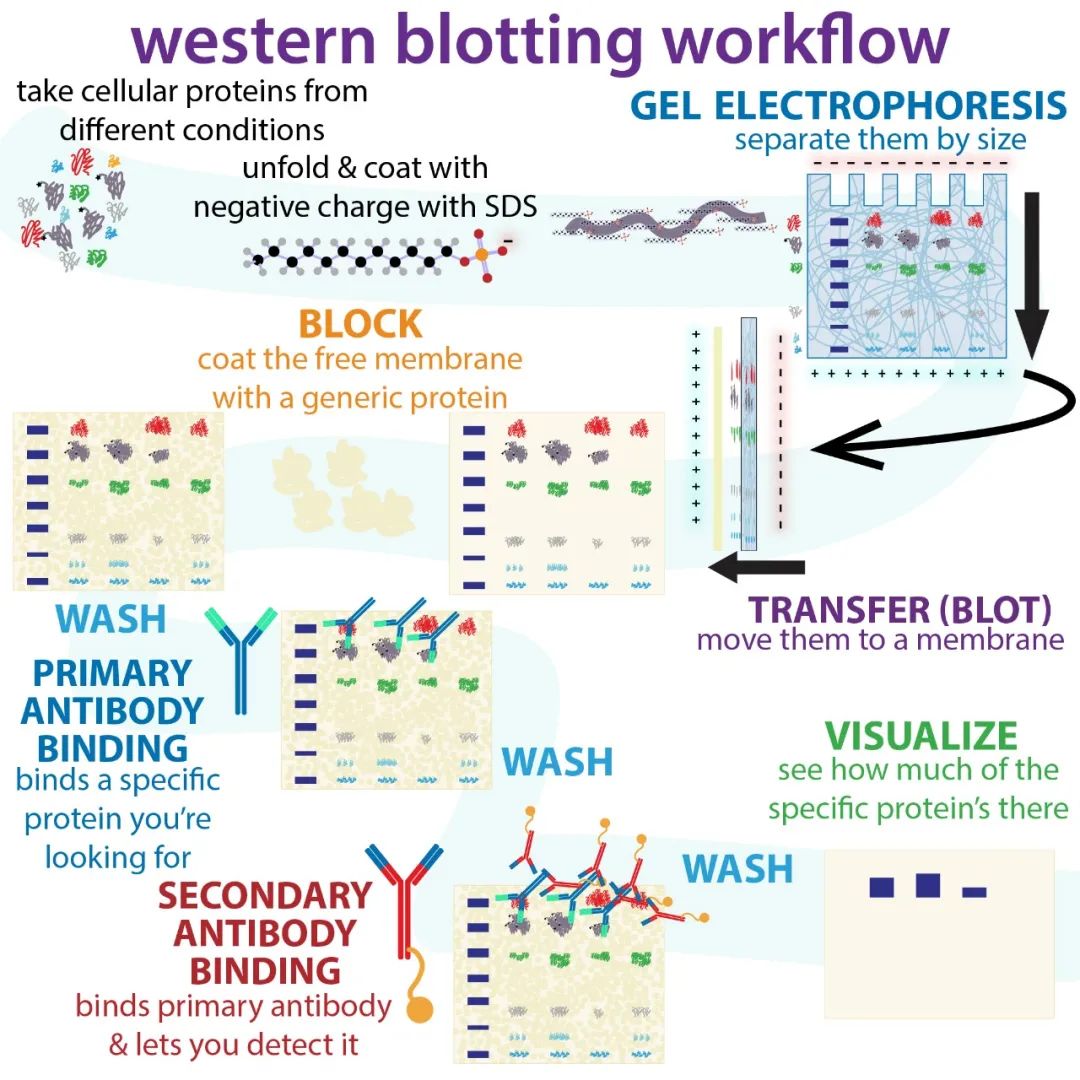
图9 Western blot实验流程图。图片来源:Biochemlife。实验流程大致分为:提蛋白,蛋白含量测定,聚丙烯酰胺凝胶电泳(SDS-PAGE)分离蛋白样品,转膜,封闭,孵育一抗,孵育二抗,显影。
4.1.1.4 在蛋白水平上通过免疫组织化学染色进行研究。
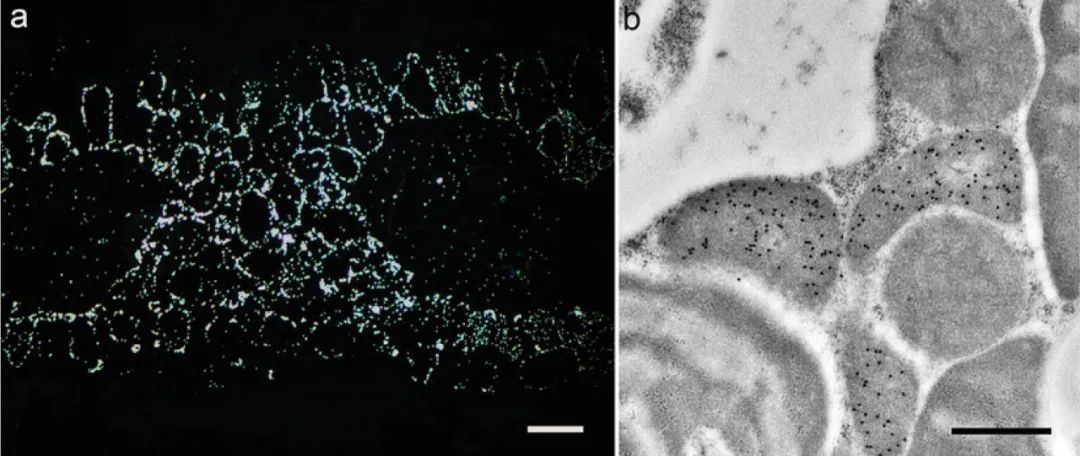
4.1.1.4.2 IHC中的免疫荧光(IF)
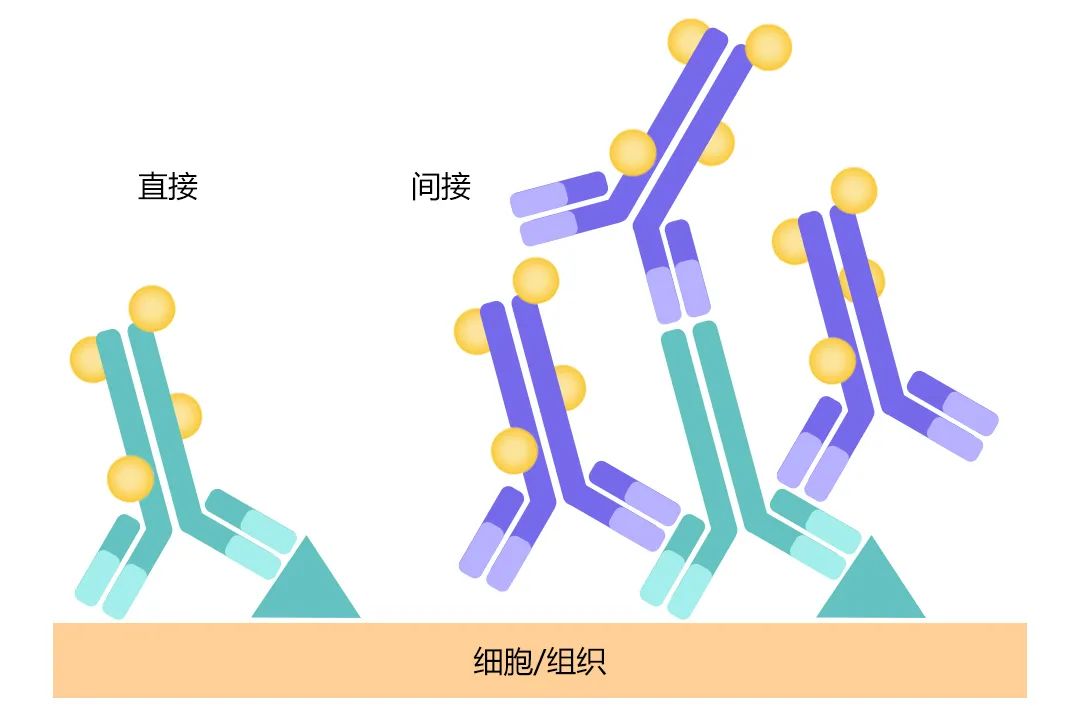
图13 IF原理图。直接法:将标记的特异性荧光抗体直接加在抗原标本上;间接法:先用特异抗体(一抗)与样本中的抗原进行反应,用水洗去未反应的抗体,再用标记的抗体(二抗)与抗原反应,使之形成抗体—抗原—抗体复合物。上述的CIH其实也分为直接法和间接法,原理与此类似。
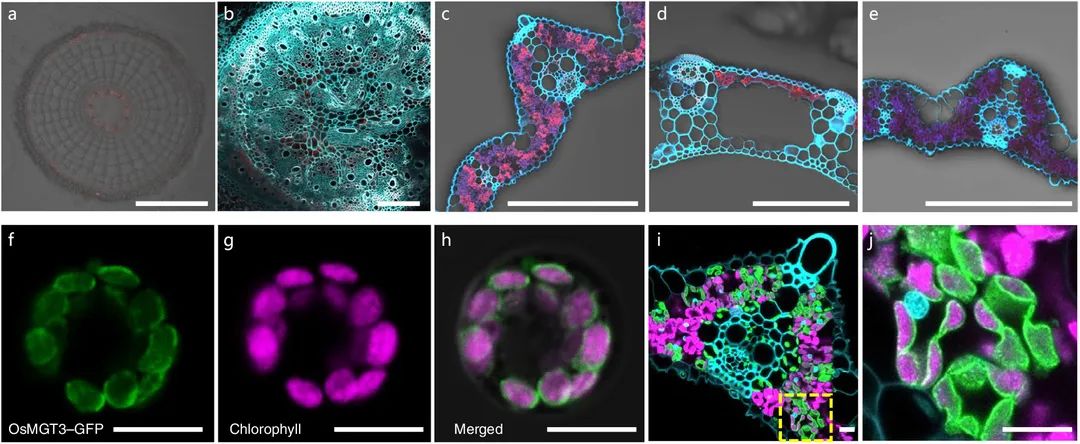
图14 OsMGT3蛋白的表达模式和亚细胞定位情况 (Li et al. 2020)。该文研究了转运蛋白OsMGT对叶绿体中Mg2+的转运调控影响了水稻的光合效率。(a-e)OsMGT3蛋白表达的组织特异性。使用GFP的抗体对pOsMGT3-GFP转基因水稻(a-d)和野生型水稻(e)进行免疫染色,组织来自距根尖10mm的根横截面(a)、基部节点(b)、叶片(c、e)、叶鞘(d)。红色表示来自GFP抗体的信号,青色表示水稻的细胞壁。(f-h)水稻原生质体中OsMGT3的亚细胞定位。OsMGT3-GFP在水稻原生质体中瞬时表达,该定位信号显示与叶绿体自发荧光信号可重叠,说明OsMGT3定位于叶绿体中。(i、j)对OsMGT3-GFP转基因水稻的叶片进行免疫染色。绿色表示来自GFP抗体的信号,粉色表示叶绿体,青色表示水稻的细胞壁。
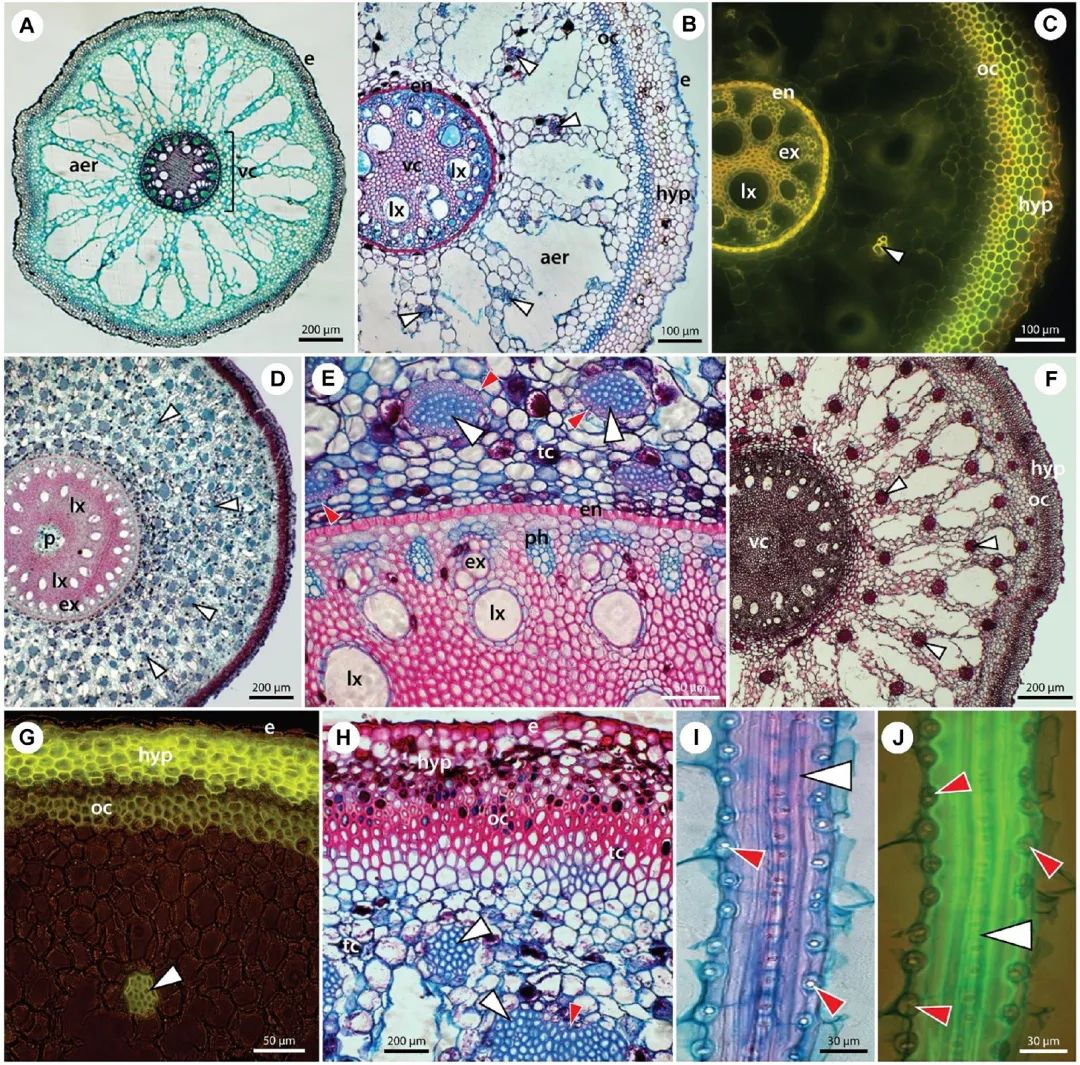
4.1.1.5 在蛋白水平上通过亚细胞定位进行研究。
4.1.1.6 在蛋白水平上通过检测报告基因的表达情况进行研究。
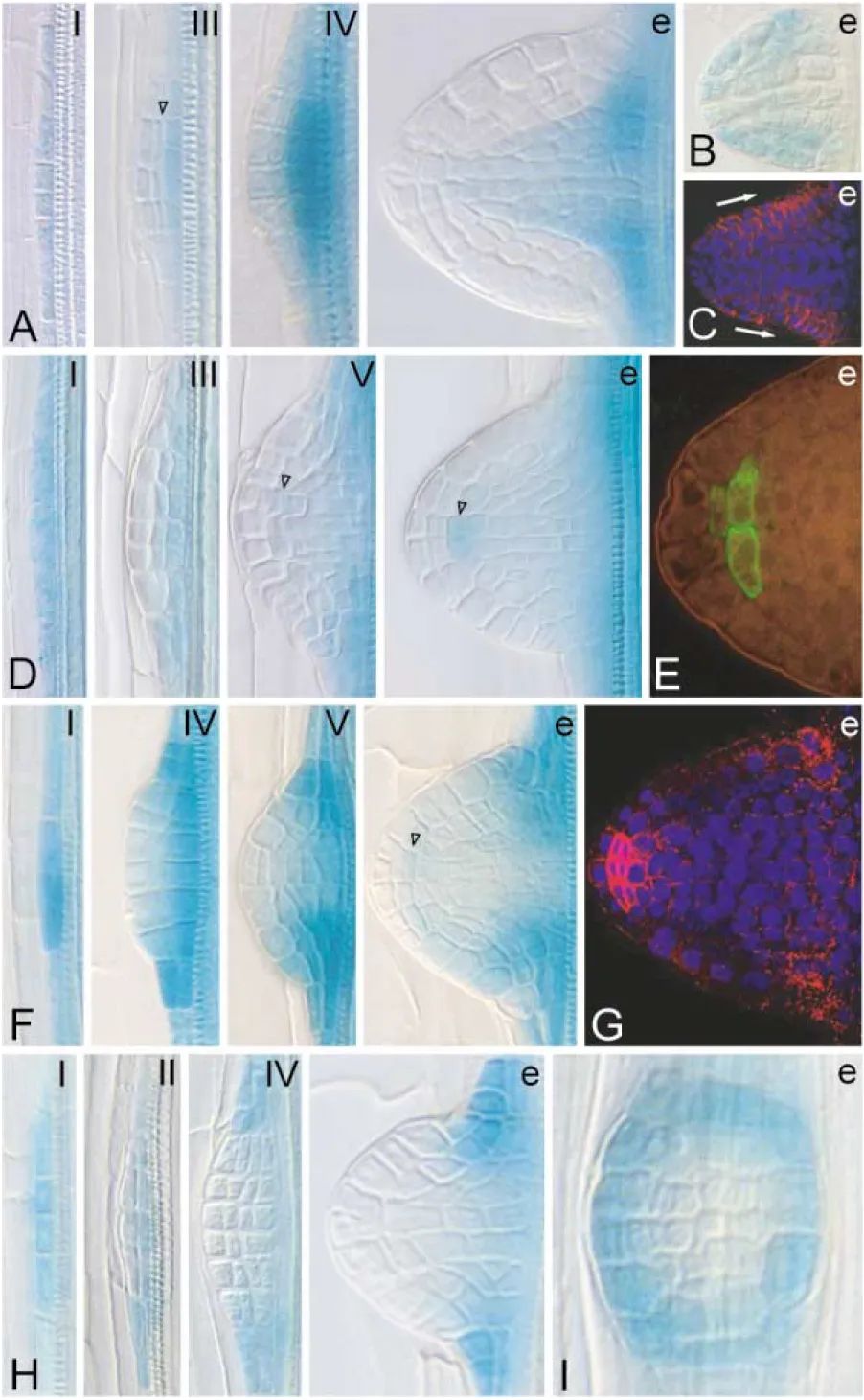
4.1.2 创建突变体库或直接购买
4.1.3 转基因研究
4.1.4 蛋白实验
图18 蛋白-核酸相互作用的筛选和点对点验证方法总结。
Bokor B, Soukup M, Vaculik M, Vd’acny P, Weidinger M, Lichtscheidl I, Vavrova S, Soltys K, Sonah H, Deshmukh R, Belanger RR, White PJ, El-Serehy HA, Lux A (2019) Silicon Uptake and Localisation in Date Palm (Phoenix dactylifera) – A Unique Association With Sclerenchyma. Front Plant Sci 10:988. doi:10.3389/fpls.2019.00988
Duncan S, Olsson TSG, Hartley M, Dean C, Rosa S (2016) A method for detecting single mRNA molecules in Arabidopsis thaliana. Plant Methods 12:13. doi:10.1186/s13007-016-0114-x
Khoshravesh R, Lundsgaard-Nielsen V, Sultmanis S, Sage TL (2017) Light Microscopy, Transmission Electron Microscopy, and Immunohistochemistry Protocols for Studying Photorespiration. Methods Mol Biol 1653:243-270. doi:10.1007/978-1-4939-7225-8_17
Li J, Yokosho K, Liu S, Cao HR, Yamaji N, Zhu XG, Liao H, Ma JF, Chen ZC (2020) Diel magnesium fluctuations in chloroplasts contribute to photosynthesis in rice. Nat Plants 6 (7):848-859. doi:10.1038/s41477-020-0686-3
Wood CC, Robertson M, Tanner G, Peacock WJ, Dennis ES, Helliwell CA (2006) The Arabidopsis thaliana vernalization response requires a polycomb-like protein complex that also includes VERNALIZATION INSENSITIVE 3. Proc Natl Acad Sci U S A 103 (39):14631-14636. doi:10.1073/pnas.0606385103
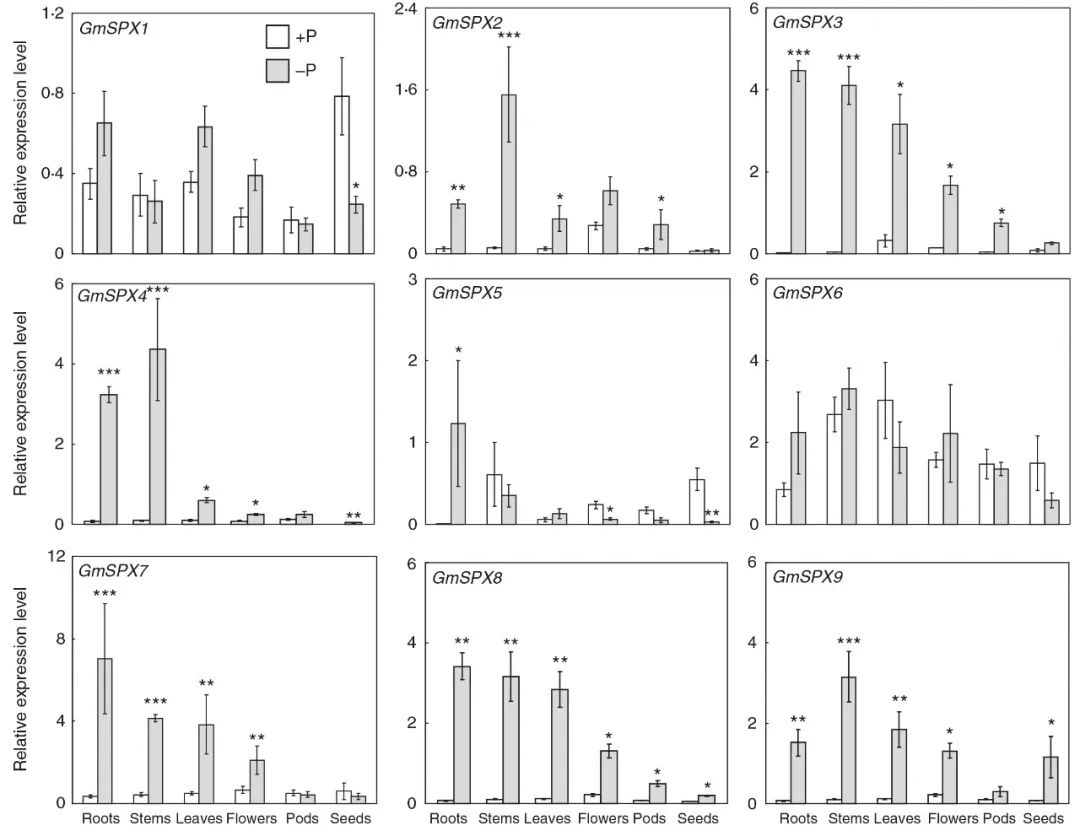
Yao Z, Tian J, Liao H (2014) Comparative characterization of GmSPX members reveals that GmSPX3 is involved in phosphate homeostasis in soybean. Ann Bot 114 (3):477-488. doi:10.1093/aob/mcu147
官网链接:plant.biorun.com