基因的定向进化通过模仿自然选择的过程,以将基因产生的蛋白功能导向自定义的目标,其过程包括突变的产生、筛选和扩增。相较于体外定向进化技术,基于CRISPR的原位定向进化技术更适用于定向进化多数作物关键基因,以达到作物改良的目的。如何创制变异类型更加丰富的饱和突变体文库是原位定向进化技术的关键。近日,万建民院士团队开发了新型的多重正交碱基编辑器MoBE和随机化多sgRNA组装技术,极大地提升了原位定向进化所需的潜在突变丰度,并获得了新的抗除草剂水稻突变植株。

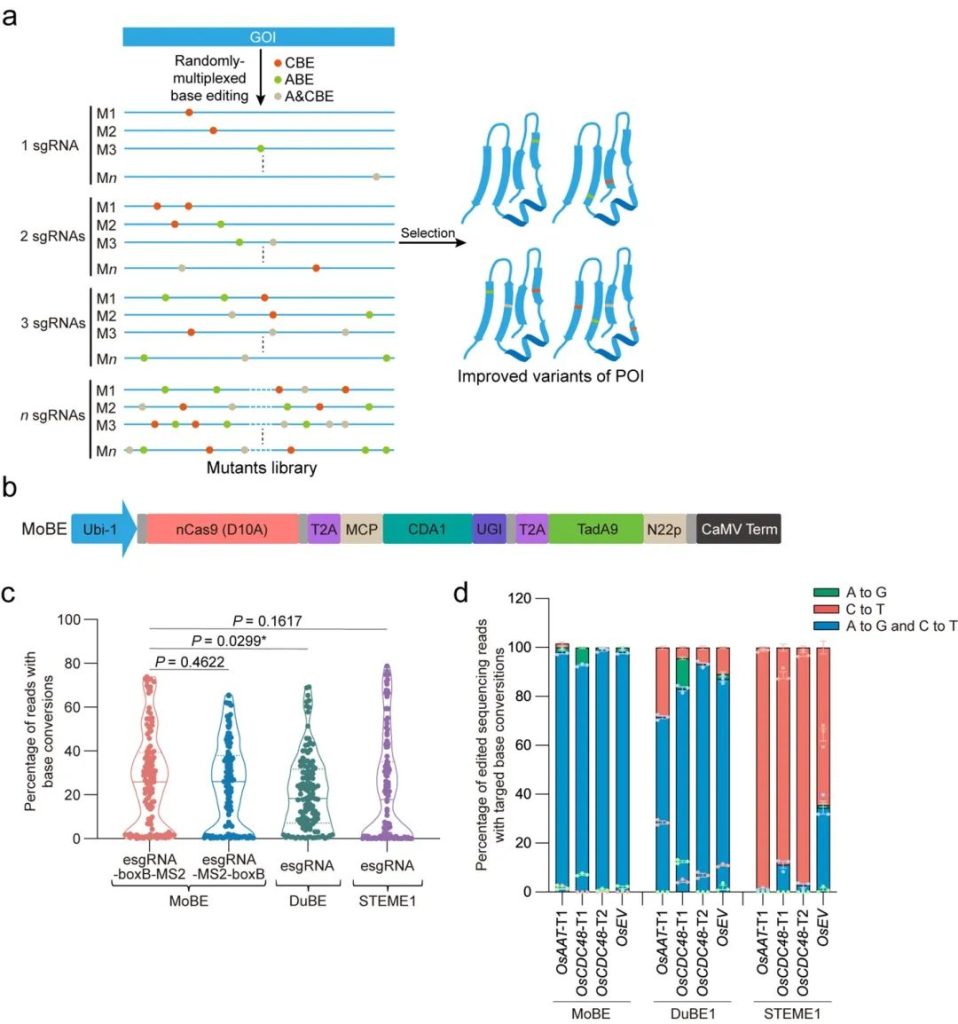 图1 多重正交碱基编辑器MoBE的开发
研究者首先采用RNA配体招募形式的碱基编辑器构象,筛选出高效的腺嘌呤脱氨酶TadA9和胞嘧啶脱氨酶CDA1。利用T2A“自剪切”肽将nCas9 (D10A)切口酶、腺嘌呤碱基编辑模块(TadA9-N22p)和胞嘧啶碱基编辑模块(TadA9-N22p)串联在同一个Ubi-1启动子后面,构建了多重正交碱基编辑器MoBE。在新开发的嵌合RNA配体作用下,MoBE介导的A>G和C>T双碱基编辑效率高达73.9%,其中两种碱基同时编辑的产物平均占比达97% (图1)。在三种不同RNA配体形式的作用下,MoBE可以在多位点上分别实现高效的ABE、CBE和A&CBE多重编辑。同时,研究者还开发了MoBE-HF,显著降低了MoBE的脱靶风险。
图1 多重正交碱基编辑器MoBE的开发
研究者首先采用RNA配体招募形式的碱基编辑器构象,筛选出高效的腺嘌呤脱氨酶TadA9和胞嘧啶脱氨酶CDA1。利用T2A“自剪切”肽将nCas9 (D10A)切口酶、腺嘌呤碱基编辑模块(TadA9-N22p)和胞嘧啶碱基编辑模块(TadA9-N22p)串联在同一个Ubi-1启动子后面,构建了多重正交碱基编辑器MoBE。在新开发的嵌合RNA配体作用下,MoBE介导的A>G和C>T双碱基编辑效率高达73.9%,其中两种碱基同时编辑的产物平均占比达97% (图1)。在三种不同RNA配体形式的作用下,MoBE可以在多位点上分别实现高效的ABE、CBE和A&CBE多重编辑。同时,研究者还开发了MoBE-HF,显著降低了MoBE的脱靶风险。
 图2 饱和sgRNA设计在线工具PlantBE-CODE
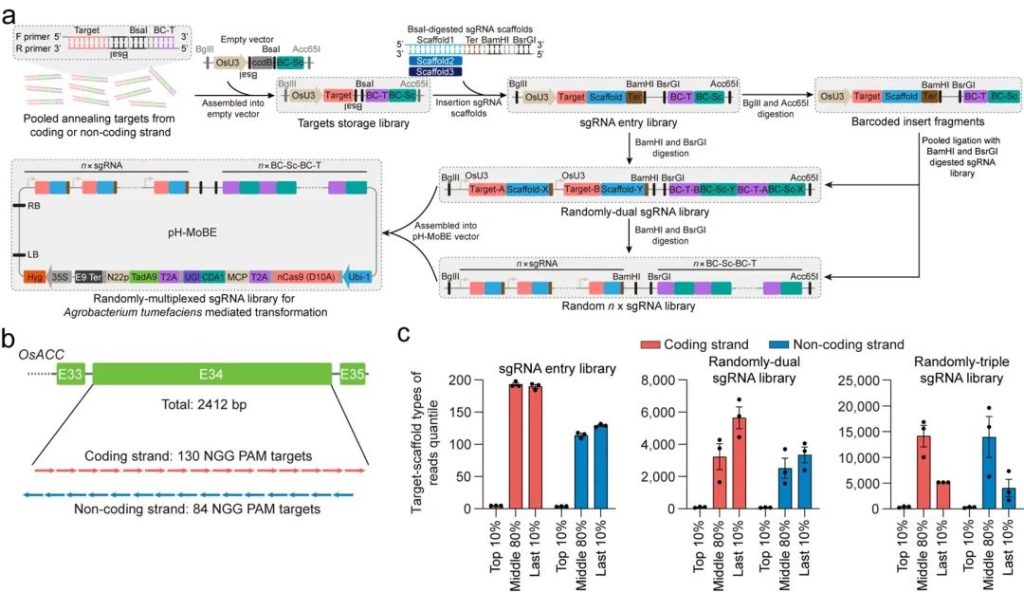
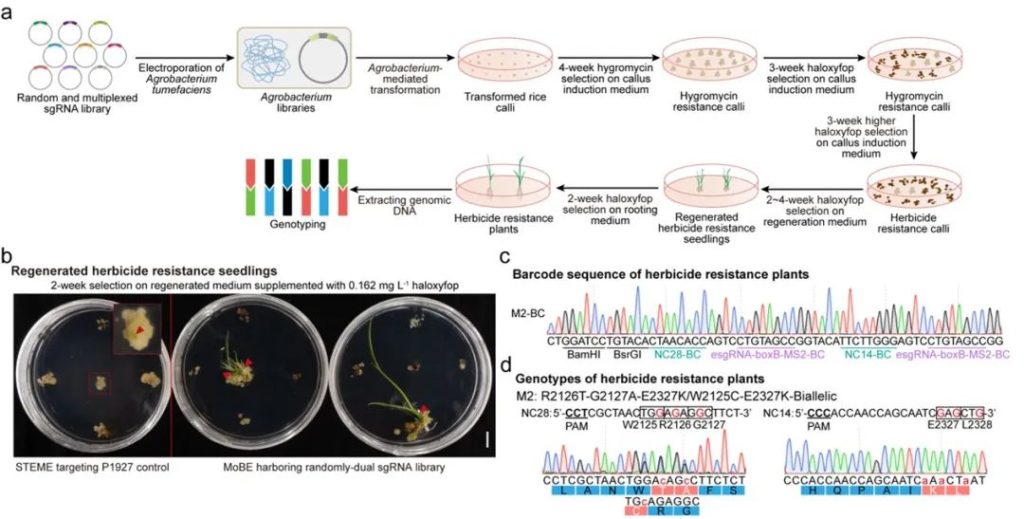
为实现高通量的饱和编辑sgRNA设计,研究者开发了在线工具PlantBE-CODE (http://pgec.njau.edu.cn/plantbe-code) (图2)。以饱和突变OsACC的第34个外显子为例,利用同尾酶开发了随机化多sgRNA组装技术,并通过添加Barcode序列来追踪靶点和所招募的编辑器信息(图3)。在随机化多sgRNA文库中,每串联一个sgRNA表达模块,不同靶点和不同编辑器的组合类型便会显著增加。将MoBE和随机化双sgRNA文库相结合,原位定向进化水稻OsACC内源基因(图4)。在含1.5倍浓度高效氟吡甲禾灵(Haloxyfop)的培养基筛选压力下,获得了新的抗除草剂的突变体V1703I-W2125C-Hetero、R2126T-G2127A-E2327K/W2125C-E2327K-Biallelic、D1970N/W2125C-Biallelic和I2139N/F2207L-Biallelic等。
图3 随机多sgRNA组装技术的开发
MoBE和随机化多sgRNA组装技术的建立,进一步拓展了基于CRISPR的作物原位定向进化技术体系,为作物基因组编辑新种质创制和挖掘优良等位变异提供了新的工具支撑。该研究成果于2023年08月12日在线发表于植物学知名期刊Plant Biotechnology Journal (doi: 10.1111/pbi.14156)。
图4 原位定向进化OsACC创制抗除草剂新种质
南京农业大学已毕业硕士生张傲、在读博士生单调风和孙岩为本文共同第一作者,万建民院士和李超教授为共同通讯作者。南京农业大学刘裕强教授、吴玉峰教授、董小鸥教授、江玲教授和中国科学院遗传与发育生物学研究所王延鹏研究员等也参与了部分研究工作。该研究得到农业农村部、江苏省前沿引领技术基础研究专项、海南省崖州湾种子实验室和中国科协等项目资助。
图2 饱和sgRNA设计在线工具PlantBE-CODE
为实现高通量的饱和编辑sgRNA设计,研究者开发了在线工具PlantBE-CODE (http://pgec.njau.edu.cn/plantbe-code) (图2)。以饱和突变OsACC的第34个外显子为例,利用同尾酶开发了随机化多sgRNA组装技术,并通过添加Barcode序列来追踪靶点和所招募的编辑器信息(图3)。在随机化多sgRNA文库中,每串联一个sgRNA表达模块,不同靶点和不同编辑器的组合类型便会显著增加。将MoBE和随机化双sgRNA文库相结合,原位定向进化水稻OsACC内源基因(图4)。在含1.5倍浓度高效氟吡甲禾灵(Haloxyfop)的培养基筛选压力下,获得了新的抗除草剂的突变体V1703I-W2125C-Hetero、R2126T-G2127A-E2327K/W2125C-E2327K-Biallelic、D1970N/W2125C-Biallelic和I2139N/F2207L-Biallelic等。
图3 随机多sgRNA组装技术的开发
MoBE和随机化多sgRNA组装技术的建立,进一步拓展了基于CRISPR的作物原位定向进化技术体系,为作物基因组编辑新种质创制和挖掘优良等位变异提供了新的工具支撑。该研究成果于2023年08月12日在线发表于植物学知名期刊Plant Biotechnology Journal (doi: 10.1111/pbi.14156)。
图4 原位定向进化OsACC创制抗除草剂新种质
南京农业大学已毕业硕士生张傲、在读博士生单调风和孙岩为本文共同第一作者,万建民院士和李超教授为共同通讯作者。南京农业大学刘裕强教授、吴玉峰教授、董小鸥教授、江玲教授和中国科学院遗传与发育生物学研究所王延鹏研究员等也参与了部分研究工作。该研究得到农业农村部、江苏省前沿引领技术基础研究专项、海南省崖州湾种子实验室和中国科协等项目资助。